Возможности и ограничения метода MLVA при идентификации бактериальных штаммов на примере изолятов C.difficile
Автор: Шахбиев И.Х., Новикова О.Б., Терлецкий В.П.
Журнал: Форум молодых ученых @forum-nauka
Статья в выпуске: 9 (25), 2018 года.
Бесплатный доступ
Клостридиозы являются серьезными заболеваниями у людей и животных. Часто заболевание возникает и прогрессирует при назначении больным антибиотиков, которые убивают другие виды микроорганизмов в желудочно-кишечном тракте и, таким образом, убирают защитный барьер, препятствующий развитию клостридий. Этот патоген относят к внутрибольничным инфекциям, поэтому генотипирование его изолятов является важной задачей профилактической медицины. В статье изложены данные по использованию генотипирования, основанного на ПЦР-амплификации повторяющихся элементов в геноме C.difficile.
Патогенны, клостридиозы, днк, генотипирование, повторяющиеся элементы
Короткий адрес: https://sciup.org/140284316
IDR: 140284316
Текст научной статьи Возможности и ограничения метода MLVA при идентификации бактериальных штаммов на примере изолятов C.difficile
Одним из распространенных видов микроорганизмов, обитающих в желудочно-кишечном тракте человека и животных и имеющих большое значение с медицинской точки зрения, является Грам-положительная бактерия Clostridium difficile [1]. C.difficile обнаруживается в микрофлоре кишечника у 80% здоровых детей в возрасте до 1 года и у 1-5% здоровых взрослых. В то же время, у госпитализированных взрослых носительство может достигать 20-40%. Данная бактерия впервые признана возбудителем колита у людей в 1978 году [5]. С тех пор ее значение как патогенного микроорганизма существенно возросло. Было показано, что бактерия развивается после назначения пациентам антибиотиков, которые угнетают конкурентные виды микробов и при этом не оказывают негативного влияния на C. difficile [2; 4]. В последние годы отмечается не только повышение частоты внутрибольничной инфекции, вызванной C. difficile, но и ее летальность [4]. В этой связи проблеме клостридиоза уделяют большое внимание, в частности, интерес представляет генетическая вариабельность штаммов.
Несмотря на значительное число публикаций по эпидемиологии C.difficile, остается спорным вопрос об эндо- и экзогенном характере инфекции и путях передачи возбудителя. Внутрибольничные случаи заболевания могут носить как спорадический, так и эпидемический характер. Эпидемическое распространение получают лишь отдельные штаммы, например риботип 1 и риботип 027, производящими в 20 раз больше токсинов А и В, чем другие патогенные штаммы. Кроме того, отмеченные штаммы устойчивы ко многим широко используемым антибиотикам.
Эффективный контроль над вспышками заболевания возможен при быстрой идентификации изолятов, позволяющей дифференцировать эпидемические и спорадические штаммы. В отношении C.difficile разработаны и используются несколько методов генотипирования. Наиболее распространенным сейчас в Европе является метод риботипирования, основанный на вариабельности интергенного участка между 16S и 23S генами рибосомной ДНК. В США предпочтение отдают методу пульс-гель электорофореза. Используется также метод выявления вариаций тандемных повторов ДНК (MLVA) и секвенирование генов «домашнего хозяйства» (MLST). К сожалению, эти методы не лишены недостатков. В частности, риботипирование часто не различает эпидемиологически не родственные изоляты, т.е. обладает недостаточной дискриминационной способностью. Пульс-гель электрофорез проводится в течение длительного времени и требует значительных материальных и людских затрат. Секвенирование (MLST) является дорогостоящей процедурой и иногда не позволяет добиться необходимого уровня дискриминации штаммов.
Способом выявления изменений в геномах микроорганизмов является генотипирование, которое приобрело большое значение в системе профилактики инфекционных заболеваний, так как позволяет установить источники инфекции и выявить пути ее распространения [3; 6]. В США референтные лаборатории часто применяют при генотипировании метод пульс-гель электрофореза (ПГЭ). В Европе более популярным является риботипирование изолятов, основанное на вариабельности участка между 16S и 23S генами рибосомной ДНК, выявляемой ПЦР. Помимо этих двух методов генотипирования используются и другие, например, метод выявления вариаций тандемных повторов ДНК (MLVA) и секвенирование по семи генам «домашнего хозяйства» (MLST). К сожалению, указанные методы не лишены недостатков. ПГЭ является длительной и трудоемкой процедурой и чувствительной к наличию эндонуклеаз, что снижает типируемость. Секвенирование (MLST) иногда не позволяет добиться необходимого уровня в дискриминации штаммов.
Материалом исследования служила геномная ДНК, выделенная от 25 культур госпитальных изолятов C. difficile . Ночную культуру микроорганизма в объеме 1 мл, выращенную в анаэробных условиях в контейнере, центрифугировали в течение 5 минут при 8000g. Осадок бактериальных клеток использовали для выделения геномной ДНК стандартным методом с использованием фенольно-хлороформенной экстракции.
Данный анализ проводили на этих же изолятах с использованием подхода, описанного в 2009 году [7]. Использовали следующие праймеры для амплификации полиморфных тандемных повторов: TR6-F 5'-TTTCAACTTGTCCAGTTTTTAAGTC-3'
TR6-R 5'-ATGACATAGCGTTTGTGGAAT-3';
TR10-F 5'-TGCATCAAATTGGTCAAGACTC-3'
TR10-R 5'-TGAAATCATTGACTATAAAGCAAAA-3'.
Условия амплификации соответствовали оригинальной методике [7]. Реакцию проводили с использованием 1 мкл геномной ДНК C. difficile , 200 мкМ каждого из четырех дезоксинуклеотидтрифосфатов, 0.1 мкМ праймеров, 1х ПЦР буфер, 1 е.а. Hot Start Taq-полимеразы (Invitrogen™). ПЦР включала начальную денатурацию ДНК при 96°C в течение 3 минут, затем проводили 35 циклов: 96°C 45 секунд, 52°C 45 секунд, 72°C 45 секунд. В качестве финального шага использовали инкубацию при 72°C в течение 7 минут. Общий объем смеси составлял 25 мкл, реакция проводилась в стандартных 96-ти луночных планшетах для ПЦР. Продукты реакции разделяли в агарозном геле, окрашивали флуоресцентым красителем GelRed™ (Bio-Rad™), фотографировали в системе гель-документации. В качестве маркера длин фрагментов ДНК использовали GeneRuler™ 100 bp Plus DNA Ladder (Thermo Fisher Scientific™).
Применение метода MLVA с двумя парами праймеров по локусам TR6 и TR10 привело к появлению амплификатов разного размера у отдельных изолятов.
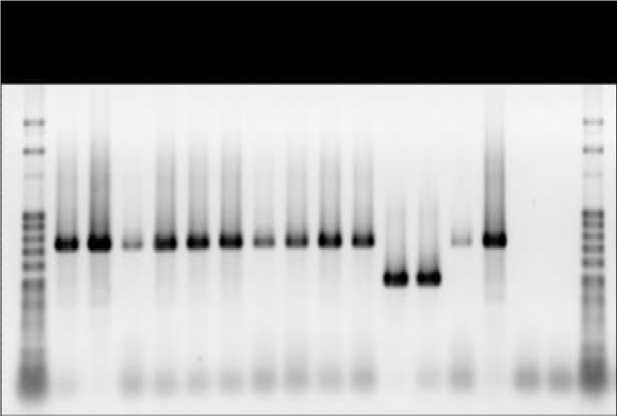
Рис. 1. MLVA-генотипирование группы изолятов C.difficile с помощью ПЦР праймеров для локуса TR6
Однако, как в отдельности, так и в комбинации, вариация размеров касалась лишь небольшого числа изолятов из 25 изученных. В частности, по локусу TR6 выявлено всего 2 генотипа, причем один из них встречался всего у 2-х изолятов – 27760 и 27762 (генотип 1, Рис. 1), а остальные изоляты были идентичными и рассматривались как генотип 2. Локус TR10 был немного информативней (Рис.2). Основным генотипом был генотип 1, выявляемый у 22 изолятов. Кроме того, были выявлены уникальный генотип 2 (изолят 27525) и генотип 3 (изоляты 27526 и 27533).
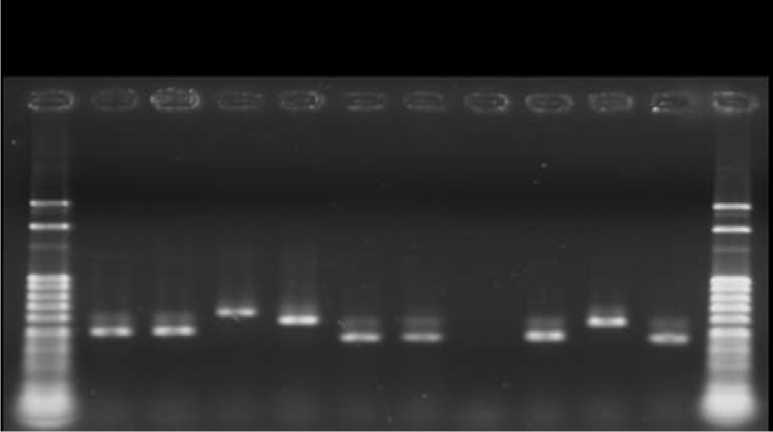
Рис. 2. MLVA-генотипирование группы изолятов C.difficile с помощью ПЦР праймеров для локуса TR10
Низкий полиморфизм локусов TR и TR10 в нашей группе изолятов не позволяет рекомендовать этот подход для генотипирования изолятов C. difficile. Наши данные не соответствуют выводам по использованию этих локусов, сделанных авторами в ранее опубликованной работе в Германии [7], где индекс дискриминации достигал значения 0,967. Одной из причин такого противоречия может быть большее исходное генетическое разнообразие изолятов в работе немецких ученых, что позволило выявить разницу в длине тандемных повторов у большинства изолятов. В нашей работе, все изоляты имели четкую временную и географическую привязку к одному месту (госпиталь кантона Во, Швейцария) и они могли оказаться генетически близкими друг к другу.
В заключение, метод генотипирования MLVA при использовании двух локусов тандемных повторов TR6 и TR10 имеет ограниченную дискриминационную способность, для повышения которой необходимо в анализ дополнительно включать другие локусы, либо в дополнение использовать другие методы генотипирования.
Работа поддержана грантом РФФИ по заявке № 18-316-00051 мол_а в части проведения генотипирования бактерий и при поддержке Государственного задания ФАНО 599.01.Х30170599-2014-0222 в части анализа полученных результатов
Список литературы Возможности и ограничения метода MLVA при идентификации бактериальных штаммов на примере изолятов C.difficile
- Лобзин Ю.В., Захаренко С.М., Иванов Г.А. Современные представления об инфекции Clostridium difficile // Клиническая микробиология и антимикробная химиотерапия. 2002. Т. 4. №3. С. 200-232.
- Малов В.А. Инфекция Clostridium difficile: современное состояние проблемы // Фарматека. 2010. Т. 4. С. 27-31.
- Терлецкий В.П., Тыщенко В.И., Новикова О.Б., Борисенкова А.Н., Белаш Д.Э., Яковлев А.Ф. Эффективный молекулярно-генетический метод идентификации штаммов сальмонелл и протея // Доклады РАСХН. 2013. № 5. С.60-63.
- Шоп И.В. Комплексный взгляд на проблему внутрибольничной диареи, вызванной Clostridium difficile и не только // Болезни и антибиотики. 2013. Т. 1. №8. С. 60-67.
- Bartlett J.G., Chang T.W., Gurwith M., Gorbach S.L., Onderdonk A.B. Antibiotic-associated pseudomembranous colitis due to toxin-producing clostridia // N. Engl. J. Med. 1978. V. 298. No 10. P.531-534.
- Terletskiy V., Kuhn G., Francioli P., Blanc D. Application and evaluation of double digest selective label (DDSL) typing technique for Pseudomonas aeruginosa hospital isolates // Journal of Microbiological Methods. 2008. V. 72 P. 283-287
- Zaiss N.H., Rupnik M., Kuijper E.J., Harmanus C., Michielsen D., Janssens K. et.al. Typing Clostridium difficile strains based on tandem repeat sequences // BMC Microbiol. 2009. V. 9. P.6.


