Алгоритм выявления и видовой идентификации бактерий в крови с использованием ПЦР
Автор: Киселва Екатерина Евгеньевна
Журнал: Вестник гематологии @bulletin-of-hematology
Рубрика: Оригинальные статьи
Статья в выпуске: 1 т.13, 2017 года.
Бесплатный доступ
Короткий адрес: https://sciup.org/170149956
IDR: 170149956 | УДК: 615.38
Текст статьи Алгоритм выявления и видовой идентификации бактерий в крови с использованием ПЦР
Введение. Актуальность проблемы ранней диагностики бактериемии и сепсиса обу-славливаетсятем,чтогенерализованные бактериальные инфекции являются тяжелыми осложнениями у гематологических больных пациентов отделений интенсивной терапии а также у лиц, подвергающихся воздействию неблагоприятных экологических и производственных факторов (ионизирующее излучение, компоненты ракетного топлива и др.) [1, 2, 3]. Ежегодно регистрируется более 18 миллионов случаев сепсиса по всему миру а количество летальных исходов достигает 30–40 % (около 135 000 смертей в год в Европе и 215 000 в США) [4, 5]. Кроме того, на лечение сепсиса расходуются немалые средства: в США на каждого пациента с подтвержденным сепсисом тратится 25000$ за госпитали-зацию,что составляет около 17 миллиардов $ в год [4]. Успех в лечении сепсиса опреде- ляется правильностью выбора стартовой эмпирической антибактериальной терапии так как существующие микробиологические методы диагностики позволяют выявить возбудителя слишком поздно для принятия своевременных лечебных мер.Поэтому существует настоятельная необходимость совершенствования методов ранней диагностики бактериемии и сепсиса [6, 7].
В настоящее время «золотым стандартом» лабораторной диагностики бактериемий и сепсиса является гемокультивирование. При подозрении на инфекцию кровотока у пациента,еще до начала эмпирической терапии антибиотиками широкого спектра действия, производится забор крови для бактериального посева. Забор крови рекомендуется проводить 2–3 раза по 20–30 мл (по 10–15 мл для двух параллельных высевов на аэробные и анаэробные среды). Исследо- вания показывают, что при меньшем объеме забираемой крови частота выявления возбудителей существенно снижается [8]. При культивировании крови наиболее широко используются две автоматические системы: BacT/ALERT (BioMerieux) и Bactec 9240 (BectonDickinson). Обе системы основаны на регистрации количества СО2,выделяемого бактериями в процессе жизнедеятельности. Разработан широкий спектр сред для аэробного и анаэробного культивирования. Также важным достоинством автоматизированного культивирования является производство флаконов с добавками сорбентов,подавляю-щих антибиотики,что дает возможность анализа образцов, полученных от пациентов, уже принимавших антибиотики.
Срок культивирования посевов составляет 5–7 суток, по истечении которых образец не давший роста, считается отрицательным. В случае же получения положительного сиг-нала,флакон немедленно вынимается из аппарата, проводится окрашивание по Граму (для определения грампринадлежности) а также пересев на твердую питательную среду (для получения чистой культуры). После получения чистой культуры происходит ее идентификация с помощью различных биохимических тестов, а также проводится тест на выявления резистентности к антибиотикам. Таким образом, результат анализа может быть получен только через несколько суток. Кроме того микробиологический метод неприемлем для прихотливых и некультивиру-емых бактерий, таких как микоплазмы, но-кардии, риккетсии, хламидии и ряд других микроорганизмов.
С недостатками классического микробиологического метода призваны бороться новые разрабатываемые методики. Перспективными являются методы, основанные на применении технологии нуклеиновых кислот (NAT (Nucleic Acid Technology) -технологий), в частности полимеразной цепной реакции (ПЦР).
В гене 16S рРНК (наиболее часто разрабатываемом в данной группе методов) существует участок, общий для всех бактерий, что позволяет обнаружить присутствие бактерий в крови. Также в гене находятся участки специфичные для определенных родов и видов бактерий. Кроме того, NAT-технологии применимы для обнаружения генов антибио-тикорезистентности микроорганизмов.
В настоящее время существует две стратегии применения этого направления для диагностики сепсиса:
-
• прямое выявление нуклеиновых кислот микроорганизмов из цельной крови;
-
• использование ПЦР для быстрой видовой идентификации микроорганизмов после автоматического культивирования.
Первая группа методов позволяет идентифицировать микроорганизмы с помощью ПЦР прямо из образцов цельной крови. Основной трудностью является выделение бактериальной ДНК непосредственно из цельной крови,поскольку при сепсисе концентрация бактерий в крови взрослого человека составляет от < 1 до 100 КОЕ (колониеобразующих единиц) на мл. В настоящее время существует система MolYsis (Molzyme, Германия), которая позволяет не только получить приемлемое для дальнейшего анализа количество микробной ДНК, но также значительно лучше, чем другие подобные системы очищает ее от человеческой ДНК и других ингибиторов ПЦР [9]. Однако выделение ДНК микроорганизмов напрямую из крови сопряжено с одним существенным недостатком — возможностью выделения ДНК уже нежизнеспособных микроорганизмов, не имеющих клинического значения, что приводит к получению ложноположительных результатов и назначению неверных антибиотиков.С другой стороны,данные методы обладают очень большим преимуществом — возможностью получения результатов анализа в течение 4–6 часов.
Вторая группа методов объединяет в себе достоинства как классического культурального метода, так и ПЦР:
-
• выявление микроорганизмов, способных к размножению, и быстрое определение их чувствительности к антибиотикам;
-
• высокая чувствительность методов (до 1 КОЕ/мл);
-
• возможность определения некультиви-руемых форм возбудителей;
-
• значительное сокращение времени анализа.
Суть этих методов заключается в том, что сразу после получения положительного сигнала из автоматического анализатора флакон с посевом крови изымают и выделяют бактериальную ДНК из гемокультуры для дальнейшей идентификации с помощью ПЦР.
Несмотря на то,что методика выявления и идентификации микроорганизмов с помощью ПЦР с предварительным гемокультивированием не лишена недостатков (для проведения анализа требуется большой объем крови, наличие некультивируемых и слож-нокультивируемых микроорганизмов), это очень перспективный метод, который успешно применялся в данном исследовании.
Целью данной работы была оценка эффективности молекулярно-биологического метода выявления и видовой идентификации бактерий в крови с использованием ПЦР «в реальном времени» (ПЦР-РВ), с последующей разработкой алгоритма применения этого метода.
Суть предлагаемого метода заключается в выявлении бактериальных возбудителей в крови при бактериемии и сепсисе с помощью автоматического бактериологического анализатора с последующей родовой и видовой идентификацией молекулярно-биологическим методом (ПЦР в «реальном времени»)
Материалы и методы. Поскольку метод выявления и видовой идентификации бактерий в крови разрабатывался в первую очередь для больных гемобластозами на основании литературных и собственных данных был определен спектр основных возбудителей бактериемии и сепсиса для этой категории пациентов (более 95 % рис. 1 и 2 ) [10,11].
Прочие 7%
Streptococcus viridans 2%
Streptococcus pneumoniae 1%
Corynebacterium spp. 7%
Enterococcus 4%
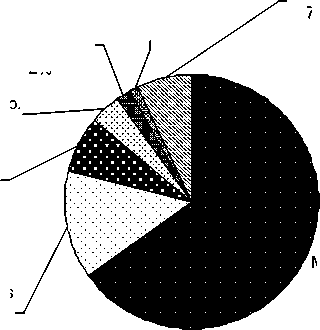
MRCoNS 65%
Рисунок 1.
Видовой состав грамположительных бактерий, наиболее частых возбудителей бактериемии и сепсиса
Staphylococcus aureus 14%
Прочие 9%
Salmonella spp. 3%
Acinetobacter spp. 5%
Pseudomonas spp 8%
Klebsiella spp. 4%
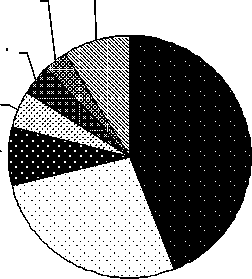
Escherichia coli 44%
Сем-во
Enterobacteriaceae
27%
Рисунок 2.
Видовой состав грамотрицательных бактерий, наиболее частых возбудителей бактериемии и сепсиса
В соответствии с выявленным спектром совместно с ФБУН ЦНИИ эпидемиологии Роспотребнадзора был разработан ряд наборов для постановки ПЦР-РВ для выявления определенных групп микроорганизмов, а также генов устойчивости к антибиотикам:
-
1. Набор реагентов «АмплиПрайм®
-
2. Набор реагентов «ИМП-G », для выявления ДНК бактерий рода Enterococcus .
-
3. Набор реагентов «G-», для выявления ДНК Acinetobacter baumannii , Klebsiella pneumoniae , Pseudomonas aeruginosa .
-
4. Набор реагентов «ИМП-скрин-FL», для выявления ДНК Escherichia coli .
-
5. Набор реагентов «АмплиСенс®MRSA-скрин-титр-FL», для выявления ДНК метициллин-чувствительного ( MSSA )
-
6. Набор реагентов «АмплиСенс®MDR
-
7. Набор реагентов «АмплиСенс®MDR KPC/ OXA-48-FL», для выявления генов приобретенных карбапенемаз групп KPC OXA-48-подобных.
-
8. Набор реагентов «АмплиСенс® Флоро-Ценоз / Кандиды-FL», для выявления ДНК C.albicans, C.glabrata, C.krusei, C.parapsilosis, C.tropicalis .
ФлороЦеноз-Аэробы», для выявления ДНК бактерий семейства Enterobacteriaceae , а также Streptococcus spp. и Staphylococcus spp .
и метициллин-резистентного ( MRSA ) Staphylococcus aureus , а также метицил-лин-резистентных коагулазонегатев-ных Staphylococcus spp . ( MRCoNS ).
MBL-FL»,для выявления генов приобретенных карбапенемаз класса металло-β-лактамаз, групп VIM, IMP, NDM.
В последние годы наметилась тенденция к возрастанию удельного веса микромице-тов среди других инфекционных агентов выявляемых в крови онкогематологических больных (в некоторые годы доля микромице-тов среди выявляемых патогенов достигала почти 10 %). Чаще всего выявляются дрожжеподобные грибы рода Candida [12]. В связи с этим было решено добавить набор реагентов для выявления ДНК грибов рода Candida :
Все представленные наборы,а также методика на основе ПЦР-РВ для расшифровки этиологии септических состояний ранее были апробированы [13]. Часть наборов уже сертифицирована и выпускается фирмой ООО «ИЛС», а часть готовится к сертификации.
Материалами для исследования послужили 20 гемокультур, полученных при посевах крови больных различными опухолевыми заболеваниями системы крови.
Для оценки эффективности молекулярнобиологического метода выявления и видовой идентификации бактерий в крови больных идентификацию выделенных культур проводили 2-мя способами: с использованием общепринятых бактериологических методов (пересев на твердую питательную среду, проведение биохимических тестов)и с использованием новой методики на основе ПЦР-РВ.
Выделение ДНК из гемокультур проводилось с помощью набора ДНК-сорб-АМ» (ООО «ИЛС»), но процесс выделения был несколько модифицирован (добавлено предварительное центрифугирование образца для получения рабочего осадка;лизис клеток проводится не в присутствии сорбента; увеличен до 200 мкл объем ТЕ-буфера,добавляемого для растворения ДНК). Процесс амплификации и анализ результатов проводился в порядке, указанном производителем в инструкциях к наборам для постановки ПЦР-РВ.
Результаты и обсуждение. При обследовании 20 гемокультур для оценки эффективности молекулярно-биологического метода выявления и видовой идентификации бактерий были получены следующие результаты. В 7 образцах бактериологическим методом была обнаружена E.coli. Методом ПЦР-РВ в этих образцах была также обнаружена ДНК E.coli. В трех случаях бактериологическим методом были выявлены неферментирующие грамотрицательные бактерии, в двух из которых идентифицирована Ps.aeruginosa и бактериологически и методом ПЦР-РВ. В третьем образце методом ПЦР-РВ не было обнаружено ДНК бактерий. Это, вероятно, связано с тем, что в разработанный набор вошли не все неферментирующие грамотри-цательные бактерии, а только наиболее часто встречающиеся. В двух образцах микробиологическим методом обнаружены бактерии рода Enterobacter. При исследовании в ПЦР-РВ в этих образцах была выявлена ДНК бактерий семейства Enterobacteriaceae, кроме того в незначительных количествах — ДНК Kl.pneumoniae. Поскольку Enterobacter не является часто встречающимся представителем семейства Enterobacteriaceae, он отдельно не был включен в разрабатываемый набор,но выявление большого количества ДНК сем. Enterobacteriaceae косвенно подтверждает результаты бактерио-логическогоанализа.В6 случаях бактериологическим методом обнаружен S.epidermidis. Методом ПЦР-РВ в 5 образцах найдена ДНК MRCoNs что совпадает с результатами микробиологии а в одном — MSSA. Данный дискордантный результат может являться следствием изменения биохимических свойств штамма стафилококка под действием химиотерапии. Таким образом эффективность метода составила более 94,4 %.
По результатам проведенных исследований был разработан алгоритм выявления и видовой идентификации бактерий в крови с использованием молекулярно-биологического метода (рис. 3) .
Предлагаемый алгоритм состоит в следующем: при подозрении на инфекции кровотока у пациента производится посев крови с дальнейшей инкубацией в автоматическом анализаторе (в соответствии с инструкцией производителя). Если рост отсутствует в течение 7 суток, врачу сообщается результат «роста нет». При наличии роста (аппарат сигнализирует об этом) флакон незамедлительно извлекается из аппарата и отбирается проба для выделения ДНК и проведения родовой и видовой идентификации с помощью ПЦР. При выявлении бактерий рода Staphylococcus дополнительно проводят определение ме-тициллин-резистентности. При выявлении грамотрицательных бактерий образец дополнительно проверяется на наличие генов приобретенных карбапенемаз. Затем общий результат анализа сразу сообщают врачу.
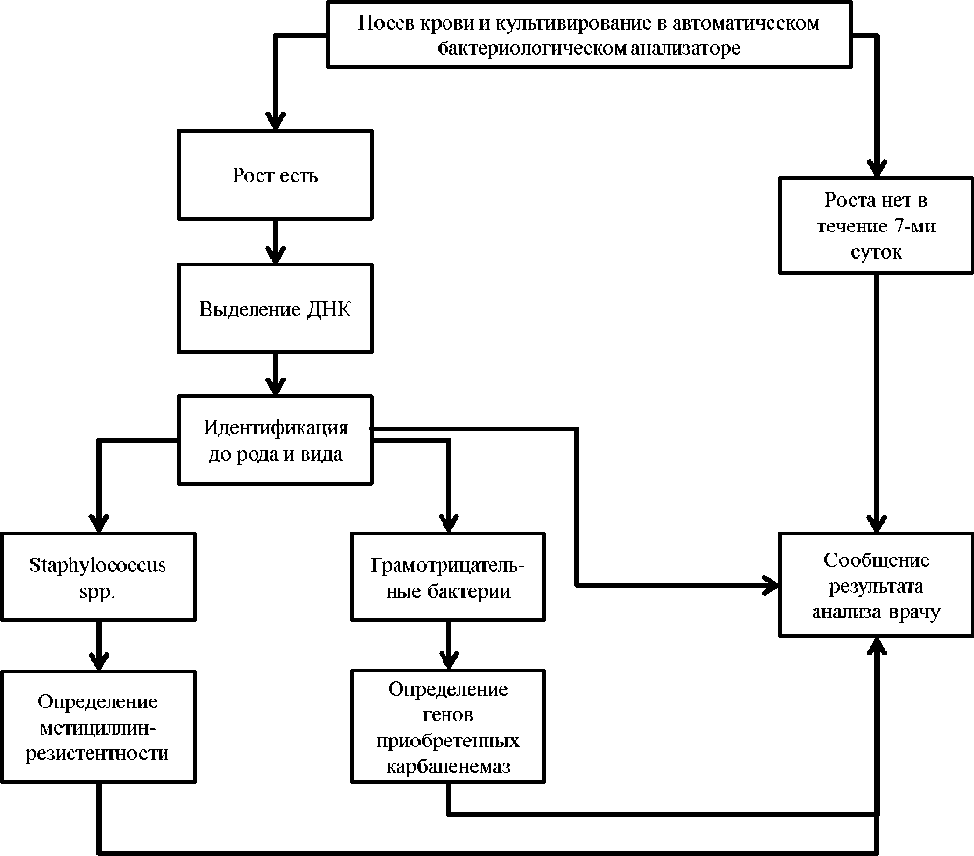
Рисунок 3.
Алгоритм выявления и видовой идентификации бактерий
Заключение . Результаты, полученные бактериологическими и молекулярно-биологическими методами, хорошо согласуются. Эффективность молекулярно-биологического метода родовой и видовой идентификации бактерий составила 94,4 %. Значимым преимуществом разработанного метода, как и алгоритма в целом, является возможность расширения круга выявляемых патогенов за счет дополнения набора тест-системами а также адаптация состава наборов для различных групп пациентов, в зависимости от спектра выявляемых у них возбудителей.
Внедрение в практику разработанного оптимизированного алгоритма позволит существенно сократить время проведения анализа, которое лимитируется только стадией роста микроорганизмов в автоматическом анализаторе (в зависимости от начальной концентрации бактерий в образце крови пациента эта стадия в среднем занимает от 8–12 часов до 5 суток и более), а процесс видовой и родовой идентификации патогенов с использованием ПЦР-РВ занимает всего 5–7 часов,в то время как идентификация патогена классическими микробиологическими методами занимает не менее 48 часов.


