Дифференциация штаммов бруцелл на основе анализа вариабельности VNTR-локусов
Автор: Анисимова Е.А., Додонова Е.А., Миргазов Д.А., Зайнуллин Л.И., Осянин К.А.
Журнал: Вестник Красноярского государственного аграрного университета @vestnik-kgau
Рубрика: Зоотехния и ветеринария
Статья в выпуске: 4, 2024 года.
Бесплатный доступ
Цель исследования - оценка эффективности применения разработанного протокола проведения MLVA для дифференциации штаммов возбудителя бруцеллеза. Данный протокол включает в себя анализ 15 VNTR-локусов с использованием модифицированных MLVA-праймеров. Для in vitro апробации предложенной MLVA схемы использовали выделенную нами ранее ДНК штаммов B. canis RM 6/66, B. suis 1330, B. suis 183-L, B melitensis 1565. MLVA проводили методом ПЦР с последующим разделением ампликонов в агарозном геле. Положительная амплификация наблюдалась для 10 из 15 VNTR-локусов, а именно Bru6, Bru7, Bru9, Bru16 и Bru18, Bru19, Bru21, Bru30, Bru43 и Bru45. Молекулярный размер данных локусов для референтных штаммов B. canis RM 6/66 и B. suis 1330 подтвердили in silico. Также представлены результаты MLVA для штаммов, представленных в базе данных GenBank. Путем поисковых запросов баз данных ресурсов NCBI нами были получены геномные последовательности 49 штаммов бруцелл видов B. canis, B. suis, B. aborus, B melitensis. C помощью биоинформационного анализа для данных штаммов определили молекулярную массу каждого из десяти VNTR-локуса и количество повторов в нем. По результатам проведенного MLVA построили дендрограмму. На основании филогенетического анализа последовательностей десяти вариабельных локусов установлено, что большинство исследуемых штаммов бруцелл распределились на дендрограмме в соответствии с их таксономическим положением. Таким образом заключили, что предложенный нами MLVA-протокол имеет потенциал использования для дифференциации штаммов бруцелл.
Brucella, mlva, пцр, филогенетический анализ, тандемные повторы, бруцеллез
Короткий адрес: https://sciup.org/140306673
IDR: 140306673 | УДК: 579.62 | DOI: 10.36718/1819-4036-2024-4-53-60
Текст научной статьи Дифференциация штаммов бруцелл на основе анализа вариабельности VNTR-локусов
Введение. Бруцеллез – опасное заболевание животных и человека, распространенное преимущественно в странах с интенсивным животноводством [1]. К бруцеллезу восприимчивы все виды теплокровных животных, но чаще всего данное заболевание встречается у крупного рогатого скота, свиней, оленей, коз, овец, лошадей и собак. В России эпидемиологическая ситуация по данному заболеванию также характеризуется как достаточно напряженная [2]. В частности, основная часть случаев заболеваемости регистрируется на территориях СевероКавказского, Южного и Сибирского федеральных округов, в которых отмечается самая высокая заболеваемость крупного рогатого скота (более 80 % от общего количества больных животных в РФ) и мелкого рогатого скота (более 90 %) [3]. Эпидемическую ситуацию по бруцеллезу осложняет возможность вспышечной заболеваемости людей данным заболеванием [4]. Для человека наиболее патогенными считаются виды B. melitensis, B. abortus, B. suis и B. canis, заражение которыми может происходить при непосредственном контакте с инфицированными животными или при употреблении в пищу непастеризованных продуктов животного происхождения [5]. Необходимость применения современных молекулярно-генетических методов для проведения точной идентификации и типи-рования возбудителей бруцеллеза не вызывает сомнений. В частности, для молекулярного ти-пирования возбудителей особо опасных инфекций успешно используется MLVA (Multiple Loci VNTR Analysis), представляющий собой сравнительный анализ вариабельности областей генома (VNTR-локусов), содержащих тандемные повторы [6]. Что касается бруцелл, данный метод отражает генетический полиморфизм у штаммов Brucella sp. и позволяет не только проводить внутривидовую дифференциацию, но и группировать штаммы возбудителей бруцеллеза в соответствии с их географическим про- исхождением [7, 8]. Ранее в качестве метода генотипирования бруцелл нами был предложен MLVA протокол, включающий в себя анализ 15 VNTR-локусов, содержащих тандемные повторы от 6 до 134 п. н. [9]. Принципиальным отличием данного подхода от других существующих схем исследования геномного полиморфизма бру-целл является использование оригинальной системы VNTR-праймеров, представляющих собой претерпевшие редизайн традиционно применяемые для MLVA олигонуклеотидные затравки. Однако отметим, что ранее нами был выполнен только теоретический подбор праймеров и необходимых условий ПЦР.
Цель исследования – оценка эффективности применения разработанного протокола проведения MLVA для дифференциации штаммов возбудителя бруцеллеза.
Материалы и методы. В работе использовали данные о геномных последовательностях 49 штаммов бруцелл, представленных в базе данных GenBank (табл. 1). Биоинформационный анализ проводили с помощью программ Vector NTI 9.1 и онлайн-ресурсов NCBI (URL: .
Таблица 1
|
Штамм |
Географическое происхождение |
Номер в базе данных GenBank |
|
1 |
2 |
3 |
|
Brucella canis strain RM6/66 |
Коллекция (США) |
CP007758.1, CP007759.1 |
|
Brucella canis strain FDAARGOS_420 |
CP023974.1, CP023973.1 |
|
|
Brucella canis ATCC 23365 |
CP000872.1, CP000873.1 |
|
|
Brucella canis str. Oliveri |
HG803175.1, HG803176.1 |
|
|
Brucella canis strain 2010009751 |
США (Массачусетс) |
CP016977.1, CP016978.1 |
|
Brucella canis strain 2009013648 |
США (Аризона) |
CP016975.1, CP016976.1 |
|
Brucella canis strain 2009004498 |
США (Луизиана) |
CP016973.1, CP016974.1 |
|
Brucella canis strain SVA13 |
Швеция |
CP007629.1, CP007630.1 |
|
Brucella canis HSK A52141 |
Корея |
CP003174.1, CP003175.1 |
|
Brucella canis strain GB1 |
Китай |
CP027643.1, CP027642.1 |
|
Brucella suis 1330 |
Коллекция (США) |
AE014291.4, AE014292.2 |
|
Brucella suis bv. 1 strain CVI_59 |
Хорватия |
CP054959.1, CP054960.1 |
|
Brucella suis bv. 1 strain CVI_58 |
CP054961.1, CP054962.1 |
|
|
Brucella suis bv. 1 str. S2 |
Китай |
CP006961.1,CP006962.1 |
|
Brucella suis bv. 2 strain CVI_50 |
Хорватия |
CP054963.1, CP054964.1 |
|
Brucella suis bv. 2 strain Bs364CITA |
Португалия |
CP007697.1, CP007698.1 |
|
Brucella suis bv. 2 strain PT09172 |
CP007693.1, CP007694.1 |
|
|
Brucella suis bv. 3 str. 686 |
Коллекция (США) |
CP007719.1, CP007718.1 |
|
Brucella suis bv. 3 strain CVI_71 |
Хорватия |
CP054957.1, CP054958.1 |
|
Brucella suis bv. 4 strain CVI_72 |
CP054955.1,CP054956.1 |
|
|
Brucella suis bv. 5 strain CVI_73 |
CP054953.1,CP054954.1 |
|
|
Brucella melitensis bv. 1 str. 16M |
Коллекция (США) |
AE008917.1, AE008918.1 |
|
Brucella melitensis bv. 2 str. 63/9 |
CP007789.1, CP007788.1 |
|
|
Brucella melitensis strain 2008724259 |
США (Калифорния) |
CP016983.1, CP016984.1 |
|
Brucella melitensis strain CIIMS-NV-1 |
Индия |
CP029756.1, CP029757.1 |
|
Brucella melitensis strain BL |
Китай |
CP022875.1, CP022876.1 |
|
Brucella melitensis strain B15 |
CP035795.1, CP035796.1 |
|
|
Brucella melitensis M5-90 |
CP001851.1, CP001852.1 |
|
|
Brucella melitensis strain CIT21 |
CP025819.1, CP025820.1 |
|
|
Brucella melitensis strain C-573 |
Россия (Ставрополь) |
CP019679.1, CP019680.1 |
|
Brucella melitensis BwIM_SYR_04 |
Сирия |
CP018512.1, CP018513.1 |
|
Brucella melitensis BwIM_SYR_26 |
CP018526.1, CP018527.1 |
|
|
Brucella melitensis BwIM_IRN_28 |
Иран |
CP018484.1, CP018485.1 |
Окончание табл. 1
|
1 |
2 |
3 |
|
Brucella melitensis BwIM_IRQ_32 |
Ирак |
CP018490.1, CP018491.1 |
|
Brucella melitensis BwIM_TUR_38 |
Турция |
CP018552.1, CP018553.1 |
|
Brucella melitensis BwIM_ITA_55 |
Италия |
CP018496.1, CP018497.1 |
|
Brucella abortus 2308 |
Коллекция (США) |
AM040264.1, AM040265.1 |
|
Brucella abortus biovar 1 str. 9-941 |
AE017223.1, AE017224.1 |
|
|
Brucella abortus bv. 2 str. 86/8/59 |
CP007765.1, CP007764.1 |
|
|
Brucella abortus bv. 6 str. 870 |
CP007709.1, CP007710.1 |
|
|
Brucella abortus 104M |
Китай |
CP009625.1, CP009626.1 |
|
Brucella abortus strain clpP |
CP044338.1, CP044339.1 |
|
|
Brucella abortus strain MC |
CP022879.1, CP022880.1 |
|
|
Brucella abortus strain CIIMS-NV-4 |
Индия |
CP025743.1, CP025744.1 |
|
Brucella abortus strain IVRI95 |
CP034695.1, CP034696.1 |
|
|
Brucella abortus strain 69841 |
Италия |
CP098117.1, CP098118.1 |
|
Brucella abortus strain 24157 |
CP098085.1, CP098086.1 |
|
|
Brucella abortus A13334 |
Южная Корея |
CP003176.1, CP003177.1 |
|
Brucella abortus strain 68 |
Украина (Луганск) |
CP066175.1, CP066176.1 |
Использованные в работе штаммы бруцелл из базы данных GenBank
Множественное выравнивание выполнили с использованием алгоритма MUSCLE в программе Mega 11 . Филогенетический анали з осуществляли методом попарного невзвешенного кла-стирования с арифметическим усреднением (UPGMA). Для оценки достоверности филогенетических связей использовали многократную генерацию методом Bootstrap для 1000 независимых построений каждого филогенетического дерева .
Для постановки полимеразной цепной реакции (ПЦР) использовали выделенную нами ранее ДНК штаммов B. canis RM 6/66, B. suis 1330, B. suis 183-L и B melitensis 1565 [10, 11]. Амплификацию VNTR-локусов проводили согласно протоколу, представленному в работе Хаммадо-ва с соавт. [9]. Полученные ПЦР-продукты разделяли электрофорезом в 1,5 % агарозном геле с последующей окраской бромистым этидием. Визуализацию проводили с помощью UV- трансиллюминатора. Размер полученных фрагментов определяли с использованием программы «Gel Analyzer» путем сравнения фрагмента с маркером молекулярной массы ДНК «100 + bp DNA Ladder» (ЗАО «Евроген», Москва).
Результаты и их обсуждение. В рамках текущего исследования провели апробацию разработанного нами ранее MLVA-подхода с использованием ДНК бактерий B. canis RM 6/66, B. suis 1330, B. suis 183-L и B. melitensis 1565, а также нуклеотидных последовательностей бру-целл, представленных в базе данных GenBank. На первом этапе работы определили размеры получаемых VNTR-локусов штаммов B. canis RM 6/66, B. suis 1330, B. suis 183-L и B. meli-tensis 1565 с помощью ПЦР и последующего электрофореза. Результаты проведенного молекулярно-генетического анализа представлены в таблице 2.
Таблица 2
Характеристика VNTR - локусов исследуемых штаммов бруцелл
|
Локус |
Молекулярный размер локуса, п.н. |
|||
|
B. canis RM 6/66 |
B. suis 1330 |
B. suis 183-L |
B. melitensis 1565 |
|
|
1 |
2 |
3 |
4 |
4 |
|
Bru4 |
– |
– |
– |
– |
|
Bru6 |
273 |
273 |
273 |
273 |
|
Bru7 |
165 |
165 |
165 |
157 |
|
Bru8 |
– |
– |
– |
– |
|
Bru9 |
160 |
144 |
160 |
152 |
|
Bru10 |
– |
– |
– |
– |
Окончание табл. 2
|
1 |
2 |
3 |
4 |
5 |
|
Bru11 |
– |
– |
– |
– |
|
Bru16 |
168 |
168 |
160 |
152 |
|
Bru18 |
146 |
138 |
166 |
138 |
|
Bru19 |
170 |
164 |
184 |
178 |
|
Bru21 |
175 |
175 |
159 |
167 |
|
Bru30 |
129 |
129 |
121 |
185 |
|
Bru43 |
163 |
163 |
151 |
175 |
|
Bru43 |
188 |
188 |
134 |
152 |
Поскольку амплификация локусов Bru4, Bru8, Bru10, Bru11 и Bru55 для используемых штаммов была отрицательной, данные локусы были исключены из дальнейшей работы. Штаммы B. canis RM 6/66 и B. suis 1330 использовали в качестве референтных. Геном данных бактерий полностью секвенирован и представлен в базе данных GenBank, поэтому для B. canis RM 6/66 и B. suis 1330 с помощью биоинформационного анализа подтвердили размер оставшихся в работе десяти VNTR-локусов, после чего путем экстраполяции также определили точный молекулярный размер данных локусов для штаммов B. suis 183-L и B. melitensis 1565.
На следующем этапе работы провели MLVA для 49 штаммов бруцелл, представленных в базе данных GenBank (см. табл. 1). Для этого с помощью программы Vector NTI 9.1 нуклеотидные последовательности I и II хромосом бру-целл ограничили разработанными для аплифи-кации VNTR-локусов праймерами, после чего определили молекулярный размер соответствующего VNTR-локуса и количество повторов в нем. Далее построили дендрограмму, представленную на рисунке.
Установили, что штаммы B. canis распределились на дендрограмме по трем близкородственным кластерам, два из которых являются общими с представителями вида B. suis. В частности, самый большой кластер сформирован штаммами B. canis и B. suis (I, IV биовара), располагающимися на соседних ветках, но выходящими из разных узлов. Штамм B. canis GB1, в свою очередь, сгруппировался вместе с кладой, состоящей из двух штаммов B. suis III биовара. Данный факт свидетельствует о генетическом родстве видов B. canis и B. suis, что находит подтверждение в исследованиях отечественных и зарубежных авторов [8, 11, 12]. Что касается остальных использованных штаммов B. suis, представители II биовара данного вида сгруппировались на дендрограмме в виде клады и близкородственной к ней ветви филогенетического древа. Обособленное положение также занял штамм B. suis V биовара, что согласуется с данными литературы о таксономии бруцелл [8]. Также родственную, но удаленную от других штаммов B. suis ветвь образует штамм B. suis 183-L, использованный в исследовании для апробации MLVA-подхода.
Другой тест-штамм – B. melitensis 1565 – сгруппировался в отдельную кладу с референтным штаммом B. melitensis 16М. Штаммы B. melitensis из базы данных GenBank образуют на дендрограмме три общих c представителями вида B. abortus кластера и группируются согласно их видовой принадлежности. В частности, штаммы B. abortus А13334, 68, IVRI95, 69841, 9-941 и CIIMS-NV-4 объединены в одну подгруппу внутри самого большого кластера, остальная часть которого представлена штаммами B. melitensis различного географического происхождения.
Штаммы B. abortus 86/8/59 и 104М образуют самостоятельную кладу, выходящую из общего узла со штаммами B. melitensis B15, M5-90, Bwl V IRQ 32. Аналогичным образом в другом кластере расположились штаммы B. abortus 870 и МС. Отдельно расположился кластер, сформированный коллекционным штаммом B. abortus 2308 и кладой, представленной изолятами из Китая ( B. abortus clP) и Италии ( B. abortus 24157).
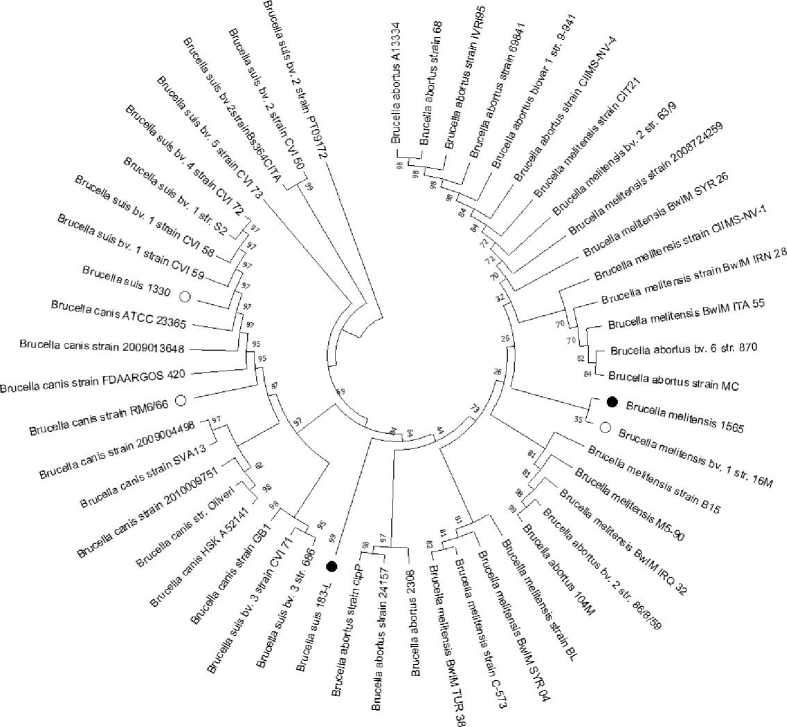
Дендрограмма, иллюстрирующая кластеризацию исследованных штаммов бруцелл на основании проведенного MLVA-анализа (черными кругами обозначены штаммы, для которых VNTR-профиль определен in vitro; белыми – референтные штаммы)
Заключение . Проведенный по десяти вариабельным локусам MLVA-анализ позволяет систематизировать большинство использованных из базы данных GenBank штаммов бруцелл в соответствии с их таксономическим положением. Из чего можно сделать вывод, что данный подход к MLVA-типированию может быть использован для дифференциации представителей рода Brucella при проведении эпизоотологических расследований вспышек бруцеллеза.
Список литературы Дифференциация штаммов бруцелл на основе анализа вариабельности VNTR-локусов
- Анализ заболеваемости людей бруцеллезом и молекулярно-биологическая характеристика изолятов Brucella melitensis на длительно неблагополучных по бруцеллезу территориях юга европейской части России / А.А. Хачатурова [и др.] // Журнал микробиологии, эпидемиологии и иммунобиологии. 2022. 99(1). С. 63-74. DOI: 10.36233/ 0372-9311-185.
- Салмаков К.М., Косарев М.А. Совершенствование системы специфической профилактики бруцеллеза крупного рогатого скота с применением вакцины из штамма B. abortus 82 и препарата из штамма B. abortus R-1096 // Ветеринария. 2023. № 8. С. 9-13. DOI: 10.30896/0042-4846.2023.26.8.09-13.
- Прошлое, настоящее, перспективы и проблемы совершенствования специфической профилактики бруцеллеза / В.А. Коршенко [и др.] // Медицинский вестник Юга России. 2021.№ 12 (3). С. 12-21. DOI: 10.21886/ 2219-8075-2021-12-3-12-21.
- Анализ заболеваемости бруцеллезом и молекулярно-генетическая характеристика популяции бруцелл на территории Российской Федерации / Д.Г. Пономаренко [и др.] // Проблемы особо опасных инфекций. 2023. № 2. С. 61-74. DOI: 10.21055/0370-1069-2023-261-74.
- In vitro antimicrobial susceptibility testing of human Brucella melitensis isolates from Qatar between 2014-2015 / A. Deshmukh [et al.] // BMC Microbiol. 2015. № 15 (121). DOI: 10.1186/s12866-015-0458-9.
- Дифференциация штаммов Bacillus anthra-cis методом анализа температур плавления продуктов ПЦР, полученных после амплификации VNTR локусов / Н.А. Фахрутдинов [и др.] // Ветеринарный врач. 2023. № 2. С. 41-46. DOI: 10.33632/1998-698X_2023_ 2_41.
- Изучение генетического разнообразия штаммов бруцелл, выделенных в СевероКавказском федеральном округе / И.В. Кузнецова [и др.] // Проблемы особо опасных инфекций. 2017. № 3. C. 58-62. DOI: 10.21055/0370-1069-2017-3-58-62.
- Кулаков Ю.К., Цирельсон Л.Е., Желудков М.М. Молекулярно-генетическая характеристика изолятов бруцелл, выделенных от собак и оленей в различных регионах России // Молекулярная генетика, микробиология и вирусология. 2012. № 4. С. 2833. DOI: 10.3103/S0891416812040052.
- Маркерные локусы генома бруцелл для дифференциальной ПЦР индикации патогенных штаммов / Н.И. Хаммадов [и др.] // Проблемы особо опасных инфекций. 2018. № 3. С. 88-93. DOI: 10.21055/0370-10692018-3-88-93.
- Использование кривых плавления амплико-нов VNTR-локусов для идентификации бруцелл / Е.А. Анисимова [и др.] // Перспективы развития современной ветеринарной науки: сб. науч. тр. по итогам Всерос. науч.-практ. конф. с междунар. участием, посвящ. 55-летию Прикаспийского зонального научно-исследовательского ветеринар. ин-та - филиала ФГБНУ «ФАНЦ РД» (22-23 сентября 2022 г.). Махачкала: Алеф, 2022. С. 22-26.
- Применение HRM-анализа кривых плавления, полученных после амплификации VNTR-локусов, для идентификации и дифференциации штаммов бруцелл / Е.А. Анисимова [и др.] // Проблемы особо опасных инфекций. 2023. № 4. С. 42-49. DOI: 10.21055/0370-1069-2023-4-42-49.
- Genetic and Phenotypic Characterization of the Etiological Agent of Canine Orchiepididymitis Smooth Brucella sp. BCCN84.3 / C. Guzman-Verri [et al.] // Front. Vet. Sci. 2019. № 6 (175). DOI: 10.3389/fvets.2019.00175.


