Филогенетический анализ российских изолятов вируса инфекционной анемии лошадей с использованием оптимизированной двухраундовой ПЦР
Автор: Герасимова Н.Н., Колбасова О.Л., Цыбанов С.Ж., Колбасов Д.В.
Журнал: Сельскохозяйственная биология @agrobiology
Рубрика: Вирусы - возбудители особо опасных инфекций: молекулярная генетика, иммуногенность, культивирование
Статья в выпуске: 6 т.50, 2015 года.
Бесплатный доступ
Инфекционная анемия лошадей (ИНАН) - болезнь однокопытных, вызываемая вирусом рода Lentivirus семейства Retroviridae. Заболевание широко распространено, и его эпизоотии зарегистрированы практически на всех континентах земного шара. В Российской Федерации из года в год при плановых исследованиях на присутствие антител к вирусу ИНАН выявляются животные, серопозитивные в реакции диффузионной преципитации (РДП). В настоящее время опасность распространения вируса ИНАН признается во всем мире, а его контролю уделяется большое внимание, в связи с чем проводятся многочисленные исследования по выявлению вируса, определению его молекулярно-генетических характеристик и происхождения. В настоящей работе представлены данные по разработке тест-системы на основе ПЦР, позволяющей не только выявлять геном вируса ИНАН в различных образцах биологического материала, но и проводить дальнейшее секвенирование фрагментов генома этого инфекционного агента. Геном вируса ИНАН состоит из трех основных генов - gag, pol, env (распложены в направлении 5'→3'), из которых наиболее полезным маркером для изучения генетического разнообразия и филогенетических взаимоотношений между различными штаммами вируса ИНАН считается gag-ген, необходимый для внутриклеточной сборки и выхода вируса из клетки. С целью проведения молекулярно-генетических исследований нами была оптимизирована двухраундовая ПЦР без этапа обратной транскрипции и рассчитаны специфические олигонуклеотидные праймеры для второго раунда реакции, фланкирующие участок gag-гена провирусной ДНК вируса ИНАН размером 1018 п.н. В результате проведенных изысканий также были определены нуклеотидные последовательности и филогенетические отношения ранее выделенного отечественного штамма вируса ИНАН и двух современных изолятов, выявленных на территории нашей страны. Полученные данные позволили отнести штамм 3-К-ВНИИТИБП-ВИЭВ к группе североамериканского происхождения (гомология 98 %), а изоляты Нижний Новгород-2011 и Омск-2012 - к группе европейских (гомология 82-83 %). Таким образом, разработанная нами система идентификации вируса ИНАН с помощью ПЦР и филогенетического анализа позволяет устанавливать принадлежность выделяемых изолятов к определенной геногруппе и, следовательно, предположить их географическое происхождение. Эта информация необходима для определения источника и путей распространения возбудителя с целью прогнозирования эпизоотической ситуации и планирования мер борьбы с болезнью.
Инан, пцр, филогенетический анализ
Короткий адрес: https://sciup.org/142133644
IDR: 142133644 | УДК: 636.1:619:616.155.194:578:577.2.08 | DOI: 10.15389/agrobiology.2015.6.803rus
Текст научной статьи Филогенетический анализ российских изолятов вируса инфекционной анемии лошадей с использованием оптимизированной двухраундовой ПЦР
Инфекционная анемия лошадей (ИНАН) — болезнь однокопытных, которая сопровождается поражением органов кроветворения и проявляется рецидивирующей или постоянной лихорадкой, анемией и нарушением функций сердечно-сосудистой системы (1-4). Этиологический агент ИНАН имеет вирусную природу (Equine infectious anemia virus — EIAV) и относится к роду Lentivirus семейства Retroviridae (4, 5). Геном вируса ИНАН представлен двумя идентичными молекулами линейной одноцепочечной РНК позитивной полярности (4, 6-8) и состоит из трех основных структурных генов, расположенных последовательно в направлении 5′→3′, — gag (кодирует вирионные коровые белки, необходим для внутриклеточной сборки и выхода вируса из клетки), pol (кодирует полипептиды, обладающие ревертазной, специфической РНК-азной и ДНК-эндонуклеазной активностью) и env (кодирует поверхностные вирионные гликопротеины, участвует в формировании оболочки вириона, специфическом узнавании клетки-хозяина и проникновении вируса через ее мембрану) (4, 8, 9). Кроме того, геном вируса ИНАН содержит три дополнительные открытые рамки считывания, с которых экспрессируются небольшие вспомогательные белки, участвующие в регуляции синтеза и процессинге вирусной РНК, — tat, rev и S2. Вспомогательный белок S2 — важная детерминанта репликации и патогенных свойств вируса ИНАН in vivo (4, 6, 8-10). Нуклеотидная последовательность вирусного генома на обоих концах имеет длинные концевые повторы (LTR), необходимые для встраивания провирусной ДНК (пДНК) в клеточный геном (8, 11).
К вирусу ИНАН восприимчивы представители семейства лошадиных ( Equidae ) — лошади, пони, ослы, мулы и лошаки (1, 2, 12-15). Заболевание инфекционной анемией негативно сказывается на промышленном разведении лошадей. В числе 11 инфекций однокопытных ИНАН входит в список World Organization for Animal Health (Международное эпизоотическое бюро, Париж, OIE — МЭБ), включающий заболевания, о вспышках которых страны обязаны информировать МЭБ (8, 16).
В связи с регистрацией болезни практически на всех континентах (17, 18-20) большое внимание уделяется проблеме контроля за распространением вируса ИНАН. Проводимый в разных странах филогенетический анализ местных изолятов и штаммов позволяет предположить, что существуют генетические подтипы вируса ИНАН, циркулирующие в настоящее время в мире (8, 14, 17).
В Российской Федерации из года в год при плановых исследованиях на присутствие антител к вирусу ИНАН выявляются животные, серопозитивные в реакции диффузионной преципитации (РДП). Так, с 2011 по 2015 год сероположительные лошади обнаружены более чем в 25 регионах России (21-23).
Несмотря на то, что случаи выявления положительно реагирующих в РДП на ИНАН животных в Российской Федерации регистрируются регулярно, в нашей стране единственным известным вариантом вируса ИНАН остается штамм З, выделенный в Запорожье в 1967 году К.П. Юровым. Позднее этот штамм был адаптирован к культурам клеток и использован в качестве культурального под названием 3-К-ВНИИТИБП-ВИЭВ (коллекция Всероссийского научно-исследовательского и технологического института биологической промышленности, Московская обл.).
Важно отметить, что молекулярная эпизоотология вируса ИНАН в России до настоящего времени не исследована. В то же время молекулярно-генетическая оценка разнообразия изолятов вируса ИНАН и их филогенетический анализ необходимы для понимания особенностей появления и циркуляции этого вируса в нашей стране, а отсутствие таких данных служит фактором, осложняющим эпизоотическую ситуацию по ИНАН. Кроме того, изучение разнообразия российских изолятов вируса ИНАН существенно расширяет представления о генетической структуре его мировой популяции.
Нашей целью стала разработка тест-системы на основе полимеразной цепной реакции (ПЦР), позволяющей выявлять вирус ИНАН, проводить секвенирование и анализ фрагментов его генома, а также исследовать молекулярно-генетические характеристики и филогенетические связи между известными и вновь выявляемыми изолятами этого вируса, циркулирующими на территории России.
Методика . Изучали единственный известный отечественный штамм вируса ИНАН 3-К-ВНИИТИБП-ВИЭВ (Всероссийский научно-исследовательский и технологический институт биологической промышленности, Московская обл.) и два изолята, выявленные в мониторинговых исследованиях на территории РФ, — Нижний Новгород-2011 и Омск-2012 (коллекция Всероссийского НИИ ветеринарной вирусологии и микробиологии).
Множественное выравнивание и расчет праймеров для секвенирования были выполнены с помощью пакетов программ BioEdit v. 7.0 804
и Oligo v. 6.0 («Molecular Biology Insights, Inc.», США), используя нуклеотидные последовательности генома разных штаммов и изолятов вируса ИНАН, опубликованные в базе данных GenBank (the National Center for Biotechnology Information — NCBI).
Нуклеиновые кислоты выделяли из анализируемого биологического материала в соответствии с методикой, основанной на применении коммерческого реагента Trizol LS («Invitrogen», США) согласно рекомендациям производителя.
С целью накопления специфических продуктов для последующего секвенирования проводили двухраундовую (гнездовую) ПЦР без этапа обратной транскрипции с праймерами, описанными S. Capomaccio с соавт. (13) (для первого раунда ПЦР), и оригинальными праймерами (для второго раунда ПЦР). Постановку гнездового варианта ПЦР и оптимизацию температурных режимов реакций осуществляли в термоциклере Gradient Palm Cycler («Corbett Research», Австралия).
Продукты реакции разделяли методом электрофореза в 1,5 % агарозном геле, содержащем 0,001 % бромистого этидия. Специфические продукты амплификации экстрагировали из агарозного геля, используя коммерческие наборы AxyPrep DNA Gel Extraction Kit («Axygen Scientific, Inc.», США) и QIAquick Gel Extraction Kit («Qiagen N.V.», Германия).
Секвенирование амплифицированных фрагментов ДНК по Сэнгеру выполняли в автоматическом анализаторе Applied Biosystems Genetic Analyzer 3130 («Applied Biosystems», США) с использованием набора реактивов Big Dye v. 3.1. Terminator и 5½Sequencing buffer («Applied Biosystems», США) в соответствии с прилагаемой инструкцией.
Филогенетический анализ нуклеотидных последовательностей проводили с помощью алгоритма Neighbor-Joining с применением численного ресамплинга, или методов генерации повторных выборок, в программе MEGA v. 5.0 (24).
Результаты . Наиболее информативным маркером для изучения патогенности и генетического разнообразия между различными штаммами вируса ИНАН считается gag -ген, кодирующий основные матричные и капсидные белки, в связи с чем он часто используется в молекулярно-генетических исследованиях (13, 14, 17). Поэтому для оценки филогенетического родства между выявленными на территории России и зарубежными штаммами и изолятами вируса ИНАН, геномы которых представлены в GenBank, мы определили нуклеотидную последовательность именно этого гена.
Для амплификации фрагмента gag -гена были использованы праймеры, описанные S. Capomaccio с соавт. (13). Эти праймеры (EIAV 1B и EIAV 5) фланкируют область вирусного генома размером 2233 п.н., включающую участок гена tat от экзона 1, полный gag -ген и начало нуклеотидной последовательности рol -гена. Постановку ПЦР осуществляли при градиенте температур 50-59 ° С; положительным контролем служила нуклеиновая кислота штамма 3-К-ВНИИТИБП-ВИЭВ. Однако анализ электрофореграмм полученных амплифицированных фрагментов показал, что используемые праймеры работают только при очень низкой температуре отжига (50-54 ° С) и дают несколько неспецифических продуктов.
Чтобы увеличить выход специфического продукта, мы рассчитали олигонуклеотидные последовательности праймеров для второго раунда гнездовой ПЦР. Эти праймеры (EA Fw/N2 и EA Rev/N2) фланкируют участок gag-гена размером 1018 п.н. внутри фрагмента, который, в свою очередь, фланкируют праймеры EIAV 1B и EIAV 5. Для постановки ПЦР с рассчитанными праймерами нами были подобраны условия: состав смеси, кон- центрация праймеров, dNTPs, количество вносимой полимеразы и температурно-временные режимы.
В пределах от 51 до 60 ° С температура 56 ° С была оптимальной для гибридизации EA Fw/N2 и EA Rev/N2 (рис. 1). Смесь для амплификации включала праймеры (по 10 пмоль каждого: EIAV 1B и EIAV 5 — для первого раунда, EA Fw/N2 и EA Rev/N2 — для второго) — 2,0 мкл; dNTPs («Fermentas», Латвия) (10 мМ) — 1 мкл; 5½ буфер для ПЦР из набора реагентов АмплиСенс Blue (Россия) — 5 мкл; Taq ДНК-полимеразу («Евроген», Россия) (2,5 ед.) — 0,4 мкл; исследуемый образец нуклеиновой кислоты — 8 мкл; деионизированная вода — до 25 мкл. В гнездовой ПЦР первый раунд амплификации (с праймерами EIAV 1B и EIAV 5) проводили при 52 ° С, второй (с праймерами EA Fw/N2 и EA Rev/N2) — при 56 ° С. Праймеры EA Fw/N2 и EA Rev/N2 также использовались для секвенирования амплифицированных фрагментов ДНК.
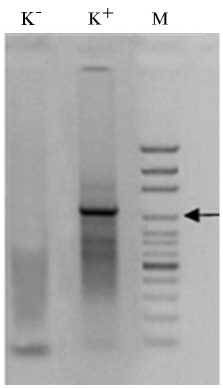
Рис. 1. Электрофореграмма продуктов амплификации участка gag -гена (1018 п.н.) вируса инфекционной анемии лошадей во втором раунде ПЦР с рассчитанными олигонуклеотидными праймерами: К - — отрицательный контроль, К + — штамм 3-К-ВНИИТИБП-ВИЭВ; М — маркер молекулярных масс 100 bp Ladder DNA Marker («Axygen», США). Стрелкой отмечен фрагмент 1000 п.н.
Оптимизированный протокол двухраундовой ПЦР позволил перейти к основной задаче работы — впервые определить молекулярно-генетические характеристики штамма 3-К-ВНИИТИБП-ВИЭВ и двух изолятов вируса ИНАН (Нижний Новгород-2011 и Омск-2012), выявленных при вспышках болезни в Российской Федерации (25), на основе анализа нуклеотидных последовательностей фрагмента gag -гена (участок размером 1018 п.н.).
При сравнении участков геномов отечествен ного штамма и изолятов с таковыми у референтных зарубежных штаммов из GenBank с помощью программы BioEdit v. 7.0 были выявлены многочисленные нуклеотидные замены. На основании множественного выравнивания и анализа полученных нами и размещенных в GenBank нуклеотидных последовательностей генома вируса ИНАН с помощью программы MEGA v. 5.0 была построена дендрограмма, отражающая филогенетические отношения изолятов и штаммов, взятых для сравнения (рис. 2).
Как видно из дендрограммы, штамм 3-К-ВНИИТИБП-ВИЭВ входит в один кластер со штаммами V70 и V26 (степень нуклеотидной идентичности — 98 %). По данным литературы, штаммы V70 и V26, выделенные в Японии, получены при пассировании североамериканского штамма EIAVWyoming (Вайоминг) на естественно восприимчивых животных и входят с ним в одну генетическую группу (8, 9) с уровнем внутригрупповых различий, не превышающим 5 %. Гомология нуклеотидной последовательности участка gag -гена штаммов 3-К-ВНИИТИБП-ВИЭВ и EIAVWyoming также составила 98 %.
Изоляты, выявленные на территории России в период с 2012 по 2014 год (Нижний Новгород-2011 и Омск-2012), оказались филогенетически наиболее близки к группе европейского происхождения. Самую высокую гомологию по нуклеотидным последовательностям участка gag -гена (1018 п.н.) отмечали между изученными российскими изолятами и 806
образцами из Германии (83 % для изолята EIAVGER-7) и Италии (82 % для изолята Ita87).
|AF033820.1|strain Wyoming (USA)
NC 001450.1 |NCBI Reference Sequence(USA)
|AF016316.1|EIAVuk (United Kingdom)
|AF247394.1| Equine infectious anemia virus complete genome (USA)
strain 3K-VNIITIBP-VIEV
6Яг |AB008196.1 |strain V70(Japan) 541— |AB008197.1|strain V26(Japan)
- |EF418582.1 |strain Can1 (Canada)
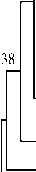
--------------------|HM177435.1| isolate 71 (Italy)
& |HM 177438.11 isolate 73 (Italy)
|EU741609.1|EIAV clone lta-4 (Italy)
|EU240733.1|EIAV clone lta-1 (Italy)
- isolate Niz.Novgorod-2011
----isolate Omsk-2012
|KF878269.11 isolate EIAVGER-7 (Germany)
-------IHQ888861.11 isolate Ita87 (Italy)
100 I-------------|GQ923952.1| EIAV clone Rom-2 (Roman)
I---------------|GU060663.1| EIAV clone Rom-5 (Roman)
----------------------|JX193070.1|strain Assebroek (Belgium)
-----------------------|HQ888862.1| isolate lta90 (Italy)
---------------------------JX003263.1 |strain Miyazaki2011-A (Japan)
-------|AB693823.1|strain Goshun (Japan)
86 r |GU385359.1| isolate FDDV-9 tat (China)
"L |GU385357.1| isolate FDDV-7 (China)
)91 |AF327877.1|strain Liaoning (China)
|GU385365.1| isolate LN-5 (China)
-
- IHM141910.11 isolate DV10-3 (China)
-
■ |HM141913.11 isolate DLV3-A (China)
|AF327878.1|vaccine strain (China)
47L |HM141921.11 isolate DLV5-10 (China)
Рис. 2. Дендрограмма, отражающая филогенетические отношения между различными штаммами и изолятами вируса инфекционной анемии лошадей. Построена на основе анализа нуклеотидных последовательностей участков gag -гена (1018 п.н.) с использованием алгоритма Neighbor-Joining в программе MEGA v. 5.0.
Нуклеотидные последовательности участка gag -гена штамма 3-К-ВНИиТИБП-ВИЭВ и изолята Нижний Новгород-2011 вируса ИНАН были депонированы в международный банк данных GenBank (¹ KM202106 и (¹ KM248275) и в настоящее время доступны для других исследователей.
Таким образом, рассчитаны оригинальные праймеры и оптимизированы условия постановки гнездовой ПЦР, что позволило амплифициро-вать специфический участок (1018 п.н.) генома вируса инфекционной анемии лошадей в области gag -гена, широко используемого для оценки генетического разнообразия и родства изолятов. При этом нами впервые определен фрагмент нуклеотидных последовательностей геномов отечественного штамма вируса ИНАН и двух современных изолятов, обнаруженных на территории России. Данные выполненного секвенирования и филогенетического анализа позволили отнести штамм 3-К-ВНИИТИБП-
ВИЭВ к группе североамериканского происхождения. Кроме того, нам удалось установить наибольшую гомологию изолятов Нижний Новгород-2011 и Омск-2012 с изолятами европейского происхождения.
Список литературы Филогенетический анализ российских изолятов вируса инфекционной анемии лошадей с использованием оптимизированной двухраундовой ПЦР
- Архипов Н.И., Бакулов И.А., Соковых Л.И. Медленные инфекции животных. М., 1987.
- Юров К.П., Заблоцкий В.Т., Косминков Н.Е. Вирусные болезни лошадей. М., 2010.
- Nagarajan M.M., Simard C. Detection of horses infected naturally with equine infectious anemia virus by nested polymerase chain reaction. J. Virol. Methods, 2001, 94: 97-109 ( ) DOI: 10.1016/S0166-0934(01)00283-X
- Leroux C., Cadoré J.L., Montelaro R.C. Equine Infectious Anemia Virus (EIAV): what has HIV's country cousin got to tell us? Vet. Res., 2004, 35(4): 485-512 ( ) DOI: 10.1051/vetres:2004020
- Sellon D.C., Fuller F.J., McGuire T.C. The immunopathogenesis of equine infectious anemia virus. Virus Res., 1994, 32(2): 111-138 ( ) DOI: 10.1016/0168-1702(94)90038-8
- Инфекционная патология животных (в 2 т.)/Под ред. А.Я. Самуйленко, Б.В. Соловьева, Е.А. Непоклонова, Е.С. Воронина. М., 2006.
- Сергеев B.A., Непоклонов Е.А., Алипер T.И. Вирусы и вирусные вакцины. М., 2007.
- Cook R.F., Leroux C., Issel C.J. Equine infectious anemia and equine infectious anemia virus in 2013: а review. Vet. Microbiol., 2013, 167(1-2): 181-204 ( ) DOI: 10.1016/j.vetmic.2013.09.031
- Dong J.B., Zhu W., Cook F.R., Goto Y., Horii Y., Haga T. Identification of a novel equine infectious anemia virus field strain isolated from feral horses in southern Japan. J. Gen. Virol., 2013, 94: 360-365 ( ) DOI: 10.1099/vir.0.047498-0
- Fagerness A.J., Flaherty M.T., Perry S.T., Jia B., Payne S.L., Fuller F.J. The S2 accessory gene of equine infectious anemia virus is essential for expression of disease in ponies. Virology, 2006, 349(1): 220-230 ( ) DOI: 10.1016/j.virol.2005.12.041
- Payne S.L., La Celle K., Pei X.F., Qi X.M., Shao H., Steagall W.K., Perry S., Fuller F. Long terminal repeat sequences of equine infectious anaemia virus are a major determinant of cell tropism. J. Gen. Virol., 1999, 80: 755-759 ( ) DOI: 10.1099/0022-1317-80-3-755
- Ataseven V.S., Arslan H.H. Equine infectious anemia in mules, donkeys, and horses: Epidemiologic studies in the different geographic regions of Turkey. Journal of Equine Veterinary Science, 2005, 25(10): 439-441 ( ) DOI: 10.1016/j.jevs.2005.09.007
- Capomaccio S., Willand Z.A., Cook S.J., Issel C.J., Santos E.M., Reis J.K., Cook R.F. Detection, molecular characterization and phylogenetic analysis of full-length equine infectious anemia (EIAV) gag genes isolated from Shackleford Banks wild horses. Vet. Microbiol., 2012, 157: 320-332 ( ) DOI: 10.1016/j.vetmic.2012.01.015
- Cappelli K., Capomaccio S., Cook F.R., Felicetti M., Marenzoni M.L., Coppola G., Verini-Supplizi A., Coletti M., Passamonti F. Molecular detection, epidemiology, and genetic characterization of novel European field isolates of equine infectious anemia virus. J. Clin. Microbiol., 2011, 4(1): 27-33 ( ) DOI: 10.1128/JCM.01311-10
- Cook S.J., Cook R.F., Montelaro R.C., Issel C.J. Differential responses of Equus caballus and Equus asinus to infection with two pathogenic strains of equine infectious anemia virus. Vet. Microbiol., 2001, 79(2): 93-109 ( ) DOI: 10.1016/S0378-1135(00)00348-5
- Manual of diagnostic tests and vaccines for terrestrial animals. Chapter 2. 5. 6. Equine infectious anaemia, 2013 (http://www.oie.int/international-standard-setting/terrestrial-manual/access-online).
- Capomaccio S., Cappelli K., Cook R.F., Nardi F., Gifford R., Maren-zoni M.L., Passamonti F. Geographic structuring of global EIAV isolates: a single origin for New World strains? Virus Res., 2012, 163(2): 656-659 ( ) DOI: 10.1016/j.virusres.2011.11.011
- List of countries by disease situation, 2015 (http://www.oie.int/wahis_2/pu-blic/wahid.php/Diseaseinformation/statuslist).
- Bicout D.J., Carvalho R., Chalvet-Monfray K., Sabatier P. Distribution of equine infectious anemia in horses in the north of Minas Gerais State, Brazil. Journal of Veterinary Diagnostic Investigation, 2006, 18(5): 479-482 ( ) DOI: 10.1177/104063870601800511
- Pagamjav O., Kobayashi K., Murakami H., Tabata Y., Miura Y., Boldbaatar B., Sentsui H. Serological survey of equine viral diseases in Mongolia. Microbiol. Immunol., 2011, 55(4): 289-292 ( ) DOI: 10.1111/j.1348-0421.2011.00312.x
- В Омской области среди лошадей началась инфекционная анемия. Россельхознадзор, 2015 (http://www.fsvps.ru/fsvps/press/4175.html).
- Усикова Т.Н., Сергеева М.И. Инфекционная анемия лошадей в Республике Хакасия. В сб.: Проблемы развития АПК Саяно-Алтая. Абакан, 2008: 101-102.
- Detailed country(ies) disease incidence, 2015 (http://www.oie.int/wa-his_2/public/wahid.php/Diseaseinformation/statusdetail).
- Tamura K., Peterson D., Peterson N., Stecher G., Nei M., Kumar S. MEGA5: Molecular evolutionary genetics analysis using maximum likelihood, evolutionary distance, and maximum parsimony methods. Mol. Biol. Evol., 2011, 28(3): 2731-2739 ( ) DOI: 10.1093/molbev/msr121
- Герасимова Н.Н., Колбасова О.Л., Янжиева Д.В., Конвисарева Т.С., Еремец В.И. Использование серологических и молекулярно-генетических методов при исследовании на ИНАН проб от однокопытных животных Российской Федерации. В сб.: Научные основы производства и обеспечения качества биологических препаратов для АПК. Щелково, 2014: 96-100.


