Генетическая дифференциация индеек разных пород по микросателлитным маркерам
Автор: Фисинин В.И., Селионова М.И., Ковалев Д.А., Шинкаренко Л.А.
Журнал: Сельскохозяйственная биология @agrobiology
Рубрика: Генетика и геномика
Статья в выпуске: 4 т.56, 2021 года.
Бесплатный доступ
Ению важная роль отводится изучению генетических особенностей, в том числе с использованием микросателлитных локусов. При реализации международного проекта по изучению генетического биоразнообразия домашних животных (Global Project for the Measurement of Domestic Animal Genetic Diversity, MoDAD) по микросателлитным маркерам было исследовано 50 популяций разных видов птицы. Изучение биоразнообразия индеек на первоначальном этапе проводилось с использованием микросателлитных локусов курицы ( Gallus gallus ), затем были установлены информативные локусы для генома индеек ( Meleagris gallopavo ). Накоплены данные о генетических профилях, сходстве, различии и межпородной дифференциации пород индеек, разводимых в США, Италии, Венгрии и других странах. В настоящей работе впервые установлено генетическое взаимоотношение между породами индеек российской селекции и генофондной популяции университета Миннесоты на основе микросателлитных маркеров. Показано, что величина генетических дистанций между породами во многом определяется их происхождением, ареалом разведения, а также вкладом генофонда одних пород при создании и совершенствовании продуктивных качеств других. Цель работы - изучить генетическое разнообразие и межпородную дифференциацию индеек российской и зарубежной селекции с использованием микросателлитных локусов. Работа выполнялась на Северо-Кавказской зональной опытной станции по птицеводству в 2019 году. У 30 особей каждой из семи пород индеек ( Meleagris gallopavo ) отечественной селекции (белой широкогрудой, BSH; бронзовой северокавказской, BrSK; белой северокавказской, BeSK; серебристой северокавказской, SSK; московской белой, MB; черной тихорецкой, CHT; узбекской палевой, UP) были отобраны образцы крови. ДНК выделяли в соответствии с протоколом к коммерческому набору АмплиПрайм ДНК-сорб-В («ИнтерЛабСервис», Россия). Количество и качество выделенной ДНК контролировали с использованием спектрофотометра NanoDrop 2000 («Thermo Scientific», США) стандартным спектрофотометрическим методом. Генотипирование проводили по 12 микросателлитным локусам MNT9-MNT20. Для сравнения с генотипами индеек отечественных пород использовали описанные генотипы индеек (AM) генофондной фермы университета Миннесоты (Nicholas Turkey Breeding Farms). Вычисляли среднее число и число эффективных аллелей на локус (Na, Ne), степень наблюдаемой и ожидаемой гетерозиготности (Но, Не), индекс Шеннона (I). Генетическую структуру популяций оценивали на основании значений Fst и генетических дистанций по M. Nei. Для построения филогенетического дерева применяли метод ближайшего соседа (Neighbor Joining Method). Российские породы индеек и популяция АМ характеризовались низким генетическим разнообразием. Число выявленных аллелей в локусах микросаттеллитов в целом по породной выборке варьировало от 1 до 4, число аллелей на один локус колебалось от 1,0 до 1,83. Наименьшее генетическое различие установлено между породами MB и BSH. Породы BeSK, SSK и BrSK сформировали отдельный узел, при этом наибольшее генетическое удаление проявляла BrSK, образуя наибольшую по генетическому расстоянию ветвь. В отдельные ветви на относительно равном удалении выделились породы CHT, UP и популяция АМ. Таким образом, получено подтверждение, что генофонд исследованных пород и популяций домашних индеек характеризуется незначительным генетическим разнообразием по сравнению с генофондом других видов сельскохозяйственных животных.
Породы индеек, микросателлиты, филогенетический анализ, генетическое разнообразие
Короткий адрес: https://sciup.org/142231375
IDR: 142231375 | УДК: 636.592:575.174:577.21 | DOI: 10.15389/agrobiology.2021.4.651rus
Текст научной статьи Генетическая дифференциация индеек разных пород по микросателлитным маркерам
Морфофункциональное строение дыхательной системы млекопитающих широко изучено, в частности исследованы морфогенез мерцательного и реснитчатого эпителия (1-3), механизмы мукоцилиарного аппарата (4, 5), в то время как особенностям анатомического строения респираторных органов птиц уделено значительно меньше внимания (6, 7). Исследовано гистологическое строение дыхательных путей разных видов птиц (8, 9), в том числе морфология трахеи и гортани перепелов (10), бройлеров (11), индеек (12) и цесарок (13, 14), а также легких и воздухоносных мешков гусей, индеек, кур, уток (15).
Функции респираторной системы птиц заключаются в газообмене и поддержании в организме температурного гомеостаза. Трахея проводит воздух из гортани к легким, при этом воздух увлажняется, согревается и очищается от механических частиц, бактерий и вирусов. У птиц (как и у млекопитающих) трахея состоит из слизистой оболочки, подслизистой основы, фиброзно-хрящевой оболочки и адвентиции (9, 16). Слизистая оболочка выстлана многорядным мерцательным эпителием, в составе которого есть несколько видов клеток. Основная масса — это высокие реснитчатые клетки, доходящие до края пласта и несущие на апикальном полюсе реснички. Также среди клеток эпителия встречаются бокаловидные клетки — простые одноклеточные железы, выделяющие слизистый секрет (1).
Соотношение разных типов клеток зависит от вида животного, возраста, состояния здоровья и внешних условий. О.А. Матвеев с соавт. (11) при исследовании трахеи цыплят-бройлеров кросса Ross 308 определили соотношение бокаловидных к остальным клеткам эпителия как 1:10. Под эпителием располагается собственная пластинка слизистой, которая сформирована рыхлой соединительной тканью. В ней залегают коллагеновые, эластические и ретикулярные волокна, между которыми находится основное аморфное вещество (12). Далее расположена подслизистая основа, тоже образованная рыхлой соединительной тканью. Некоторые авторы описывают собственную пластинку и подслизистую основу как плотную соединительную ткань (9). За ней следует фиброзно-хрящевая оболочка, состоящая из гиалиновых колец, между которыми находится плотная соединительная ткань. У птиц хрящи трахеи замкнутые, заходят друг за друга и у некоторых видов могут окостеневать (12, 16). Слизь, выделяемая бокаловидными клетками и трахеальными железами, вязкая, содержит ШИК-положительные гликопротеиды и гликозаминокликаны, которые окрашиваются альциано-вым синим (1). Гликозаминогликаны представляют собой высокосульфати-рованные полисахариды, образующие с молекулами белка протеогликаны (17). Эта слизь находится поверх ресничек, и к ней прилипают механические частички и бактерии, попадающие с вдыхаемым воздухом. Деятельность реснитчатого аппарата, бокаловидных клеток и трахеальных желез отличается синхронностью и формирует мукоцилиарную систему транспорта в трахее и бронхах, тем самым создавая защитный механизм. Нарушения в этой системе приводят к воспалительной реакции (1).
Выполняя защитные функции, трахея в первую очередь оказывается подвержена повреждению и изменению гистологической структуры под влиянием окружающей среды. Так, отмечена способность мерцательного эпителия при агрессивных внешних воздействиях (пары ядовитых веществ, механические и термические воздействия) преобразовываться в типичный многослойный эпителий (1). В литературе описано угнетающее действие радиоактивного излучения на все элементы стенки трахеи (7) и отрицательное влияние углекислого газа, приводящее к отекам слизистой (2). В ряде работ отмечено влияние низких температур на анатомо-гистологическое строение трахеи (4, 18, 19).
В большинстве случаев возникновению неинфекционных респираторных заболеваний бройлеров способствует несоответствие условий со- держания физиологическим нормам развития птицы (20). Аллергические реакции респираторной системы птиц происходят не только из-за наличия патогенной микрофлоры в воздушно-пылевом биоаэрозоле (21, 22), но и вследствие недостаточной циркуляции воздуха в закрытых производственных помещениях (23, 24).
В настоящее время проведено достаточно исследований гистологической структуры дыхательной системы у сельскохозяйственных птиц в зависимости от резистентности особей к респираторным заболеваниям (25), заражения инфекционным бронхитом кур (26, 27) и вирусом ларинготрахеита (28, 29), контаминации воздуха (20), равномерности микроклимата (30, 31), концентрации вредных газов (32, 33), однако не изучена гистологическая и гистохимическая структура трахеальной стенки птиц, выращиваемых в закрытых помещениях с разной циркуляцией воздуха.
В представленной работе впервые показано, что циркуляция воздуха в закрытых помещениях способствует поддержанию продуктивности, улучшению гистологической структуры и гистохимического статуса трахеальной стенки цыплят-бройлеров.
Цель эксперимента — определение гистологической и гистохимической структуры трахеальной стенки, а также динамики живой массы у цыплят-бройлеров ( Gallus gallus domesticus ) кросса Ross 308, содержащихся в вентилируемых помещениях и при отсутствии циркуляции воздуха.
Методика. Работу выполняли в 2020-2021 годах на птицефабрике ООО «Челны-Бройлер» (Республика Татарстан), благополучной по эпизоотическому состоянию. Цыплят-бройлеров кросса Ross 308 выращивали до 39-суточного возраста в пяти закрытых производственных корпусах, которые различались по равномерности распределения воздушных потоков и циркуляции воздуха (пять групп по 35 гол. в каждой, отобраны методом пар-аналогов). Птицу содержали на глубокой подстилке. Кормление полнорационными комбикормами было семифазовым. На 3-ю, 4-ю и 5-ю нед оценивали живую массу индивидуальным взвешиванием.
Трахеи получали от всех 35 бройлеров (средняя живая масса 2500 г) в каждой из пяти групп (всего 175 образцов). Образцы фиксировали в 10 % формалине, затем промывали водой и заливали в парафин. Срезы тканей толщиной 5 мкм (по три среза каждого образца, всего 525 препаратов) готовили по стандартным методикам (17) и окрашивали гематоксилином и эозином для приготовления обзорных препаратов. Для гистохимических исследований кислых и нейтральных муцинов срезы окрашивали комбинированным методом выявления полисахаридов с применением реакции Шифф-йодной кислоты (ШИК-реакции) и альцианового синего (кислые гликозаминогликаны окрашиваются в синий цвет, ШИК-положительные гликопротеины — в фиолетовый) (17). Для более точного дифференцирования гликопротеинов от протеогликанов, содержащих в своем составе кислые гликозаминогликаны, срезы, полученные с тех же образцов, окрашивали при помощи только ШИК-реакции. Использовали наборы реактивов фирмы «ООО Лабико» (Россия) в соответствии с методикой, рекомендованной производителем.
Препараты просматривали под световым биологическим микроскопом Биолам М-3 (АО «ЛОМО», Россия) с разными кратностями увеличения (15½8, 15½20 и 15½40), фотографировали и описывали. Оценку толщины эпителия и подсчет ресничек трахеи проводили с помощью окуляр-микро-метра с последующим переводом относительных значений в абсолютные на объект-микрометре.
Во всех помещениях, где содержалась птица, воздухообмен обеспечивался приточно-вытяжной системой вентиляции, работающей по принципу отрицательного давления. Циркуляционные осевые вентиляторы SF-550-02 («AgroKurs», Россия) производительностью 8,5 тыс. м3/ч каждый (суммарная производительность 42,5 тыс. м3/ч) в залах ¹ 1 (I группа) и ¹ 5 (V группа) были установлены на одной высоте с газогенераторами, на расстоянии 10,8 м от выходных отверстий газогенераторов, под наклоном 5° вниз по направлению к птице. Электропитание циркуляционных вентиляторов осуществлялось совместно с газогенераторами, включение обогрева и циркуляции воздуха в помещениях происходило одновременно. В зале ¹ 1 движение воздушных потоков от циркуляционных вентиляторов было направлено к вытяжной вентиляции, в зале ¹ 5 — от вытяжной вентиляции. Циркуляционные вентиляторы запускали с 10-суточного возраста бройлеров. В помещениях, где содержались II, III и IV контрольные группы, циркуляционные вентиляторы отсутствовали. Контрольные группы различались между собой возрастом родительского стада птицы, от которой были отведены цыплята-бройлеры для выращивания: II группа — 28 нед, III группа — 47 нед, IV группа — 38 нед.
При проведении эксперимента соблюдали требования рекомендаций «Guide for the Care and Use of Agricultural Animals in Research and Teaching, 3d edition» (Federation of Animal Science Societies, 2010). Были предприняты все меры, чтобы свести к минимуму страдания птиц и уменьшить число особей, подвергнутых эвтаназии.
Статистическую обработку проводили методом вариационной статистики с использованием t -критерия Стьюдента в пакете Microsoft Excel 2010. Рассчитывали средние значения ( M) и стандартные ошибки средних (±SEM). Различия считали статистически значимыми при р < 0,01 для биологических значений, р < 0,05 — для зоотехнических значений.
Результаты. У цыплят кросса Ross 308 морфогенез трахеи заканчивается к 35-м сут постнатального онтогенеза (11), поэтому все изменения в гистологическом строении, наблюдаемые нами, не были возрастными. Образцы тканей у птицы из I и V групп отличались от образцов из II, III и IV групп меньшей толщиной слизистой оболочки и собственной пластинки.
1. Толщина слоев (мкм) слизистой оболочки трахеи у цыплят-бройлеров ( Gallus gallus domesticus ) кросса Ross 308 в зависимости от циркуляции воздуха в помещении ( M ±SEM, условия птичника, ООО «Челны-Бройлер», Республика Татарстан, 2020-2021 годы)
|
Стенка трахеи |
Группа ( n = 105) |
||||
|
I |
II |
III |
IV |
V |
|
|
Слизистая оболочка Собственная пластинка |
147,2±3,3 |
267,5±4,1* |
404,7±9,4* |
298,1±10,5* |
161,2±2,9* |
|
слизистой |
129,1±3,1 |
253,9±4,2* |
395,7±9,4* |
285,3±10,4* |
144,8±2,9* |
|
Эпителиальный слой |
16,1±0,4 |
16,2±0,3 |
13,2±0,3* |
15,1±0,3* |
18,4±0,4* |
|
Реснички |
4,3±0,1 |
2,6±0,1* |
1,8±0,1* |
1,4±0,1* |
4,3±0,1* |
Примечание. Описание групп см. в разделе «Методика».
* Различия с I опытной группой статистическим значимы при р < 0,01.
-
У птицы из I опытной группы толщина слизистой оболочки и входящей в ее состав собственной пластинки оказалась минимальной (соответственно 147,2±3,3 и 129,1±3,1 мкм), в III контрольной — максимальной (404,7±9,4 мкм и 395,7±9,4 мкм) (р < 0,01). Толщина эпителиального слоя трахеи была достоверно выше в I и V опытных группах в среднем на 14 % в сравнении со II, III и IV контрольными группами (р < 0,01). Отсутствие циркуляции воздуха в помещениях привело к статистически значимому снижению высоты ресничек во II, III и IV группах соответственно на 39,5;
58,1 и 67,5 % по отношению к I и V опытным группам (p ≤ 0,01) (табл. 1).
С увеличением толщины эпителия и высоты ресничек возрастает способность удерживания в слизистой частиц вдыхаемого воздуха. Реснички как органеллы движения играют решающую роль в защите дыхательных путей от экзогенных частиц и участвуют в реакциях неспецифического иммунитета (1). Статистически значимое увеличение размера ресничек в группах с циркуляцией воздуха указывало на лучшие условия для выращивания птицы. Тенденция увеличения размера ресничек в зависимости от толщины эпителия отсутствовала.
Микрофотографии гистоструктуры трахеальной стенки птиц приведены для групп I, V и III, исходя из минимальных и максимальных значений показателей.
Гистологические исследования подтвердили существующие данные о том, что стенка трахеи птиц состоит из слизистой, подслизистой, фиброзно-хрящевой оболочек и адвентиции (9, 16, 34). Слизистая, в свою очередь, включала многорядный мерцательный эпителий и собственную пластинку слизистой оболочки, представленную рыхлой соединительной тканью. Кроме волокнистого компонента и основного аморфного вещества, в ней встречались клеточные формы, характерные для этого вида ткани, и клетки крови лейкоцитарного ряда. Подслизистая оболочка состояла из рыхлой соединительной ткани с преобладанием волокнистого компонента, что также согласуется с данными, полученными ранее (9, 11-13). Фибрознохрящевая оболочка была представлена замкнутыми кольцами гиалинового хряща и плотной соединительной тканью между ними. Из-за того, что анатомически кольца трахеи перекрывали друг друга, на препарате можно было увидеть одновременно срез двух соседних колец. Адвентиция была представлена рыхлой соединительной тканью (рис. 1).
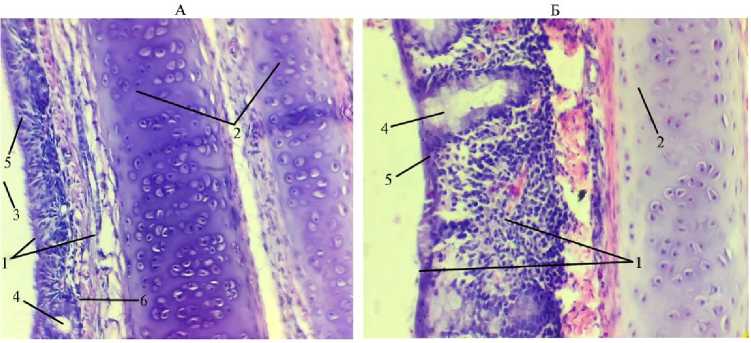
Рис. 1. Гистологическая структура стенки трахеи у цыплят-бройлеров ( Gallus gallus domesticus ) кросса Ross 308 из I группы, которую содержали в вентилируемом помещении (А) , и III группы, находившейся в помещении без циркуляции воздуха (Б) : 1 — слизистая оболочка, 2 — фибрознохрящевая оболочка, 3 — просвет трахеи, 4 — слизистые железы, 5 —эпителий, 6 — собственная пластинка слизистой (условия птичника, ООО «Челны-Бройлер», Республика Татарстан, 20202021 годы). Окрашивание гематоксилином и эозином, увеличение 15½20, микроскоп Биолам М-3 (АО «ЛОМО», Россия).
Комплексное проведение ШИК-реакции и окрашивание срезов аль-циановым синим позволило проследить распределение кислых гликозаминогликанов и ШИК-положительных гликопротеинов в структурах стенки органа (рис. 2).
Дополнительное проведение ШИК-реакции подтвердило локализа- цию нейтральных мукополисахаридов, поскольку в некоторых случаях они перекрывались более ярким окрашиванием альциановым синим. Так, кислые мукополисахариды, содержащие сульфатированные гликозаминогликаны и окрашенные на препарате в ярко-синий цвет, в основном локализовались в хряще и входили в состав слизи, выделяемой как одноклеточными, так и многоклеточными железами. Дополнительная идентификация ШИК-реак-цией выявила, что нейтральные гликопротеины, окрашиваемые в фиолетовый цвет, обнаруживаются в секрете желез в максимальной концентрации и значительно слабее окрашены в области хряща. В фиброзно-хрящевой оболочке они в основном локализовались в области надхрящницы и в зоне молодого хряща. Наблюдаемое распределение химических групп указывает на то, что хондромукоид был в большей степени представлен сульфатированными (кислыми) мукополисахаридами, чем нейтральными. Слизь, выделяемая железами, состояла в равной мере из кислых и нейтральных мукополисахаридов. Распределение этих групп веществ по другим структурам стенки органа показало, что как кислые гликозаминогликаны, так и нейтральные гликопротеины в умеренных количествах обнаруживались в межклеточном веществе рыхлой соединительной ткани.
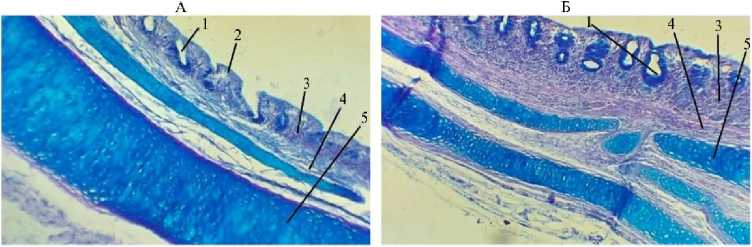
Рис. 2. Гистологическая структура стенки трахеи у цыплят-бройлеров ( Gallus gallus domesticus ) кросса Ross 308 из V группы, которую содержали в вентилируемом помещении (А) , и III группы, находившейся в помещении без циркуляции воздуха (Б) : 1 — железы, 2 — эпителий, 3 — собственная пластинка слизистой, 4 — подслизистая оболочка, 5 — фиброзно-хрящевая оболочка (условия птичника, ООО «Челны-Бройлер», Республика Татарстан, 2020-2021 годы). Окрашивание альциановым синим + ШИК-реакция, увеличение 15½8, микроскоп Биолам М-3 (АО «ЛОМО», Россия).
В трахее у птиц из V группы патологические изменения в реснитчатом эпителии отсутствовали. В собственной пластинке слизистой оболочки были видны ядра фиброцитов, волокнистый компонент и основной аморфный компонент. В однослойном многорядном мерцательном эпителии мы обнаружили высокие цилиндрические клетки с ресничками на апикальном полюсе и камбиальные клетки, не доходящие до края пласта и расположенные около базальной мембраны. Апикальный полюс клеток не был разрушен (см. рис. 1). Местами в пласте эпителия присутствовали бокаловидные клетки, выделяющие слизь. Кроме бокаловидных клеток в пласте эпителия мы обнаружили простые эндоэпителиальные железы (рис. 3), что отмечалось (9, 12, 13). Их содержимое имело одновременно ШИК-положительную реакцию и окрашивалось альциановым синим. Бокаловидные клетки встречались как поодиночке, так и небольшими группами в несколько клеток, лежащих рядом (рис. 4), что подтверждают некоторые исследования (4, 12).
Кроме того, в собственной пластинке располагались простые трахеальные слизистые железы, имеющие такую же окраску. Зона ресничек более активно окрашивалась альциановым синим, в то время как слизь в железах одинаково хорошо окрашивалась и тем, и другим методами. Вероятно, слизь, находящаяся около ресничек и составляющая более жидкую фракцию (4), содержала больше кислых мукополисахаридов. В соединительной ткани слизистой и подслизистой оболочки преобладали кислые мукополисахариды (см. рис. 2). При изучении гистоструктуры трахеи птицы из I группы результаты были схожие с полученными для V группы.
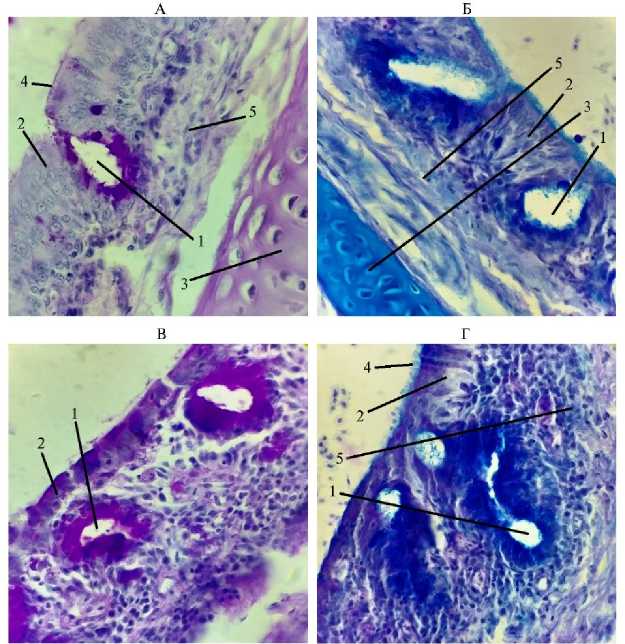
Рис. 3. Гистологическая структура стенки трахеи у цыплят-бройлеров ( Gallus gallus domesticus ) кросса Ross 308 из V группы, которую содержали в вентилируемом помещении (А, Б) , и III группы, находившейся в помещении без циркуляции воздуха (В, Г) : 1 — железы, 2 — эпителий, 3 — хрящ, 4 — реснички, 5 — собственная пластинка слизистой (условия птичника, ООО «Челны-Бройлер», Республика Татарстан, 2020-2021 годы). Слева — ШИК-реакция, справа — окрашивание альциановым синим + ШИК-реакция, увеличение 15½40, микроскоп Биолам М-3 (АО «ЛОМО», Россия).
В трахее у птиц из III группы отмечали заметное уменьшение толщины эпителия (см. рис. 1). Местами эпителий подвергается метаплазии, в остальных участках его высота была достоверно ниже (p ≤ 0,01) в сравнении с I и V группами, кое-где реснички практически отсутствовали. В просвете трахеи иногда встречались группы клеток или отдельно лежащие клетки, среди которых присутствовали эритроциты, макрофаги и лейкоциты, мигрировавшие из собственной пластинки слизистой на поверхность эпителия. Это согласуется с данными о застойных явлениях в дыхательной системе птиц в связи с недостаточной циркуляцией воздуха (4). Частичный недостаток воздухообмена приводит к сверхнормативной концентрации вредных газов и повышенной запыленности, что может вызывать воспалительный процесс в трахеальной стенке. Подобный эффект был получен в эксперименте по выращиванию бройлеров с 42-суточного возраста в птичнике с концентрацией 100 ppm NH 3 /л воздуха в течение 1 нед (32) и в опыте по содержанию индеек в птичнике с концентрацией 10 ppm NH 3 /л воздуха
-
(33). В этих работах не обнаружено повреждений реснитчатого эпителия и бокаловидных клеток в трахее птиц, однако трахея индеек имела угнетенное состояние мукоцилиарного аппарата, характеризующееся спутанными ресничками и участками децилирования (32, 33).
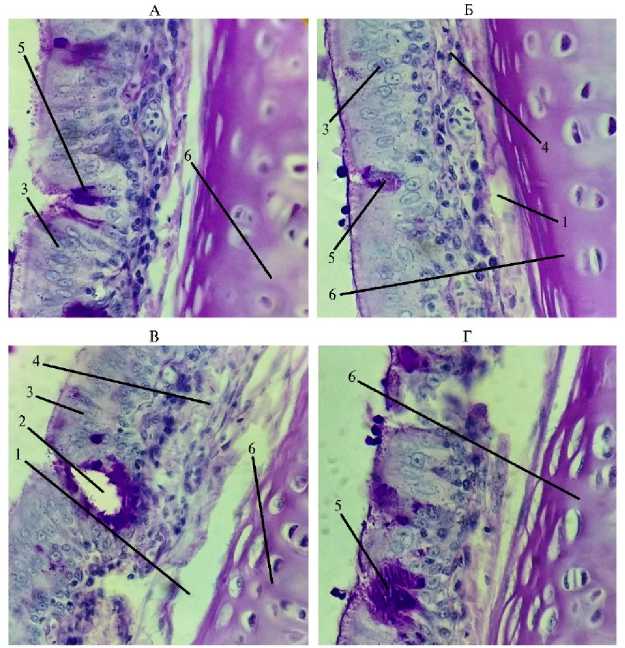
Рис. 4. Эндоэпителиальные железы и бокаловидные клетки в трахее у цыплят-бройлеров ( Gallus gallus domesticus ) кросса Ross 308 из V группы, которую содержали в вентилируемом помещении: 1 — подслизистая оболочка, 2 — эндоэпителиальные железы, 3 — эпителий, 4 — собственная пластинка слизистой, 5 — бокаловидные клетки, 6 — хрящ (условия птичника, ООО «Челны-Бройлер», Республика Татарстан, 2020-2021 годы). ШИК-реакция, увеличение 15½40, микроскоп Биолам М-3 (АО «ЛОМО», Россия) .
Полученные в нашем опыте данные частично сопоставимы с описанием патологий, которые встречаются при инфицировании цыплят. Так, перестройка реснитчатого эпителия, катаральное воспаление и отек эпителия слизистой за счет инфильтрации лимфоцитами наблюдаются при инфицировании изолятом вируса гриппа птиц и ларинготрахеите (28, 29). Отмечена децилиация, десквамация и альтерация клеток реснитчатого эпителия трахеи при заражении вирусом инфекционного бронхита кур (26, 27, 35).
При выявлении мукополисахаридов в III контрольной группе мы наблюдали активную ШИК-реакцию и окрашивание альциановым синим пласта эпителия, чего не было в I и V опытных группах. В собственной пластинке слизистой оболочки трахеи птиц из III группы преобладал клеточный компонент, волокна были малочисленны. Слизистая оказалась инфильтрирована лейкоцитами, среди которых преобладали лимфоциты. В рыхлой соединительной ткани слизистой и подслизистой у птиц из III (контрольной) группы выявляли больше гликопротеинов, чем в I и V опытных группах (см. рис. 2). Многорядность эпителия была слабо выражена, местами наблюдалась децилиация и метаплазия. У птиц из II и IV групп гистоструктура трахеи была схожа с наблюдаемой в образцах от цыплят из III группы.
К отеку слизистой может приводить повышенная концентрация углекислого газа во вдыхаемом воздухе, что подтверждается ранними исследованиями (2). Слои слизистой оболочки трахеи птиц, инфицированных штаммами вируса инфекционного бронхита кур разных серотипов, характеризуются умеренным отеком собственной пластинки слизистой оболочки и отслоением эпителиальных клеток трахеи, увеличением количества бокаловидных клеток (26, 27).
Считается, что бокаловидные клетки и трахеальные железы выделяют вязкую слизь под влиянием местных раздражающих факторов и не иннервируются адренергическими и холинергическими рецепторами (35). Кроме того, бокаловидные клетки имеют короткий период жизни, всего 24 сут (1), поэтому могут быть чувствительными к внешним воздействиям. Следовательно, различные местные воздействия на слизистую приводят к количественным и качественным изменениям в этих железах.
Мы показали, что в начальном участке трахеи у птиц из I и V опытных групп присутствовали и трахеальные железы, и бокаловидные клетки, но наблюдался сдвиг баланса в сторону желез. Эти результаты согласуются с ранее полученными данными (9, 12, 13) о том, что железы преобладают в начальных участках, а бокаловидные клетки — в каудальной части трахеи. При этом бокаловидные клетки лежали как поодиночке, так и группами. Гистохимические методы окраски выявили клетки, заполненные слизистым секретом полностью, изредка встречались бокаловидные клетки с незначительным окрашиванием, предположительно генерализованно выделившие секрет гранул (см. рис. 4). Подобным образом описаны бокаловидные клетки у человека (1). Некоторые авторы отмечают увеличение количества бокаловидных клеток при воздействии неблагоприятных факторов среды на эпителий трахеи (4, 7, 26, 27).
В наших исследованиях во II, III и IV контрольных группах при недостаточной вентиляции мы обнаружили уменьшение количества бокаловидных клеток, что, по-видимому, было связано со структурными изменениями в эпителии, однако наблюдалось увеличение количества трахеальных желез. Это подтверждалось повышенным количеством ШИК-положительных мукополисахаридов в рыхлой соединительной ткани вокруг желез. Следует отметить ярко выраженное увеличение содержания кислых и нейтральных мукополисахаридов в контрольных группах по сравнению с опытными. Такие гистохимические и морфологические изменения в стенке трахеи у птицы, которую содержали в условиях недостаточной вентиляции, видимо, служат компенсаторным механизмом восстановления мукоцилиарного транспорта.
2. Динамика живой массы (г) у цыплят-бройлеров ( Gallus gallus domesticus ) кросса Ross 308 в зависимости от циркуляции воздуха в помещении ( M ±SEM; условия птичника, ООО «Челны-Бройлер», Республика Татарстан, 20202021 годы)
|
Группа ( n = 35) 1 |
3-я нед 1 |
4-я нед |
5-я нед |
|
I опытная |
1047,6±10,6 |
1712,2±12,4 |
2366,9±19,2 |
|
II контрольная |
1080,9±10,2 |
1602,0±19,2* |
2214,1±29,1* |
|
III контрольная |
1149,3±11,7 |
1688,2±15,5 |
2290,7±18,5* |
|
IV контрольная |
1110,4±10,3 |
1591,1±10,4* |
2198,2±21,0* |
|
V опытная |
1159,6±11,2 |
1690,1±16,5 |
2317,6±20,0 |
Примечание. Описание групп см. в разделе «Методика».
*Различия с I опытной группой статистическим значимы при p ≤ 0,05.
Мы изучили изменение прироста живой массы у цыплят-бройлеров на фоне разной циркуляции воздуха (табл. 2). Прирост живой массы птицы в 5-недельном возрасте в I опытной группе был выше в сравнении со II, III и IV контрольными группами соответственно на 6,5; 3,2 и 7,1 % (p ≤ 0,05). В 4-недельном возрасте в I опытной группе различия по отношению ко II и IV контрольным группам составили 6,4 и 7,1 % (p ≤ 0,05), в то время как до 4-недельного возраста статистически значимые различия отсутствовали.
Таким образом, мы подтвердили зависимость состояния гистоструктуры и гистохимического статуса трахеальной стенки у цыплят-бройлеров кросса Ross 308 от циркуляции воздуха в производственных помещениях закрытого типа. Недостаточность циркуляции воздушных потоков в залах выращивания птиц вызывает деструктивные изменения слизистой оболочки трахеи, которые выражаются в разрастании собственной пластинки слизистой, отеке, уменьшении высоты эпителия, разрушении ресничек, снижении продуктивности. Это приводит к метаплазии эпителия и нарушению мукоцилиарного транспорта. С обеспечением циркуляции воздушных потоков в закрытых помещениях толщина слизистой оболочки уменьшается, а высота эпителиального слоя и ресничек трахеи увеличивается. О благоприятном влиянии циркуляции воздуха в закрытых помещениях для выращивания птиц достоверно свидетельствует истончение слизистой оболочки трахеи в 2,6 раза (на 61,9 %), уменьшение толщины собственной пластинки в 2,9 раза (на 65,4 %), увеличение толщины эпителиального слоя в 1,3 раза (на 23,3 %) и высоты ресничек в 3,1 раза (на 67,5 %), а также увеличение прироста живой массы птицы (до 7,1 %). Полученные гистологические препараты позволяют предположить наличие у птиц в пласте эпителия трахеи простых многоклеточных эндоэпителиальных желез. Описанные изменения гистоструктуры трахеальной стенки могут служить индикаторами неблагоприятных условий содержания птицы. Выращивание мясных кроссов непродолжительно, поэтому для изучения последствий более длительного воздействия высокой загазованности и запыленности воздуха следует использовать другие технологические группы птицы (например, родительское стадо).
Список литературы Генетическая дифференциация индеек разных пород по микросателлитным маркерам
- Столповский Ю.А., Захаров-Гезехус И.А.Проблема сохранения генофондов доместицированных животных. Вавиловский журнал генетики и селекции, 2017, 21(4): 477-486 (doi: 10.18699/VJ17.266).
- Jarne P., Lagoda P.J.L. Microsatellites, from molecules to populations and back. Trends in Ecology & Evolution, 1996, 11(10): 424-429 (doi: 10.1016/0169-5347(96)10049-5).
- Gholizadeh M., Mianji G.R. Use of microsatellite markers in poultry research. International Journal of Poultry Science, 2007, 6(2): 145-153 (doi: 10.3923/ijps.2007.145.153).
- Putman A.I., Carbone I. Challenges in analysis and interpretation of microsatellite data for population genetic studies. Ecology Evolution, 2014, 4(22): 4399-4428 (doi: 10.1002/ece3.1305).
- Tan C., Bian C., Yang D., Li N., Wu Z.-F., Hu X.-X. Application of genomic selection in farm animal breeding. Yi Chuan, 2017, 39(11): 1033-1045 (doi: 10.16288/j.yczz.17-286).
- Weigend S., Romanov M.N. The world watch list for domestic animal diversity in the context of conservation and utilisation of poultry biodiversity. World's Poultry Science Journal, 2002, 58(4): 411-430 (doi: 10.1079/WPS20020031).
- Groeneveld L.F., Lenstra J.A., Eding H., Toro M. A., Scherf B., Pilling D., Negrini R., Finlay E.K., Jianlin H., Groeneveld E., Weigend S., the GLOBALDIV Consortium. Genetic diversity in farm animals — a review. Animal Genetics, 2010, 41(s1): 6-31 (doi: 10.1111/j.1365-2052.2010.02038.x).
- Soller M., Weigend S., Romanov M.N., Dekkers J.C.M., Lamont S.J. Strategies to assess structural variation in the chicken genome and its associations with biodiversity and biological performance. Poultry Science, 2006, 85(12): 2061-2078 (doi: 10.1093/ps/85.12.2061).
- Wilkinson S., Wiener P., Teverson D., C.S.Haley, Hjcking P.M. Characterization of the genetic diversity, structure and admixture of British chicken breeds. Animal Genetics, 2012, 43(5): 552-563 (doi: 10.1111/j.1365-2052.2011.02296.x).
- Reed K.M., Mendoza K.M., Beattie C.W. Comparative analysis of microsatellite loci in chicken and turkey. Genome, 2000, 43(5): 796-802.
- Colombo E., Strillacci G., Cozzi M.C., Madeddu M., Mangiagalli M.G., Mosca F., Zaniboni L., Bagnato A., Cerolini S. Feasibility study on the FAO chicken microsatellite panel to assess genetic variability in the turkey (Meleagris gallopavo). Italian Journal of Animal Science, 2014, 13(4): 887-890 (doi: 10.4081/ijas.2014.3334).
- Новгородова И.П., Гладырь Е.А., Фисинин В.И., Зиновьева Н.А. Идентификация породной принадлежности кур на основе микросателлитного анализа. Достижения науки и техники АПК,2015, 29(11): 88-90.
- Новгородова И.П., Волкова В.В., Гладырь Е.А., Селионова М.И., Растоваров Е.И., Фисинин В.И., Зиновьева Н.А. Изучение информативности микросателлитов кур G. Gallusдля характеристики аллелофонда индеек M. gallopavo. Достижения науки и техники АПК,2011, 10: 66-67.
- Reed K.M., Chaves L.D., Garbe J.R., Da Y., Harry D.E. Allelic variation and genetic linkage of avian microsatellites in a new turkey population for genetic mapping. Cytogenetic and Genome Research, 2003, 102(1-4): 331-339 (doi: 10.1159/000075771).
- Reed K.M., Chaves L.D., Knutson T.P., Krueth S.B., Ashwell C.M., Burt D.W. Integration of microsatellite — based genetic maps for the turkey (Meleagris gallopavo). Genome, 2006, 49(10): 1308-1318 (doi: 10.1139/g06-084).
- Smith E.J., Geng T., Long E., Pierson F.W., Sponenberg D.P., Larson C., Gogal R. Molecular analysis of the relatedness of five domesticated turkey strains. Biochemical Genetics, 2005, 43(1-2): 35-47 (doi: 10.1007/s10528-005-1065-5).
- Фисинин В.И., Селионова М.И., Шинкаренко Л.А., Щербатова Н.Г., Кононова Л.В. Исследование микросателлитных локусов в породах индеек российской селекции. Сельскохозяйственная биология, 2017, 52(4): 739-748 (doi: 10.15389/agrobiology.2017.4.739rus).
- Reed K.M., Chaves L.D., Rowe J.A. Twelve new turkey microsatellite loci. Poultry Science, 2002, 81(12): 1789-1791 (doi: 10.1093/ps/81.12.1789).
- Reed K.M., Roberts M.C., Murtaugh J., Beattie C.W., Alexander L.J. Eight new dinucleotide loci in turkey (Meleagris gallopavo). Animal Genetics, 2000, 31(2): 140-157 (doi: 10.1046/j.1365-2052.2000.00571.x).
- Peakall R., Smouse P.E. GenAIEx 6.5: genetic analysis in Excel. Population genetic software for teaching and research — an update. Bioinformatics, 2012, 28(19): 2537-2539 (doi: 10.1093/bioinformatics/bts460).
- Weir B.S., Cockerham C.C. Estimating F-statistics for the analysis of population structure. Evolution, 1984, 38(6): 1358-1370 (doi: 10.2307/2408641).
- Nei M. Genetic distance between populations. American Naturalist, 1972, 106: 283-392.
- Falush D., Stephens M., Pritchard J.K. Inference of population structure using multilocus genotype data: linked loci and correlated allele frequencies. Genetics, 2003, 164(4): 1567-1587.
- Latch E.K., Smith E.J., Rhodes O.E. Isolation and characterization of microsatellite loci in wild and domestic turkeys (Meleagris gallopavo). Molecular Ecology Note, 2002, 2(2): 176-178 (doi: 10.1046/j.1471-8286.2002.00183.x).
- Kusza S., Mihók S., Czeglédi L., Javor A., Arnyasi M. Testing the breeding strategy of Hungarian Bronze turkey strains for maintaining genetic diversity with microsatellites. Arch. Anim. Breed., 2011, 54(4): 419-429 (doi: 10.5194/aab-54-419-2011).
- Шинкаренко Л.А., Терлецкий В.П., Тыщенко В.И. Генетические особенности пород индеек биоресурсной коллекции селекционно-генетического центра «СКЗОСП». Птицеводство, 2020, 9: 17-21 (doi: 10.33845/0033-3239-2020-69-9-17-21).
- Kamara D., Gyenai K.B., Geng T., Hammade H. Microsatellite marker-based genetic analysis of relatedness between commercial and heritage turkeys (Meleagris gallopavo). Poultry Science, 2007, 86(1): 46-49 (doi: 10.1093/ps/86.1.46).
- Knutson T.P., Chaves L.D., Hall M.K., Reed K.M. One hundred fifty-four genetic markers for the turkey (Meleagris gallopavo). Genome, 2004, 47(6): 1015-1028 (doi: 10.1139/g04-076).
- Mock K.E., Theimer T.C., Rhodes O.E., Greenberg D.L., Keim P. Genetic variation across the historical range of the wild turkey (Meleagris gallopavo). Molecular Ecology, 2002, 11(4): 643-657 (doi: 10.1046/j.1365-294X.2002.01467.x).
- Aslam M.L., Bastiaansen J.W.M., Elferink M.G, Megens H.J., Crooijmans R.P.M.A., Blomberg L.A., Fleischer R.C., Tassell C.P., Sonstegard T.S., Schroeder C.G., Groenen M.A.M, Long J.A. Whole genome SNP discovery and analysis of genetic diversity in Turkey (Meleagris gallopavo). BMC Genomics, 2012, 13: 391-404 (doi: 10.1186/1471-2164-13-391 ).
- Flicek P., Amode M.R., Barrell D., Beal K., Brent S., Carvalho-Silva D., Clapham P., Coates G., Fairley S., Fitzgerald S., Gil L., Gordon L., Hendrix M., Hourlier T., Johnson N., Kähäri A. K., Keefe D., Keenan S., Kinsella R., Komorowska M., Koscielny G., Kulesha E., Larsson P., Longden I., McLaren W., Muffato M., Overduin M.M.B., Pignatelli M., Pritchard B., Riat H.S., Ritchie G., Ruffier M., Schuster M.R.B., Sobral D., Tang A., Taylor T., Trevanion S., Vandrovcova J., White S.J., Wilson M., Wilder S.P., Aken B.L., Birney E., Cunningham F., Dunham I., Durbin R., Fernandez-Suarez X., Harrow J., Herrero J., Hubbard T., Parker A., Proctor G., Spudich G., Vogel J., Yates A., Zadissa A., Searle S. Ensembl 2012. Nucleic Acids Research. 2012, 40(D1): D84-D90 (doi: 10.1093/nar/gkr991).
- Dalloul R. A., Long J.A., Zimin A.V., Aslam L., Beal K., Blomberg L. A., Bouffard P., Burt D.W., Crasta O., Crooijmans R.P.M.A., Cooper K., Coulombe R.A., De S., Delany M.E., Dodgson J.B., Dong J.J., Evans C., Frederickson K.M., Flicek P., Florea L., Folkerts O., Groenen M.A.M., Harkins T., Herrero J., Hoffmann S., Megens H.-J., Jiang A., Jong P., Kaiser P., Kim H., Kim K-W., Kim S., Langenberger D., Lee M-K., Lee T., Mane S., Marcais G., Marz M., McElroy A., Modise T., Nefedov M., Notredame C., Paton I.R., Payne W.S., Pertea G., Prickett D., Puiu D., Qioa D., Raineri E., Ruffier M., Salzberg S.L., Schatz M.C., Scheuring C., Schmidt C.J., Schroeder S., Searle S.M.J., Smith E.J., Smith J., Sonstegard T.S., Stadler P.F., Tafer H., Tu Z.J., Tassell C.P., Vilella A.J., Williams K.P., Yorke J.A., Zhang L., Zhang H.-B., Zhang X., Zhang Y., Reed K.M. Multi-platform next-generation sequencing of the domestic turkey (Meleagris gallopavo): Genome Assembly and Analysis. PLoS Biology, 8(9): e1000475 (doi: 10.1371/journal.pbio.1000475).


