Идентификация SNPS и генов-кандидатов, ассоциированных с отложением абдоминального жира у перепелов Coturnix japonica
Автор: Волкова Н.А., Герман Н.Ю., Ларионова П.В., Ветох А.Н., Романов М.Н., Зиновьева Н.А.
Журнал: Сельскохозяйственная биология @agrobiology
Рубрика: Генетика и геномика
Статья в выпуске: 6 т.58, 2023 года.
Бесплатный доступ
Интенсивность отложения жира, в том числе абдоминального, - один из важных показателей, характеризующих как мясную продуктивность и качество продукции, так и общее здоровье сельскохозяйственной птицы. Этот признак положительно коррелирует с быстрым ростом птицы и в значительной степени зависит не только от условий кормления и содержания, но и от генетических факторов. В основном данные о генетических механизмах, обусловливающих жировой обмен и интенсивность отложения жира, получены на курах, идентифицированы SNPs и гены-кандидаты, детерминирующие отложение как внутримышечного, так и брюшного (абдоминального) жира. Число подобных исследований на перепелах относительно невелико. К настоящему времени в специальной литературе недостаточно информации о локусах количественных признаков (QTL), достоверно связанных с показателями жирового обмена у перепелов. В представленной работе впервые сообщается о выявленных SNPs, с высокой достоверностью (p 2 модельной ресурсной популяции. В области выявленных SNPs установлены гены-кандидаты, достоверно связанные с этим признаком. Целью работы были поиск SNPs и идентификация генов-кандидатов, связанных с отложением абдоминального жира у перепелов. Исследования проводили на самцах F2 модельной ресурсной популяции ( n = 146), полученной при скрещивании двух контрастных по скорости роста и мясным качествам пород - японских (медленный рост) и техасских (быстрый рост) перепелов. Особей F2 генотипировали методом GBS (genotyping-by-sequencing, генотипирование посредством секвенирования). Для выявления ассоциаций между данными полногеномного генотипирования и количеством абдоминального жира использовали программное обеспечение PLINK 1.9 с принятыми ограничениями (geno 0,1; mind 0,1; maf 0,05). В качестве порогового критерия достоверности установили p 2 ресурсная популяция перепелов характеризовалась высокой вариабельностью по содержанию абдоминального жира в туше. В возрасте 56 сут этот показатель варьировал от 0,01 до 10,46 г и составил в среднем 2,41±0,16 г. На основании проведенного GWAS-анализа (genome-wide association study) идентифицировали 29 SNPs и 11 генов-кандидатов, находящихся в областях расположения этих SNPs, которые ассоциированы с отложением абдоминального жира у перепелов. Обнаруженные SNPs локализуются на хромосомах 1, 2, 7, 8, 17, 19, 21, 24 и 28. Установленные гены-кандидаты ( CNTN5, GNAL, PDE1A, RBMS1, PTPRF, SH3GLB2, SLC27A4, TRIM62, IGSF9B, USHBP1, NR2F6 ) идентифицированы на хромосомах CJA1 (1), CJA2 (1), CJA7 (2), CJA8 (1), CJA17 (2), CJA21 (1), CJA24 (1) и CJA28 (2). Детектированные SNP и гены-кандидаты могут послужить генетическими маркерами в программах селекции на улучшение мясных качеств перепелов и снижение содержания жира в тушках.
Coturnix japonica, перепел, qtl, snp, genotyping-by-sequencing, gbs, genome-wide association study, gwas, гены-кандидаты, абдоминальный жир
Короткий адрес: https://sciup.org/142240679
IDR: 142240679 | УДК: 636.5:577.2 | DOI: 10.15389/agrobiology.2023.6.1079rus
Текст научной статьи Идентификация SNPS и генов-кандидатов, ассоциированных с отложением абдоминального жира у перепелов Coturnix japonica
Продукция птицеводства занимает прочные позиции в общей структуре продуктов питания животного происхождения (1, 2). Большим спросом пользуются как яйца, так и мясо сельскохозяйственной птицы (3, 4). В отличие от других видов сельскохозяйственных животных, у птицы мясо представляет собой диетический продукт с высокой пищевой ценностью и хорошими вкусовыми качествами (5, 6 ) . В составе мяса птицы, как правило, содержится небольшое количество жира и значительная доля белков (3, 6).
Основу устойчиво развивающегося и конкурентоспособного мясного птицеводства составляют высокопродуктивные породы и кроссы. Поэтому требуется постоянная селекционная работа по поиску и идентификации ценных генотипов. Для этого используются современные методы, позволяющие выявлять молекулярно-генетические механизмы формирования
∗ Работа выполнена при финансовой поддержке Российского научного фонда, грант ¹ 21-16-00086
и проявления селекционно значимых признаков. Одна из задач геномной селекции на улучшение качества птицеводческой продукции и повышение рентабельности отрасли — поиск SNPs и идентификация генов-кандидатов, детерминирующих селекционно значимые признаки. За последние годы сформированы значительные базы данных SNPs и генов-кандидатов, ассоциированных с показателями мясной продуктивности сельскохозяйственной птицы (7, 8).
К важным показателям мясных качеств птицы относится интенсивность отложения жира (9, 10). Различают внутримышечный и внутренний (брюшной, абдоминальный) жир. Внутримышечный жир определяет питательность мяса, его вкусовые качества и текстуру (9). Абдоминальный жир откладывается у птицы в брюшной полости и служит источником энергии. Его содержание в тушке может достигать 3-4 % ее массы (11). Избыточное отложение абдоминального жира негативно влияет на здоровье птицы (12, 13) и качество тушек (10, 11). В ряде исследований рассматривается использование абдоминального жира для улучшения текстуры и вкусовых качеств, для повышения пищевой ценности и питательности продукции, производимой из мяса птицы (14, 15). На жировой обмен и накопление жировых отложений влияют ряд факторов: условия содержания (10), кормления (11, 16, 17), возраст (18, 19), пол (19), генетическая предрасположенность (2022). У самок жировые отложения больше, чем у самцов (19). Интенсивность накопления жира положительно коррелирует с быстрым ростом птицы, что затрудняет проведение отбора птицы одновременно по скорости роста и снижению количества жира в тушках (20).
Полногеномные исследования ассоциаций (genome-wide association study, GWAS) однонуклеотидных полиморфизмов (single nucleotide polymorphism, SNP) с экономически значимыми признаками позволяют эффективно идентифицировать связанные с их проявлением гены-кандидаты (23). У кур по сравнению с другими видами сельскохозяйственной птицы генетические механизмы, связанные с жировым обменом и интенсивностью отложения жира, изучены более подробно (24, 25), идентифицированы SNPs и гены-кандидаты, детерминирующие отложение как внутримышечного, так и брюшного (абдоминального) жира (26-28). В отношении перепелов такие данные ограничены (29). К настоящему времени недостаточно информации о локусах количественных признаков (QTL), достоверно связанных с показателями жирового обмена у перепелов.
В настоящем сообщении в F 2 модельной ресурсной популяции перепелов идентифицированы новые SNPs и гены-кандидаты, с высокой достоверностью (p < 0,00001) ассоциированные с интенсивностью отложения жира у перепелов. Детектированные SNPs могут быть рассмотрены как генетические маркеры в селекции на улучшение мясных качеств перепелов и снижение содержания жира в тушках.
Цель настоящей работы — поиск SNPs и идентификация генов-кандидатов, ассоциированных с отложением абдоминального жира у перепелов.
Методика. Исследования проводили на самцах F 2 модельной ресурсной популяции перепелов ( n = 146; виварий ФГБНУ ФИЦ ВИЖ им. академика Л.К. Эрнста, 2022-2023 годы), полученной при скрещивании двух контрастных по скорости роста и мясным качествам пород — японских (медленный рост) и техасских (быстрый рост) перепелов. На первом этапе сформировали 4 семьи, каждая из которых состояла из одного самца и пяти самок исходных пород. От каждой семьи получили по 20-30 особей F 1 , которых использовали для получения F 2 ресурсной популяции. С этой целью подобрали 12 семей (F 1 _1-F 1 _12), состоящих из одного самца и трех самок 1080
F 1 , не являющихся близкими родственниками. От этих особей F 1 получили и отобрали для исследования самцов F 2 (группы F 2 _1-F 2 _12).
После экспериментального убоя в возрасте 8 нед тушку разделывали, отбирали абдоминальный жир из брюшной области и определяли массу тушки и абдоминального жира взвешиванием. Для полногеномных исследований ассоциаций использовали показатели абсолютного и относительного содержания абдоминального жира в тушках. Относительное содержание абдоминального жира рассчитывали как процентное отношение массы абдоминального жира к общей массе тушки.
Для выделения ДНК использовали пульпу пера. ДНК экстрагировали с помощью набора Syntol для выделения ДНК из ткани животных (ООО «НПФ Синтол», Москва). Концентрацию растворов ДНК определяли на флуориметре Qubit® 3.0 («Thermo Fisher Scientific», США). Для проверки чистоты извлеченной ДНК определяли соотношение OD260/280 (NanoDrop-2000 («Thermo Fisher Scientific», США). Генотипирование перепелов проводили методом GBS (genotyping-by-sequencing) согласно описанному нами ранее протоколу (30). В качестве референсного использовали геном Japanese quail (Coturnix_japonica_2.0) . Удаление адаптеров и демультиплексирование файла fastq проводили в программе Сutadapt . Контроль качества fastq файлов осуществляли в программе FastQC (31). Для выравнивания относительно референсного генома использовали пакет bowtie2 (32). С использованием программного обеспечения R был получен файл допустимого для дальнейшего анализа формата (33). Качество детектирования SNPs контролировали в программе PLINK 1.9 . К полученным генотипам перепелов применили фильтр по параметру эффективности генотипирования (mind 0,1; maf 0,05), SNPs, генотипированные менее чем в 90 % образцов (geno 0,1), исключили из анализа.
Для выявления ассоциаций SNPs с количеством абдоминального жира у перепелов применяли регрессионный анализ в PLINK 1.9. Достоверность влияния SNP и определение значимых регионов в геноме перепелов оценивали с использованием теста для проверки нулевых гипотез при пороговом значении p < 0,00001. Данные визуализировали в пакете qqman с помощью языка программирования R (33). Поиск генов-кандидатов, расположенных в области идентифицированных SNPs, осуществляли с помощью геномного ресурса Ensembl Coturnix japonica 2.0 .
Статистические показатели рассчитывали в Microsoft Excel 2013. Вычисляли средние значения ( М ), стандвртные ошибки средних (±SEM), отмечали минимальные (min) и максимальные (max) значения, коэффициент вариации ( Cv , %).
Результаты. Содержание абдоминального жира в тушках 56-суточных перепелов F 2 из модельной ресурсной популяции варьировало от 0,01 до 10,46 г и составило в среднем 2,41±0,16 г. Следует отметить высокую вариабельность показателя и значительный разброс его значений у исследованной птицы — 78,7 %. При этом доля абдоминального жира от общей массы тушки изменялась от 0,01 % до 4,82 % при средней величине коэффициента вариации 73,2 %.
Анализ также выявил высокую вариацию абсолютного и относительного содержания абдоминального жира в тушках в группах перепелов F 2 _1-F 2 _12 (табл. 1).
-
1. Содержание абдоминального жира в тушках самцов у перепелов ( Coturnix japo-nica) F2 модельной ресурсной популяции (n = 146; виварий ФГБНУ ФИЦ ВИЖ им. академика Л.К. Эрнста, 2022-2023 годы)
Группа
n
Масса абдоминального жира, г
Доля абдоминального жира в тушке, %
M
±SEM
max
min
Cv , %
M
±SEM
max
min
Cv , %
F 2 _1
8
1,93
0,51
4,24
0,01
74,6
1,09
0,29
2,48
0,01
75,2
F 2 _2
15
2,95
0,48
6,10
0,10
63,5
1,53
0,25
3,19
0,06
63,2
F 2 _3
7
1,35
0,15
2,02
0,82
29,5
0,71
0,06
1,00
0,48
23,8
F 2 _4
18
2,49
0,40
7,64
0,35
68,6
1,31
0,21
4,03
0,22
67,3
F 2 _5
7
2,54
0,87
6,95
0,71
90,9
1,53
0,51
4,13
0,46
87,4
F 2 _6
14
2,84
0,68
10,46
0,55
89,7
1,47
0,31
4,82
0,30
80,1
F 2 _7
18
1,96
0,46
8,72
0,10
99,5
1,05
0,23
4,29
0,06
92,9
F 2 _8
11
2,30
0,52
5,39
0,10
75,1
1,32
0,29
2,92
0,08
71,7
F 2 _9
10
2,13
0,43
5,41
0,73
63,5
1,18
0,19
2,44
0,45
50,2
F 2 _10
6
2,13
0,66
3,63
0,01
76,3
1,22
0,35
1,99
0,01
71,0
F 2 _11
18
2,97
0,56
8,83
0,49
80,3
1,52
0,26
4,13
0,32
71,2
F 2 _12
14
2,32
0,51
5,36
0,01
78,6
1,31
0,28
3,04
0,01
76,2
В среднем
146
2,41
0,16
10,46
0,01
78,7
1,30
0,08
4,82
0,01
73,2
-
2. SNPs и потенциальные гены-кандидаты (p < 0,00001) , ассоциированные с отложением абдоминального жира в тушке 8-недельных самцов перепелов ( Cotur-nix japonica ) F 2 модельной ресурсной популяции (виварий ФГБНУ ФИЦ ВИЖ им. академика Л.К. Эрнста, 2022-2023 годы)
Хромосома
Число SNPs
SNP
р
Ген
в пределах SNP ±0,2 Mb
1
11
1:14239735
1,03E-05
-
GTSE1 , TRMU , RAMD4 , CERK
1:17986717
4,74E-05
-
IL17REL , MLC1 , MOV10L1 , PANX2 , TRABD , SELENOO , TUBGCP6 , HDAC10
1:161416406
8,12E-05
-
ATM , NPAT , ACAT1 , ELMOD1
1:161611015
7,37E-06
-
ALKBH8 , CWF19L2
1:161611163
3,26E-05
-
ELMOD1 , ALKBH8 , CWF19L2 , GUCY1A2
1:161950040
6,55E-06
-
GUCY1A2 , ASDHPPT , KBTBD3 , MSANTD4
1:162669404
6,99E-06
-
PDGFD
1:164041425
6,53E-05
-
PGR , ARHGAP42 , CNTN5
1:164300065
9,84E-06
CNTN5
-
1:18579785
9,30E-05
-
PLXNB2 , ZNF800 , GRM8
2
2
2:85104703
5,62E-05
-
TMX3
2:87734460
1,35E-05
GNAL
SPIRE1 , AFG3L2 , PRELID3A , MPPE1
7
7
7:11751354
6,63E-05
PARD3B , NRP2 , INO80D
7:11751372
6,63E-05
-
7:12205885
1,88E-05
-
DNAJC10 , PIKFYVE , CYP20A1 , NBEAL1 ,
7:12292293
1,88E-05
PDE1A
IDH1
7:13285939
6,29E-06
-
CWC22 , ZNF385B , SESTD1 , CCDC141
7:25004119
9,48E-05
RBMS1
LY75 , PLA2R1 , ITGB6
7:3916979
9,88E-05
-
AGAP1 , GBX2 , ASB18 , IQCA1 , ACKR3
8
2
8:17793153
9,35E-05
PTPRF
IPO13 , KDM4A , ST3GAL3
8:17793157
9,35E-05
-
17
2
17:4080998
4,98E-05
SH3GLB2
SPTAN1 , TBC1D13 , ENDOG , LRRC8A , PHYHD1 , NUP188 , TBC1D13 , PTPA , NTMT1
17:3724688
1,71E-05
SLC27A4
NAIF1 , EEIG1 , SH2D3C , DPM2 , AK1
19
1
19:601237
1,42E-05
-
CLDN4 , LIMK1 , CALN1 , MTMR4 , ABHD11 , METTL27 , SBDS , GALNT17
21
1
21:5674628
8,43E-05
TRIM62
WNT4 , P3H1 , ZMYND12 , PHC2 , USP48 , ECE1 , EIF4G3 , CDC42 , C1orf50 , PPIH , SLC2A1 , ALPL
24
1
24:2070512
4,36E-05
IGSF9B
OPCML , NCAPD3 , THYN1 , B3GAT1 , JAM3 , VPS26B , ACAD8
28
2
28:2442148
4,46E-05
USHBP1
REEP6 , THOP1 , GADD45B , ABHD8 ,
28:2436106
1,79E-06
NR2F6
ANO8 , YJEFN3 , MAU2 , NCAN
Примечан
ие.
Прочерки в таблице означают,
что в области выявленных SNPs не локализованы гены.
На рисунке 1 представлена диаграмма, отражающая распределение перепелов исследуемой выборки по содержанию абдоминального жира в тушке в зависимости от генотипа.
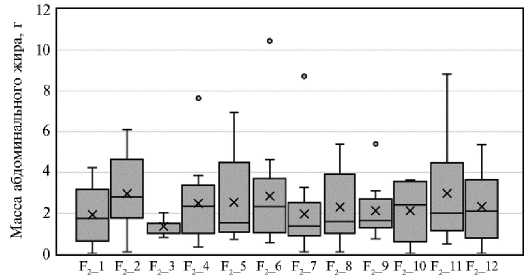
Рис. 1. Распределение самцов перепелов ( Coturnix japonica ) F 2 модельной ресурсной популяции по содержанию абдоминального жира в тушке в зависимости от генотипа (группы F 2 _1-F 2 _12, численность групп см. в табл. 1; виварий ФГБНУ ФИЦ ВИЖ им. академика Л.К. Эрнста, 2022-2023 годы). Приведены Me , M , min, max, выбросы единичных данных.
А
W'
S'
Chromosome
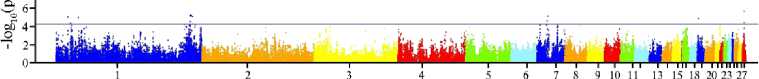
Рис. 2. Манхэттенский (А) и квантиль - квантильный (Q - Q) (Б) графики результатов GWAS для содержания абдоминального жира в тушке 8-недельных самцов перепелов ( Coturnix japonica ) из F 2 модельной ресурсной популяции. Манхеттен-плот отражает распределение однонуклеотидных мутаций (SNPs) в хромосомах перепелов до уровня достоверности - log10(p) в соответствии с предполагаемым вероятностным значением (p < 0,0001) и тестом Бонферрони (p < 1,05½10 - 8 ) для содержания абдоминального жира в
Б
Expected, -log10(p)
тушке; 1-28 — хромосомы, нижняя горизонтальная линия соответствует p < 0,0001, верхняя линия — p < 1,05½10 - 8 (виварий ФГБНУ ФИЦ ВИЖ им. академика Л.К. Эрнста, 2022-2023 годы).
Полногеномный анализ ассоциаций (whole-genome association study, GWAS) количества абдоминального жира в тушке показал, что у перепелов исследуемой популяции в возрасте 8 нед анализируемый показатель ассо- циирован с 29 SNPs (p < 0,00001), которые локализовались на хромосомах 1, 2, 7, 8, 17, 19, 21, 24 и 28 (рис. 2). Наибольшее число SNPs содержали хромосомы 1 и 7 (соответственно 11 и 7 SNPs), наименьшее — хромосомы 2, 17, 19, 21 и 24 (1-2 SNPs).
Выявленные SNPs (p < 0,00001) использовались для аннотирования генов-кандидатов, ассоциированных с отложением абдоминального жира у перепелов. Структурная аннотация выявила 124 гена, в том числе 11 генов, локализованных в области идентифицированных SNPs, — CNTN5 , GNAL , PDE1A , RBMS1 , PTPRF , SH3GLB2 , SLC27A4 , TRIM62 , IGSF9B , USHBP1 и NR2F6 (табл. 2). Перечисленные гены были идентифицированы на 8 хромосомах — CJA1 (1 ген), CJA2 (1 ген), CJA7 (2 гена), CJA8 (1 ген), CJA17 (2 гена), CJA21 (1 ген), CJA24 (1 ген) и CJA28 (2 гена).
Обнаруженные гены-кандидаты и значимые SNPs (p < 0,00001), ассоциированные с отложением абдоминального жира у перепелов, приведены в таблице 2.
Анализ информационных источников показал, что ни для одного из выявленных нами 11 генов-кандидатов с локализацией в области идентифицированных SNPs (CNTN5, GNAL, PDE1A, RBMS1, PTPRF, SH3GLB2, SLC27A4, TRIM62, IGSF9B, USHBP1, NR2F6) ранее не сообщалось о связи с отложением абдоминального жира у перепелов. Однако для семи выявленных генов-кандидатов было установлено влияние на липидный обмен и накопление жировых отложений у других видов животных и птицы. Так, сообщалось о связи генов PTPRF и GNAL с развитием, формированием и накоплением жировой ткани (34) и отложением абдоминального жира (35) у кур, генов TRIM62 и SLC27A4 — с толщиной (36) и липидным составом (37) шпика свиней. На лабораторных мышах показано влияние генов NR2F6, PDE1A и RBMS1 на адипогенез и липидный обмен, в частности на накопление жировых отложений в условиях холода (38) и ожирение (39-41).
Для других обнаруженных нами генов-кандидатов установлена связь с показателями мясной продуктивности и качеством мяса у других сельскохозяйственных животных и птицы. В частности, сообщалось о связи гена CNTN5 с эффективностью потребления корма у пекинских уток в возрасте до 42 сут (42), что может свидетельствовать о воздействии этого гена на пищевое поведение птицы и, следовательно, на накопление внутримышечного и брюшного жира. Также показано влияние гена CNTN5 на рН мяса длиннейшей мышцы спины у овец F 2 ресурсной популяции тексель ½ алтайская порода (43). Выявлена связь гена PTPRF с показателем живой массы у коров голштинской породы (44).
Таким образом, выполненные нами полногеномные исследования ассоциаций содержания абдоминального жира в тушках перепелов ресурсной популяции F 2 позволили с высокой достоверностью (p < 0,00001) выявить 29 SNPs на хромосомах 1, 2, 7, 8, 17, 19, 21, 24 и 28. В области выявленных SNPs установлены 11 генов-кандидатов, достоверно связанных с отложением абдоминального жира у перепелов в возрасте 8 нед. Проведенная функциональная аннотация показала участие семи идентифицированных генов в процессах липидного метаболизма у других видов сельскохозяйственных животных. Полученные данные составят основу для поиска ассоциаций выявленных мутаций с другими селекционно значимыми показателями продуктивности.
Список литературы Идентификация SNPS и генов-кандидатов, ассоциированных с отложением абдоминального жира у перепелов Coturnix japonica
- Фисинин В.И., Буяров В.С., Буяров А.В., Шуметов В.Г. Мясное птицеводство в регионах России: современное состояние и перспективы инновационного развития. Аграрная наука, 2018, 2: 30-38.
- Зыков С.А. Современные тенденции развития птицеводства. Эффективное животноводство, 2019, 4(152): 51-54.
- Priti M., Satish S. Quail farming: an introduction. International Journal of Life Sciences, 2014, 2: 190-193.
- Minvielle F. The future of Japanese quail for research and production. World’s Poultry Science Journal, 2004, 60(4): 500-507 (doi: 10.1079/WPS200433).
- Quaresma M.A.G., Antunes I.C., Gil Ferreira B., Parada A., Elias A., Barros M., Santos C., Partidário A., Mourato M., Roseiro L.C. The composition of the lipid, protein and mineral frac-tions of quail breast meat obtained from wild and farmed specimens of Common quail (Coturnix coturnix) and farmed Japanese quail (Coturnix japonica domestica). Poultry Science, 2022, 101(1): 101505 (doi: 10.1016/j.psj.2021.101505).
- Genchev A., Mihaylova G., Ribarski S., Pavlov A., Kabakchiev M. Meat quality and composition in Japanese quails. Trakia Journal of Sciences, 2008, 6(4): 72-82,
- Hu Z.-L., Park C.A., Reecy J.M. Bringing the Animal QTLdb and CorrDB into the future: meeting new challenges and providing updated services. Nucleic Acids Research, 2022, 50(D1): D956-D961 (doi: 10.1093/nar/gkab1116).
- Коршунова Л.Г., Карапетян Р.В., Комарчев А.С., Куликов Е.И. Ассоциации однонуклео-тидных замен в генах-кандидатах с хозяйственно полезными признаками у кур (Gallus gallus domesticus L.) (обзор). Сельскохозяйственная биология, 2023, 58(2): 205-222 (doi: 10.15389/agrobiology.2023.2.205rus).
- Sarsenbek A., Wang T., Zhao J., Jiang W. Comparison of carcass yields and meat quality between Baicheng-You chickens and Arbor Acres broilers. Poultry Science, 2013, 92: 2776-2782 (doi: 10.3382/ps.2012-02841).
- Yang Y., Wen J., Fang G.Y., Li Z.R., Dong Z.Y., Liu J. The effects of raising system on the lipid metabolism and meat quality traits of slow-growing chickens. Journal of Applied Animal Research, 2015, 43(2): 147-152 (doi: 10.1080/09712119.2014.928631).
- Fouad A., El-Senousey H. Nutritional factors affecting abdominal fat deposition in poultry: a review. Asian-Australasian Journal of Animal Sciences, 2014, 27: 1057-1068 (doi: 10.5713/ajas.2013.13702).
- Zhao S.M., Ma H.T., Zou S.X., Chen W.H., Zhao R.Q. Hepatic lipogenesis gene expression in broiler chicken with different fat deposition during embryonic development. Journal of Veterinary Medicine, 2007, 54: 1-6 (doi: 10.1111/j.1439-0442.2007.00898.x).
- Xing J., Kang L., Hu Y., Xu Q., Zhang N., Jiang Y. Effect of dietary betaine supplementation on mRNA expression and promoter CpG methylation of lipoprotein lipase gene in laying hens. Journal of Poultry Science, 2009, 46: 224-228 (doi: 10.2141/jpsa.46.224).
- Dikici A., Nacak B., Yel N., Zaimoğulları K., İpek G., Özer M. Quality characteristics and oxi-dative stability of chicken kavurma formulated with chicken abdominal fat as beef fat replacer. Journal of the Hellenic Veterinary Medical Society, 2022, 73(3): 4525-4534 (doi: 10.12681/jhvms.27738).
- Santos M.F., Lima D.A.S., Madruga M. S., Silva F.A.P. Lipid and protein oxidation of emulsified chicken patties prepared using abdominal fat and skin. Poultry Science, 2020, 99(3): 1777-1787 (doi: 10.1016/j.psj.2019.11.027).
- Li J., Cheng Y., Chen Y., Qu H., Zhao Y., Wen C., Zhou Y. Effects of dietary synbiotic supple-mentation on growth performance, lipid metabolism, antioxidant status, and meat quality in Par-tridge shank chickens. Journal of Applied Animal Research, 2019, 47(1): 586-590 (doi: 10.1080/09712119.2019.1693382).
- Sugiharto S., Pratama A.R., Yudiarti T., Wahyuni H.I., Widiastuti E., Sartono T.A. Effect of acidified turmeric and/or black pepper on growth performance and meat quality of broiler chick-ens. International Journal of Veterinary Science and Medicine, 2020, 8(1): 85-92 (doi: 10.1080/23144599.2020.1830691).
- Narinc D., Karaman E., Aksoy T. Effects of slaughter age and mass selection on slaughter and carcass characteristics in 2 lines of Japanese quail. Poultry Science, 2014, 93(3): 762-769 (doi: 10.3382/ps.2013-03506).
- Abou-Kassem D.E., El-Kholy M.S., Alagawany M., Laudadio V., Tufarelli V. Age and sex-related differences in performance, carcass traits, hemato–biochemical parameters, and meat quality in Japanese quails. Poultry Science, 2019, 98(4): 1684-1691 (doi: org/10.3382/ps/pey543).
- Lotfi E., Zerehdaran S., Azari M.A. Genetic evaluation of carcass composition and fat deposition in Japanese quail. Poultry Science, 2011, 90(10): 2202-2208 (doi: 10.3382/ps.2011-01570).
- Narinc D., Aksoy T., Karaman E., Aygun A., Firat M.Z., Uslu M.K. Japanese quail meat quality: Characteristics, heritabilities, and genetic correlations with some slaughter traits. Poultry Science, 2013, 92(7): 1735-1744 (doi: 10.3382/ps.2013-03075).
- Jiang M., Fan W.L., Xing S.Y., Wang J., Li P., Liu R.R., Li Q.H., Zheng M.Q., Cui H.X., Wen J., Zhao G.P. Effects of balanced selection for intramuscular fat and abdominal fat percentage and estimates of genetic parameters. Poultry Science, 2017, 96(2): 282-287 (doi: 10.3382/ps/pew334).
- Глазко В.И., Косовский Г.Ю., Глазко Т.Т., Федорова Л.М. ДНК маркеры и «микроса-теллитный код» (обзор). Сельскохозяйственная биология, 2023, 58(2): 223-248 (doi: 10.15389/agrobiology.2023.2.223rus).
- Wang D., Qin P., Zhang K., Wang Y., Guo Y., Cheng Z., Li Z., Tian Y., Kang X., Li H., Liu X. Integrated LC/MS-based lipidomics and transcriptomics analyses revealed lipid composition het-erogeneity between pectoralis intramuscular fat and abdominal fat and its regulatory mechanism in chicken. Food Research International, 2023, 172: 113083 (doi: 10.1016/j.foodres.2023.113083).
- Moreira G.C.M., Boschiero C., Cesar A.S.M., Reecy J. M., Godoy T. F., Trevisoli P. A., Cantão M.E., Ledur M.C., Ibelli A.M.G., Peixoto J.O., Moura M.T., Garrick D., Coutinho L.L. A genome-wide association study reveals novel genomic regions and positional candidate genes for fat deposition in broiler chickens. BMC Genomics, 2018, 19: 374 (doi: 10.1186/s12864-018-4779-6).
- Zhang H., Hu X., Wang Z., Zhang Y., Wang S., Wang N., Ma L., Leng L., Wang S., Wang Q., Wang Y., Tang Z, Li N., Da Y., Li H. Selection signature analysis implicates the PC1/PCSK1 region for chicken abdominal fat content. PLoS ONE, 2012, 7(7): e40736 (doi: 10.1371/journal.pone.0040736).
- Trevisoli P.A., Moreira G.C.M., Boschiero C., Cesar A.S.M., Petrini J., Margarido G.R.A., Le-dur M.C., Mourão G.B., Garrick D., Coutinho L.L. A missense mutation in the MYBPH gene is associated with abdominal fat traits in meat-type chickens. Frontiers in Genetics, 2021, 11(12): 698163 (doi: 10.3389/fgene.2021.698163).
- Zhang H., Shen L.Y., Xu Z.C., Kramer L.M., Yu J.Q., Zhang X.Y., Na W., Yang L.L., Cao Z.P., Luan P., Reecy J.M., Li H. Haplotype-based genome-wide association studies for carcass and growth traits in chicken. Poultry Science, 2020, 99(5): 2349-2361 (doi: 10.1016/j.psj.2020.01.009).
- Tavaniello S., Maiorano G., Siwek M., Knaga S., Witkowski A., Di Memmo D., Bednarczyk M. Growth performance, meat quality traits, and genetic mapping of quantitative trait loci in 3 gen-erations of Japanese quail populations (Coturnix japonica). Poultry Science, 2014, 93(8): 2129-2140 (doi: 10.3382/ps.2014-03920).
- Volkova N.A., Romanov M.N., Abdelmanova A.S., Larionova P.V., German N.Y., Vetokh A.N., Shakhin A.V., Volkova L.A., Anshakov D.V., Fisinin V.I., Narushin V.G., Griffin D.K., Sölkner J., Brem G., McEwan J.C., Brauning R., Zinovieva N.A. Genotyping-by-sequencing strategy for integrating genomic structure, diversity and performance of various Japanese quail (Coturnix ja-ponica) breeds. Animals, 2023, 13: 3439 (doi: 10.3390/ani13223439).
- Andrews S. FastQC: A quality control tool for high throughput sequence data; Version 0.10.1. Bioin-formatics Group, Babraham Institute, Cambridge, UK, 2012. Режим доступа: http://www.bioin-formatics.babraham.ac.uk/projects/fastqc. Дата обращения: 25.09.2023.
- Langmead B. bowtie2: a fast and sensitive gapped read aligner. Version 2.4.4. GitHub, Inc. 2021. Режим доступа: https://github.com/BenLangmead/bowtie2 Дата обращения: 25.09.2023.
- Turner S.D. qqman: an R package for visualizing GWAS results using Q-Q and Manhattan plots. Journal of Open Source Software, 2018, 3(25): 731 (doi: 10.21105/joss.00731).
- Gao Z., Ding R., Zhai X., Wang Y., Chen Y., Yang C-X., Du Z-Q. Common gene modules identified for chicken adiposity by network construction and comparison. Frontiers in Genetics, 2020, 11: 537 (doi: 10.3389/fgene.2020.00537).
- Zhang H., Du Z.Q., Dong J.Q. Detection of genome-wide copy number variations in two chicken lines divergently selected for abdominal fat content. BMC Genomics, 2014, 15: 517 (doi: 10.1186/1471-2164-15-517).
- Lee Y.S., Shin D. Genome-wide association studies associated with backfat thickness in landrace and Yorkshire pigs. Genomics Inform, 2018, 16(3): 59-64 (doi: 10.5808/GI.2018.16.3.59).
- Revilla M., Puig-Oliveras A., Crespo-Piazuelo D., Criado-Mesas L., Castelló A., Fernández A.I., Ballester M., Folch J.M. Expression analysis of candidate genes for fatty acid composition in adipose tissue and identification of regulatory regions. Scientific Reports, 2018, 8(1): 2045 (doi: 10.1038/s41598-018-20473-3).
- Maganti A., Chi J., Cohen P. Arx and Nr2f6 transcriptionally regulate visceral and brown fat phenotype. Diabetes, 2021, 70(S1): 201-LB (doi: 10.2337/db21-201-LB).
- Zhou B., Jia L., Zhang Z., Xiang L., Yuan Y., Zheng P., Liu B., Ren X., Bian H., Xie L., Li Y., Lu J., Zhang H., Lu Y. The nuclear orphan receptor NR2F6 promotes hepatic steatosis through upregulation of fatty acid transporter CD36. Advanced Science, 2020, 7(21): 2002273 (doi: 10.1002/advs.202002273).
- Kim N.J., Baek J.H., Lee J., Kim H., Song J-K., Chun K-H. A PDE1 inhibitor reduces adipo-genesis in mice via regulation of lipolysis and adipogenic cell signaling. Experimental & Molecular Medicine, 2019, 51: 1-15 (doi: 10.1038/s12276-018-0198-7).
- Dairi G., Al Mahri S., Benabdelkamel H., Alfadda A.A., Alswaji A.A., Rashid M., Malik S.S., Iqbal J., Ali R., Al Ibrahim M., Al-Regaiey K., Mohammad S. Transcriptomic and proteomic analysis reveals the potential role of RBMS1 in adipogenesis and adipocyte metabolism. Interna-tional Journal of Molecular Sciences, 2023, 24(14): 11300 (doi: 10.3390/ijms241411300).
- Li G-S., Liu W-W., Zhang F., Zhu F., Yang F-X., Hao J-P., Hou Z.-C. Genome-wide association study of bone quality and feed efficiency-related traits in Pekin ducks. Genomics, 2020, 112(6): 5021-5028 (doi: 10.1016/j.ygeno.2020.09.023).
- Zhao Y., He S., Huang J., Liu M. Genome-wide association analysis of muscle pH in Texel sheep ½ Altay sheep F2 resource population. Animals, 2023, 13(13): 2162 (doi: 10.3390/ani13132162).
- Hardie L.C., VandeHaar M.J., Tempelman R.J., Weigel K.A., Armentano L.E., Wiggans G.R., Veerkamp R.F., de Haas Y., Coffey M.P., Connor E.E., Hanigan M.D., Staples C., Wang Z., Dekkers J.C.M., Spurlock D.M. The genetic and biological basis of feed efficiency in mid-lactation Holstein dairy cows. Journal of Dairy Science, 2017, 100(11): 9061-9075 (doi: 10.3168/jds.2017-12604).


