Использование SNP-маркеров для идентификации локусов PL5 и PL8, контролирующих устойчивость подсолнечника к Plasmopara halstedii (Farl.) Berl. et de Toni
Автор: Рамазанова С.А., Антонова Т.С., Бадьянов Е.В., Сыксин С.В., Меркулов П.Ю.
Рубрика: Селекция и семеноводство сельскохозяйственных растений
Статья в выпуске: 3 (179), 2019 года.
Бесплатный доступ
Ложная мучнистая роса, вызываемая оомицетом Plasmopara halstedii (Farl.) Berl. et de Toni, является одной из самых вредоносных болезней подсолнечника. К настоящему времени в мире обнаружено 50 физиологических рас патогена. Эффективным методом контроля возбудителя болезни является введение доминантных генов устойчивости к нему в растение-хозяина. Современные технологии генотипирования с помощью ДНК-маркеров позволяют контролировать наличие этих генов на каждом этапе селекции. Разработаны и апробированы на линиях-дифференциаторах устойчивости подсолнечника, входящих в международный тест-набор для идентификации рас P. halstedii 13 SNP-маркеров генов Pl5 и Pl8, контролирующих устойчивость к 16 расам. Из них были отобраны три SNP-маркера: 8R2, 5S2 и 5 S3, пригодные для идентификации генов Pl5 и Pl8, контролирующих устойчивость подсолнечника к возбудителю ложной мучнистой росы.
Днк-маркеры, мас, r-гены, устойчивость, подсолнечник
Короткий адрес: https://sciup.org/142222537
IDR: 142222537 | УДК: 633.854.78:575.113.1
Текст научной статьи Использование SNP-маркеров для идентификации локусов PL5 и PL8, контролирующих устойчивость подсолнечника к Plasmopara halstedii (Farl.) Berl. et de Toni
Введение. Повсеместно распространенной и крайне опасной болезнью подсолнечника является ложная мучнистая роса, вызываемая оомицетом Plasmopara halstedii (Farl.) Berl. et de Toni. Потери урожая при условиях, благоприятных для развития болезни, могут составлять 50–
70 %. По данным разных авторов, в мире обнаружено в 2006 г. 35 рас, 41 – в 2014 г. и 50 – в 2019 г. [1; 2; 3; 4; 5]. Каждый год публикуются сообщения о возникновении новых рас. При этом ранние, менее вирулентные патотипы, такие как расы 100, 300, 700 и другие, замещаются новыми, более агрессивными [1; 2].
Одним из самых эффективных методов контроля над P. halstedii является введение в растение-хозяина доминантных генов Pl , обеспечивающих устойчивость к болезни. К настоящему времени уже картировано 33 гена Pl , расположенных в пяти группах сцепления LG: 1, 2, 4, 8 и 13 [8].
В настоящее время перспективными для использования в селекции являются кластеры генов Pl 5 и Pl 8 , контролирующие устойчивость к 16 расам P. halstedii . . Они были клонированы с помощью метода ПЦР с вырожденными праймерами, построенными на основе аминокислотной последовательности домена NBS-LRR [6; 7]. Авторы показали, что гены Pl 5 и Pl 8 относятся к non-TIR-NBS-LRR классу R-генов и картированы в тринадцатой группе сцепления на генетической карте SSR. Проведенные в последние годы исследования показали, что между генами Pl 5 и Pl 8 расположены Pl 21 , Pl 31 и Pl 32. На этой же хромосоме картирован ген Pl 22 [8]. Это, вероятно, связано с тем, что в области гена Pl 8 расположены 14 RGCs (Re-sistence Gene Candidate), т.е. «кандидатов» в гены устойчивости, и поэтому многие гены картируют в этом районе.
Целью наших исследований было разработать молекулярные маркеры генов Pl 5 и Pl 8 и изучить возможность их использования для определения наличия этих локусов в линиях-дифференциаторах устойчивости подсолнечника, используемых для идентификации рас P. halstedii .
Материалы и методы. Объектом исследования служили 14 линий-дифференциаторов устойчивости подсолнечника, входящих в международный тест-набор для идентификации рас P. halstedii .
Для выделения ДНК применяли метод, основанный на использовании лизирующего буфера, содержащего гексадеци-лтриметиламмоний бромид (СТАВ), с модификациями [11; 12]. Концентрацию ДНК в полученном препарате определяли визуально по интенсивности свечения пробы объемом 10 мкл в ультрафиолетовом свете в 1%-ном агарозном геле с добавлением 2 мкл бромистого этидия. Электрофорез препаратов ДНК проводили при напряжении 100 V в течение 30 мин в трис-ацетатном буферном растворе (ТАЕ).
Для ПЦР-анализа применили 13 пар праймеров, разработанных для маркирования локуса Pl 8 (табл. 1).
Таблица 1
Характеристика праймеров для маркирования локуса Pl 8
|
Наз ва-ние |
Последовательность |
Размер ампликона (п.н.) |
|
|
8R1 |
F:GATGATTTGGTATTGACGACAGG R:AGAAAGAGATGTGACTAAGCGTAATG G |
642 |
|
|
8R2 |
F: R |
CATTGGATCAACCCAAAAGC : ACGAGTCTGGTAAATCATGGG |
276 |
|
8R3 |
F: R |
CTGCTGCTGCCCTCG : AGAGCAATAAGCAAACAATCGC |
668 |
|
8R4 |
F: R |
AGTTGCCTGAGAATGTTGGC : CCAACTCGACATATCTTCAAACC |
608 |
|
8R5 |
F: R |
AGCGTTAGATGCTTCGTTATCG : CCCATATTGACAAAGAGTTGAGG |
632 |
|
8R6 |
F: R |
AGCGTTAGATGCTTCGTTATCG : CGTCTCTGGTAGATCGTTCACC |
624 |
|
8R7 |
F: R |
ACGACATCGTACCACCATCC : CCGATACCATACCTGAAACCG |
556 |
|
5R2 |
F: R |
CTTCTTCTTCTTTCCCTGTAGTCG : ATGTAACCCAACCCAACTCG |
601 |
|
8S1 |
F: R |
AAATGTTGCAGGGACATATAACC : TCTTCAACTCTAGTCAAGGGTGG |
470 |
|
8S2 |
F: R |
GAATCTCTCATATCCCCTACATCCC : ATACCATACCATACCTGAAACCGG |
379 |
|
5S1 |
F: R |
TCATATGCTCTTGTTTACTAGGATGGC : GGCGAAATTGGTTCCCG |
171 |
|
5S2 |
F: R |
CCATACAAATCGTTGCAGTTCC : GTCTGGTAAATCATGGATCAACTCC |
423 |
|
5S3 |
F: R |
AGTGGGGGGATCTAAATTTTCC : CGTCTCTGGTAGATCGTTCACC |
230 |
Полимеразную цепную реакцию выполняли в реакционной смеси (25 мкл) следующего состава: 67 мМ Трис-HCl (рН 8,8); 16,6 мM сульфата аммония; 1,5– 3,0 мM MgCl 2 ; 0,01 % Tween 20; по 0,2 мM дезоксирибонуклеозидфосфатов; по
10 пМ праймеров; 10 нг матричной ДНК и 1 ед. рекомбинантной термостабильной ДНК-полимеразы (НПО «СибЭнзим», Россия). Реакции проводили в термоциклере S1000тм (BioRad, США) при следующих температурных режимах: начальная денатурация при 95 °С в течение 5 мин, далее 37 циклов с последовательной сменой температур: денатурация при 95 ºС, в течение 30 сек., отжиг праймера при 63 ºС – 30 сек., элонгация при 72 ºС – 1 мин 24 сек., и заключительная элонгация при 72 ºС в течение 10 мин.
Электрофорез продуктов амплификации проводили в геле, содержащем 2 % агарозы и SВ-буфер, с использованием камеры для горизонтального электрофореза SE-2 (Хеликон, Россия) при напряжении 200 В, силе тока 100 мА, в течение 30 мин. Для некоторых праймеров электрофорез дополнительно проводили в 8%-ном акриламидном геле в камере для вертикального электрофореза (Хеликон, Россия). Гели окрашивали бромистым этидием. Для визуализации и документирования результатов электрофореза применяли систему цифровой документации видеоизображения BIO-PRINT (Vilber Lourmat, Франция).
Результаты и обсуждение. На начальном этапе работы по поиску ДНК-маркеров для генов устойчивости Pl5 и Pl8 к P. halstedii исследовали молекулярногенетический полиморфизм шести STS (Sequence-Tagged Site) локусов ДНК [6; 7]. В результате ПЦР с этими праймерами не было получено специфичных фрагментов ДНК, характерных для линий XRQ, YVQ, 803-1, PM-17 и DM-2 – носителей кластеров генов Pl5/Pl8 [9]. Нам не удалось разделить линии на устойчивые и восприимчивые так, как это описано в работе Radwan с соавторами [6; 7]. По нашему мнению, это связано с большим размером фланкируемого праймерами участка ДНК и, как следствие, амплификаций фрагментов длиною более 1000 пар нуклеотидов. К тому же сравнение нуклеотидных последовательностей этих ге- нов показало наличие большого количества участков ДНК, совпадающих по нуклеотидному составу. Это также создает проблемы при маркировании данных локусов.
Поэтому на базе известных последовательностей 13 STS-маркеров [6] нами были созданы SNP-праймеры (Single Nucleotide Polymorphism). Работа проводилась на кафедре генетики, биотехнологии и семеноводства МСХА имени К.А. Тимирязева. С целью упрощения проведения исследования и анализа результатов для амплификации были выбраны небольшие участки: от 171 до 668 п.н. Их характеристика и нуклеотидный состав представлены в таблице 1. Температура отжига для этих праймеров подбиралась с минимальными пошаговыми различиями, которые позволили бы их комплексное применение в ПЦР -смеси в дальнейшем.
На начальном этапе разработанные SNP-праймеры апробировали на 14 линиях-дифференциаторах устойчивости подсолнечника из международного тест-на-бора для идентификации рас P. halstedii. Каждая из этих линий содержит один или несколько генов Pl, обеспечивающих их устойчивость к определенным расам патогена. Подробная информация об этих линиях, соответствующих им генах устойчивости и группах сцепления, в которых они картированы на генетической карте SSR-локусов, опубликована ранее [9; 10; 12–21].
Результаты ПЦР с разработанными SNP-маркерами показали, что только с двумя (5R2 и 5S1) не было отжига праймера на матрице, с остальными одиннадцатью были получены продукты амплификации.
По локусу 8R1 при амплификации ДНК линий подсолнечника выявлен фрагмент длиною около 642 п.н. только у линии DM-2 – носителя генов Pl5, Pl11, Pl12 [10]. У остальных линий, содержащих локус Pl5 (803-1, YVQ, РМ-17 и XRQ), рав- но как и у линий с его отсутствием, этой фракции выявлено не было.
С праймером 8R2 получены продукты амплификации разной длины (рис. 1). У линий XRQ и DM-2 получены фракции, отличающиеся от остальных (дорожки 2 и 14). Также у линий YVQ и PM-17 (дорожки 9–10) обнаружены фрагменты одной длины, не выявленные у остальных. Однако для линии 803-1 не получено таких фрагментов (дорожка 13). По нашему мнению, апробацию этого праймера необходимо продолжить на других линиях, являющихся носителями генов Pl 5 и Pl 8.
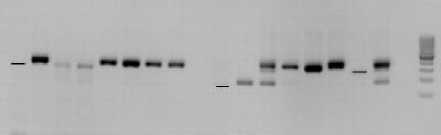
1 2 3 4 5 6 7 8 9 10 И 12 13 14 15 16 К"М
Рисунок 1 – Фореграмма продуктов амплификации ДНК линий-дифференциаторов устойчивости подсолнечника с праймером 8R2. Дорожки: 1 – RHA419;
2 – XRQ; 3, 4 – 83HR4RM; 5, 6 – HIR 34;
7, 8 – PSC8; 9 – YVQ; 10 – PM-17; 11 – HAR-5;
12 – HAR-4; 13 – 803-1; 14 – DM-2;
15 – RHA274; 16 – RHA265, Kˉ – отрицательный контроль, M – маркер молекулярного веса, 2019 г. (ориг.)
С праймером 8R3 отжиг состоялся, однако вместо ожидаемого фрагмента длиною 668 п.н. была выявлена фракция размером около 1100 п.н. Эти фрагменты присутствуют почти у всех линий. Аналогичный результат получен и с праймером 8R4. Получены фрагменты ДНК разных размеров: от 750 до 800 п.н., однако не было выявлено фрагмента специфичного для линий с локусами Pl 5 и Pl 8. С праймером 8R7 также не удалось получить ожидаемый фрагмент размером 556 п.н., а у всех образцов был амплифицирован фрагмент длиною 100 п.н. Точно также и с праймером 8S1 не было получено нужной фракции и не выявлено полиморфизма.
C праймером 8R5 получена фракция размером 632 п.н. только у линий RHA-19 и HA-335, носителей генов Pl arg и Pl 6 соответственно. У остальных линий не выявлено амплифицированных фрагментов.
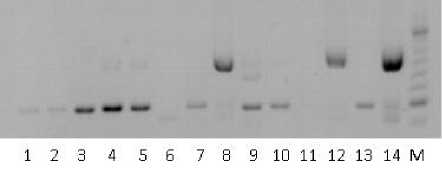
Рисунок 2 – Фореграмма продуктов амплификации ДНК линий-дифференциаторов устойчивости подсолнечника с праймером 8S2. Дорожки: 1 – RHA419, 2 – XRQ, 3 – 83HR4RM, 4 – HIR 34, 5 – PSC8, 6 – YVQ, 7 – Ha335, 8 – HAR-5, 9 – HAR-4, 10 – 803-1, 11 – PM-17, 12 – DM-2, 13 – RHA274, 14 – RHA265, M – маркер молекулярного веса, 2019 г. (ориг.)
На рисунке 2 показано, что при амплификации ДНК с праймером 8S2 у девяти линий получены четкие фракции размером 500 п.н., пока не идентифицированные. У линий YVQ (дорожка 6) (носитель гена Pl 8 ) и RHA265 выявлена фракция длиною 423 п.н. По нашему мнению, маркер 8S2 можно использовать для идентификации данного локуса, однако это требует дополнительных исследований.
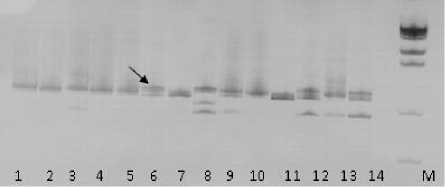
Рисунок 3 – Фореграмма продуктов амплификации ДНК линий дифференциаторов устойчивости подсолнечника с праймером 8S3. Дорожки: 1 – RHA419, 2 – XRQ, 3 – 83HR4RM, 4 – HIR 34, 5 – PSC8, 6 – YVQ, 7 – Ha335, 8 – HAR-5, 9 – HAR-4, 10 – 803-1, 11 – PM-17, 12 – DM-2, 13 – RHA274, 14 – RHA265,
M – маркер молекулярного веса, 2019 г.
(ориг.)
С праймером 8S3 были получены фракции размером 230 п.н., но полиморфизм удалось выявить только при электрофорезе в 8%-ном акриламидном геле (рис. 3). Так же, как и в случае с праймером 8S2, у линии YVQ (носитель гена Pl 8 ) выявлены фракции, не обнаруженные у других линий. По нашему мнению, SNP-праймеры 8S3 и 8S2 пригодны для идентификации локуса Pl 8 в селекционном материале ВНИИМК.
Заключение. Таким образом, наши исследования показали, что три сконструированных нами SNP-праймера – 8R2, 5S2 и 5S3 можно использовать для маркирования локусов Pl 5 и Pl 8 в линиях подсолнечника. Эти маркеры могут стать звеньями системы ДНК-маркеров для идентификации указанных генов и проведения маркер-вспомогательной селекции на устойчивость подсолнечника к возбудителю ложной мучнистой росы.
Список литературы Использование SNP-маркеров для идентификации локусов PL5 и PL8, контролирующих устойчивость подсолнечника к Plasmopara halstedii (Farl.) Berl. et de Toni
- Gulya T.J. Distribution of Flasmopara halstedii races from sunflower around the world // Advances in Downy Mildew Research / Lebeda A., Spencer-Phillips P.T.N. (eds). - Czech Republic, Olomouc, 2007. - Vol. 3. - Р. 121-134.
- Viränyi F., Gulya T.J., Tourvierille de Labrouhe D. Recent changes in the pathogenic variability of Plasmopara halstedii (sunflower downy mildew) populations from different continents // Helia. - 2015. - V. 38. - P. 149-162.
- Sedlářová M., Pospíchalová R., Drábková Trojanová Z., Bartůšek T., Slobodianová, L., Lebeda A. First report of Plasmopara halstedii new races 705 and 715 on sunflower from the Czech Republic - short communication // Plant Protect. Sci. - 2016. - V. 52. - 182187. DOI: 10.17221/7/2016-PPS
- Spring O., Zipper R. New highly aggressive pathotype 354 of Plasmopara halstedii in German sunflower fields // Plant Protect. Sci. - 2018. - V. 54. - P. 83-86. DOI: 10.17221/99/2017-PPS
- Spring О. Spreading and global pathogenic diversity of sunflower downy mildew - Review // Plant Protect. Sci. - 2019. - V. 55. - Р. 149-158. DOI: 10.17221/32/2019-PPS
- Radwan O., Bouzid, M.F., Vear F., Philippon J., Tourvieille de Labrouhe D., Nicolas F., Mouzeyar S. Identification of non-TIR-NBS-LRR markers linked to the locus for resistance to downy mildew in sun flower // Theoretical and Applied Genetics. - 2003. - V. 106. - P. 1438-1446.
- DOI: 10.1007/s00122-003-1196-1
- Radwan O., Bouzidi M.F., Nicolas P., Mouzeyar S. Development of PCR markers for the locus for resistance Flasmopara halstedii in sunflower, Helianthus annuus L. from complete CC-NBS-LRR sequences // Theoretical and Applied Genetics. - 2004. -V. 109. - P. 176-185.
- DOI: 10.1007/s00122-004-1613-0
- Pecrix Y., Penouilh-Suzette C., Munos S., Vear F., Godiard L. Ten broad spectrum resistances to downy mildew physically mapped on the sunflower genome // Front. Plant Sci. - 2018. - V. 9.
- DOI: 10.3389/fpls.2018.01780
- Рамазанова С.А., Антонова Т.С. К вопросу о маркировании локусов P1, контролирующих устойчивость подсолнечника к возбудителю ложной мучнистой росы // Масличные культуры. - 2019. - Вып. 1 (177). - С. 17-23.
- Rahim M., Jan C.C., Gulya T.J. Inheritance of resistance to sunflower downy mildew races 1, 2 and 3 in cultivated sunflower // Plant Breeding. - 2002. - V. 121. - P. 57-60.
- Jocic S., Miladinovic D., Imerovski I., Dimitrijevic A., Cvejic S., Nagl N., Kondic-Spika A. Towards sustainable downy mildew resistance in sunflower // Helia. - 2012. - No 35. - P. 61-72.
- Dußle C.M., Hahn V., Knapp S.J., Bauer E. Plarg from Helianthus argophyllus is unlinked to other known downy mildew resistance genes in sunflower // Theoretical and Applied Genetics. - 2004. - V. 109 (5). - Р. 1083-1086.
- DOI: 10.1007/s00122-004-1722-9
- Imerovski I., Dimitrijevic A., Miladinovic D., Jocic S., Dedic B., Cvejic S., Surlan-Momirovic G. Identification and validation of breeder-friendly DNA markers for Plarg gene in sunflower // Molecular Breeding. -2014. - V. 34 (3). - P. 779-788.
- DOI: 10.1007/s11032-014-0074-7
- Wieckhorst S., Bachlava E., Dußle C.M., Tang S., Gao W., Saski C., Bauer E. Fine mapping of the sunflower resistance locus Plarg introduced from the wild species Helianthus argophyllus // Theoretical and Applied Genetics - 2010. - V. 121 (8). - Р. 1633-1644.
- DOI: 10.1007/s00122-010-1416-4
- Bert P.F., Tourvieille de Labrouhe D., Philippon J., Mouzeyar S., Jouan I., Nicolas P., Vear F. Identification of a second linkage group carrying genes controlling resistance to downy mildew (Plasmopara halstedii) in sunflower (Helianthus annuus L.) // Theoretical and Applied Genetics. - 2001. - V. 103. - P. 992-997.
- Bertero de Romano A., Romano C., Bulos M., Altieri E., Sala C. A new gene for resistance to downy mildew in sunflower // Proc. Intern. Symposium "Sunflower Breeding on Resistance to Diseases", Russia, Krasnodar, 2010, June 23-24. - P. 141-146.
- Mulpuri S., Liu Z., Feng J., Gulya T. J., Jan C.-C. Inheritance and molecular mapping of a downy mildew resistance gene, Pl13 in cultivated sunflower (Helianthus annuus L.) // Theoretical and Applied Genetics. -2009. - V. 119 (5). - P. 795-803.
- DOI: 10.1007/s00122-009-1089-z
- Liu Z., Gulya T.J., Seiler G.J., Vick B.A., Jan C-C. Molecular mapping of the Pl16 downy mildew resistance gene from HA-R4 to facilitate marker-assisted selection in sunflower // Theoretical and Applied Genetics. -2012. - V. 125. - P. 121-131.
- Molinero-Ruiz M. L., Dominguez J., Melero-Vara J.M. Races of Isolates of Plasmopara halstedii from Spain and studies on their virulence // Plant Disease - 2002. - V. 86 (7). - P. 736-740.
- DOI: 10.1094/pdis.2002.86.7.736
- Gedil M.A., Slabaugh M.B., Berry S., Johnson R., Michelmore R., Miller J., Gulya T., Knapp S.J. Candidate disease resistance genes in sunflower cloned using consereved nucleotide-binding site motifs: Genetic mapping and linkage to the downy mildew resistance gene Ph // Genom. - 2001. - V. 44 (2). - Р. 205-212.
- Ahmed S., Tourvieille de Labrouhe D., Delmotte F. Emerging virulence arising from hybridisation facilitated by multiple introductions of the sunflower downy mildew pathogen Plasmopara halstedii // Fungal Genetics and Biology. - 2012. - V.49 (10). - P. 847-855.
- DOI: 10.1016/j.fgb.2012.06.012


