Классификация отечественных сортов капусты Brassica oleracea L. c использованием SSR маркеров
Автор: Домблидес А.С., Домблидес Е.А., Бондарева Л.Л., Пивоваров В.Ф.
Журнал: Овощи России @vegetables
Рубрика: Селекция и семеноводство сельскохозяйственных растений
Статья в выпуске: 5 (43), 2018 года.
Бесплатный доступ
Для получения новых селекционных форм среди разнообразных представителей вида Brassica oleraceae L. (CC, 2n = 18) необходимо знать генетическую основу используемого селекционного материала. Традиционные сорта и новые сорта, гибриды, появляющиеся в последнее время, составляют основные генетические ресурсы. Классификация коллекций с использованием ДНК маркеров позволяет выделить ценные генотипы и установить родословные селекционного материала, с тем, чтобы в дальнейшем получать новые формы с набором ценных признаков. Использование микросателлитных маркеров (SSR) в виде B. oleraceae L. показали высокую эффективность по выявлению полиморфизма между разновидностями, между сортами и внутри сортов. В данной работе было взято 16 пар праймеров для амплификации микросателлитных локусов геномной ДНК 24 отечественных селекционных образцов капусты. Все оцененные локусы характеризовались высокой информативностью: 14 из 16 имели уровень PIC > 0,5. На основе полученных данных была построенная дендрограмма на основе коэффициента Джаккарта...
Капуста, ssr маркеры, генетические расстояния, микросателлитные локусы, полиморфизм
Короткий адрес: https://sciup.org/140238383
IDR: 140238383 | УДК: 635.33:573.6 | DOI: 10.18619/2072-9146-2018-5-9-12
Текст научной статьи Классификация отечественных сортов капусты Brassica oleracea L. c использованием SSR маркеров
Домблидес А.С.* – кандидат с.-х наук, зав. лабораторией генетики и цитологии Домблидес Е.А. – кандидат с.-х. наук, зав. лабораторией биотехнологии Бондарева Л.Л. – доктор с.-х. наук, зав. лабораторией селекции и семеноводства капустных культур
Китай, Индия и Россия являются основными производителями капусты в мире, в то время как Россия оставляет за собой лидерство в ее потреблении. Капуста обладает антиоксидантными свойствами по причине содержания полифенолов, антоцианов, галловой, ванилиновой и кумаровой кислот. В биохимическом составе есть противовоспалительные вещества, такие как глутамин и флавоноиды. Капуста также является прекрасным источников витамина С и глюкозинола-тов. Из вида B. oleracea L. (CC, 2n = 18) наиболее известной является капуста белокочанная (B. oleracea L. сonvar. capitata (L.) Alef. var. capitata L. f.alba). В меньшей степени используют капусту краснокочанную (B. oleracea L. сonvar. capitata (L.) Alef. var. capitata L. f. rubra), савойскую (B. oleracea L. convar. capitata (L.) Alef. var. sabauda L.), цветную (B. oleracea L. convar.
botrytis (L.) Alef. var. botrytis L.), брокколи ( B. oleracea L. convar. botrytis (L.) Alef. var. italica Plenck), брюссельскую ( B. oleracea L. convar. gemmifera (DC.) Gladis var. gemmifera DC.), кольраби ( B. oleracea L. convar. acephala (DC.) Alef. var. gongylodes L.) и декоративную листовую ( B. oleracea convar. acephala (DC.) Alef. var. sabellica L.). Традиционные сорта и высокопродуктивные гибриды составляют основу генетической коллекции вида B. oleracea L. Однако для рационального использования генетических ресурсов и получения новых форм с высоким показателем гетерозиса и стабильности проявления хозяйственно ценных признаков необходимо знать генетическую основу используемого генетического материала. Кроме того, с появлением нового селекционного материала необходимо отличать все используемые генотипы и идентифицировать среди них одинаковые или близкородственные. Использование ДНК маркеров на основе микросателлитных повторов (SSR) облегчает задачу точной генетической классификации селекционных образцов. Относительные преимущества микросателлитных маркеров над другими заключаются в их надежности, воспроизводимости и высокой способности выявления генетического полиморфизма (Formisano et al. 2012; Powel et al. 1996). Микросателлитные маркеры достаточно много используют для генетической классификации вида B. oleracea L. Получены данные о генетических взаимоотношениях как между разновидностями капусты, так и среди сортов этих разновидностей (Tonguc and Griffiths, 2004). В другом исследовании уже 91 коммерческий образец B. oleracea L. был проанализирован на основе изменчивости микросателлитных маркеров, где была выявлена генетическая структура среди представителей шести разновидностей (Izzah и др. 2013). Филогенетические связи между разновидностями были установлены на основе изменчивости микросателлитов также в целом виде B. oleracea L. (Артемьева и др., 2009). Даже селекционные образцы только капусты белокочанной показали сильную генетическую изменчивость в микросател-литных локусах с возможностью точного типирования каждого растения в образце (Шаптуренко, 2016). Основываясь на предыдущих результатах, необходимо отметить, что фактически нет публикаций, посвященных генетической классификации традиционных отечественных сортов и разновидностей капусты. Несмотря на то, что известны работы по изучению генетического разнообразия сортов, в данной работе мы использовали большее количество образцов для изучения родословной старых сортов и соотнесение их с известными сортотипами.
Целью исследований была генетическая классификация и типирование разновидностей и сортов капусты на основе изменчивости известных микросателлитных локусов для планирования селекционной работы с использованием MAS (маркер ассоциированной селекции).
Материалы и методы
Растительный материал. Для исследований были взяты представители вида B. oleracea L. коллекции ФГБНУ ФНЦО, включающие 12 сортов и гибридов капусты белокочанной (var. capitata L. f. alba ): Северянка F 1 , Аврора F 1 , Стахановка 1513, Парус, Белорусская 455, Амагер 611, Подарок 2500, Зимовка 1474, Слава 1305, Слава грибовская 231, Номер первый гри-бовский 147, Июньская 3200; два сорта капусты краснокочанной (var. capitata L. f. rubra ): Гако 741, Каменная головка 447; два сорта капусты савойской (var. sabauda L.): Вертю 1340, Московская кружевница; два сорта капусты цветной (convar. botrytis (L.) Alef. var. botrytis L.): Ранняя грибовская 1355, Полярная звезда; один сорт брокколи (convar. botrytis (L.) Alef. var. italica Plenck): Тонус; один сорт кольраби (convar. acephala (DC.) Alef. var. gongylodes L.): Венская белая 1390; один сорт капусты листовой декоративной (convar. acephala (DC.) Alef. var. sabellica L.): Малиновка; один сорт капусты брюссельской
(convar. gemmifera (DC.) Gladis var. gemmifera DC.): Геркулес 1342 и один сорт капусты китайской ( B. rapa L.), используемый как внегрупповой контроль.
Выделение ДНК. Ткань молодых листочков от пяти растений каждого образца подвергали гомогенизации в экстракционном буфере при помощи прибора TissueLyser II Qiagen, Германия. Частота и время гомогенизации составляли 27 сек и 2 мин соответственно. К гомогенезированной ткани добавляли 200 мкл экстракционного буфера. Каждый образец подвергали обработке Протеиназой К. Дальнейшую экстракцию ДНК проводили на основе CTAB метода с использованием набора реагентов ≪ Сорб-ГМО-Б ≫ ( ≪ Синтол ≫ , Россия), куда входят все основные компоненты для выделения согласно прилагаемому протоколу.
Конечную чистоту и концентрацию тотальной ДНК определяли в спектрофотометре Smart Spec Plus (BioRad, USA). Собирали данные абсорбции при 260 nm и 280 nm для вычисления концентрации выделенной ДНК. Соотношение A260 к A280 определяет чистоту выделенной ДНК, отбирали образцы с данным соотношением ДНК около 1,8. После выделения раствор ДНК разбавляли до нужной концентрации и хранили в холодильнике для непосредственной работы при +2°С; для длительного хранения и последующего анализа – при -20°С.
SSR анализ. Для проведения микросателлитного анализа было использовано 16 микросателлитных локусов с известными последовательностями праймеров для амплификации, показывающих при анализе PIC праймера не менее 0,5 (Tongucё and Griffiths, 2004; Louarn et al., 2007) (табл.).
Базовую постановку ПЦР проводили в объеме 25 μl, включая 1x ПЦР буфер Б, 2,5 mM MgCl 2 , 0,25 mM каждого dNTP, 0,3 μM каждого праймера, 1,5 единиц Taq ДНК-полимеразы («Синтол», Россия) и 3 μl ДНК каждого исследуемого растительного образца.
Для проведения амплификации использовали прибор: С1000 Touch (BioRad). Базовый протокол амплификации состоял из шага денатурации 2-5 мин при 92...95°С; шага отжига 30 сек при температуре от 52°С до 58°С (в зависимости от пары праймеров); шага элонгации 30 сек – 1 мин при 72°С. Программа была рассчитана на 35 циклов амплификации.
Анализ ПЦР продуктов
Полученные продукты амплификации разгоняли методом вертикального электрофореза с использованием системы Sequi-Gen GT (BioRad) в 6% полиакриламидном секвенирую-щем геле при напряжении 1600 вольт и продолжительности 1,5-2 час.
После электрофореза гели подвергали окрашиванию раствором бромистого этидия и затем документировали с использованием системы ChemiDoc XRS+ (BioRad). Размеры амплифицированных фрагментов определяли в сравнении с маркером молекулярных масс GeneRuler100 bp plus DNA ladder, Thermo scientific. Полученные цифровые фотографии продуктов амплификации анализировали с использованием программы Image Lab 3.0 (BioRad).
Математическая обработка данных
Значение и величина информационного полиморфизма (анг. рolymorphism information content, PIC) для каждого праймера были посчитаны согласно A.Kumar и др., 2014. Для анализа ДНК-спектров каждому компоненту присваивали соответствующий порядковый номер, а затем данные переводили в бинарный вид, то есть присутствие и отсутствие компонента в спектре кодировали соответственно цифрами 1 или 0. В качестве критерия парного сходства был выбран коэффициент Джаккарда (Jaccard coefficient 1908). Кладограмма была построена методом UPGMA с использованием программы Genstat 6 для Windows (Genstat 6 Committee, Numerical Algorithms Group, Oxford, UK).
Таблица. Характеристика микросателлитных маркеров Table. Characteristics of microsatellite markers
|
Название маркера GenBank NCBI |
Происхождение |
мотив |
Ta (С°) |
PIC |
|
AF051772 |
reproductive meristem gene 1 (REM1) mRNA |
(gaa)5 |
55 |
0,81 |
|
AF458409 |
deoxycytidine deaminase (DCTD1), mRNA |
(aga)6 |
55 |
0,9 |
|
AJ427337 |
mRNA for calmodulin 1 (cam1 gene) |
(ga)5 |
55 |
0,61 |
|
BZ523957 |
genomic clone BOKAH45 |
(ttg)6 |
49,3 |
0,86 |
|
CC969431 |
genomic clone BOIAB19 |
(ga)6 |
54,5 |
0,61 |
|
CC969459 |
genomic clone BOIAB94 |
(cgg)5 |
55 |
0,25 |
|
CC969497 |
genomic clone BOIAA26 |
(tgc)5 |
56,2 |
0,83 |
|
CC969507 |
genomic clone BOIAB15 |
(ct)5 |
55,7 |
0,74 |
|
Х94979 |
mRNA for pollen coat protein |
(atg)5 |
55,5 |
0,44 |
|
AF241115 |
isolate HRI/CGN 5688 cauliflower gene |
(at)5(ta)6 |
52,6 |
0,51 |
|
X92955 |
mRNA for pollen coat protein |
(tttta)2(ata)7 |
54 |
0,57 |
|
AF180355 |
isolate B265 ABI1 protein (ABI1) gene |
(tc)16 |
55 |
0,3 |
|
AF113919 |
phospholipase D2 (PLD2) gene |
(at)6(gt)5 |
56 |
0,58 |
|
AF273844 |
thioredoxin-h-like protein 1 (THL1) mRNA |
(ctt)7 |
54,2 |
0,73 |
|
AF230693 |
stearoyl-ACP desaturase (DELTA9-BO-1) gene |
(ctt)3(ct)6(cttg)6 |
56 |
0,81 |
|
AF113918 |
phospholipase D1 (PLD1) gene |
(ct)7(at)7-1 |
56,4 |
0,6 |
Результаты и обсуждения
В результате ДНК-типирования рассмотрен 91 аллель в 16 SSR-локусах. Уровень наблюдаемого полиморфизма составил 85,7%. Количество рассмотренных аллелей на локус достигало 211 и в среднем составило 5,7 на один локус. Индекс информативности (PIC) анализируемых SSR-локусов варьировал от 0,25 (для CC969459) до 0,9 (для AF458409) (рис. 1) и в среднем достигал 0,63 (табл.).
Все оцененные локусы характеризовались высокой информативностью: 14 из 16 имели уровень PIC > 0,5. Полученные данные согласуются с результатами S. Louarn с коллегами (2007), которые использовали 11 пар «трансферабельных» SSR со сходным уровнем PIC для сортовой идентификации B. оleracea L.
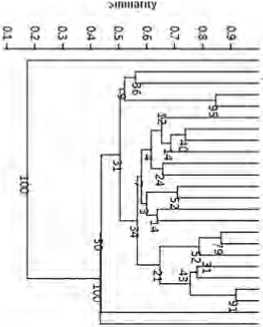
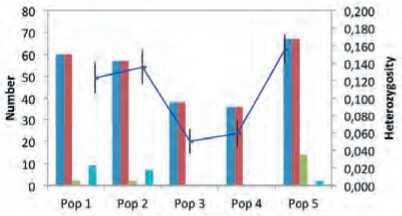
При изучении генетической гетерогенности капусты белокочанной на основе полиморфизма SSR локусов рассчитаны генетические дистанции (GD) по коэффициенту Джаккарта, между образцами была построена дендрограмма (рис. 2). Самую большую генетическую разницу наблюдали между разновидностями капусты, так наибольшая генетическая разница (31,5% генетической схожести) была между брокколи Тонус, кольраби Венская белая 1390 и брюссельской Геркулес 1342. Однако нужно отметить, что сорт брокколи, два образца капусты цветной и один сорт кольраби образовали общий подкластер с максимальной генетической схожестью 48,8%. В данном подкластере наиболее генетически близкими оказались сорта капусты цветной Ранняя грибовская и Полярная звезда со схожестью между собой 84,6%. Капуста брюссельская Геркулес 1342 оказалась генетически далеко от других разновидностей и образовывала отдельную ветвь дедро-граммы. Также капуста листовая декоративная Малиновка была генетически отдалена от остальных разновидностей и образовывала отдельную ветвь. Большой кластер кочанных капуст состоял из трех подкластеров: в первом – среднеспелые и скороспелые образцы капусты белокочанной, во втором – краснокочанные и савойские капусты, в третьем – позднеспелые и среднеспелые образцы капусты белокочанной. У кочанных разновидностей наиболее генетически близкими оказались сорта капусты белокочан- ной Зимовка и Подарок с генетической схожестью 86,5%. Максимальное генетическое расстояние в кластере кочанных капуст с генетической схожестью всего 44,7% было обнаружено между капустой белокочанной Слава 231 и капустой савойской Вертю 1340. Два сорта капусты краснокочанной: Гако 741 и Каменная головка были генетически сходны между собой на 71,1%, известно также, что эти сорта принадлежат к разным сор-тотипам. Два сорта капусты савойской: Московская кружевница и Вертю 1340 были сходны всего лишь на 63,9% и принадлежат к разным группам спелости. В то же время не было найдено уникальных локусов для савойских и краснокочанных капуст. На основании данных о распределении микросателлитных аллелей, приведенных на рисунке 3, где подкластеры дендрограммы соответствуют оцененным популяциям, можно cказать, что образцы капусты белокочанной показали высокую гетерозиготность, сравнимую среди разновидностей. Тем не менее, было выявлено только 3% и 3,5% редких аллелей у групп капусты белокочанной. Больше специфичных аллелей – 14 было обнаружено среди разновидностей капусты. Среди популяций капусты белокочанной общих аллелей было 9 и 7 соответственно.
Идентификация и характеристика генетических ресурсов имеет большое значение для отбора и типирования селекционных форм. Одним из наиболее эффективных инструментов оценки генетического полиморфизма являются микросателлиты, которые являются высокополиморфными маркерами для растительных геномов.
Высокое разнообразие экспериментальных форм, в основном у капусты белокочанной, свидетельствует о широкой генетической основе селекционной коллекции. Межсортовой полиморфизм, выраженный в аллельном разнообразии изученных SSR-локусов, в значительной мере облегчает сортовую идентификацию. Информация о топологической дифференциации коллекции капусты белокочанной, полученная в результате кластерного анализа, является основой для селекционного отбора генетически выровненного материала с использованием ДНК (т.е. маркер вспомогательной селекции).
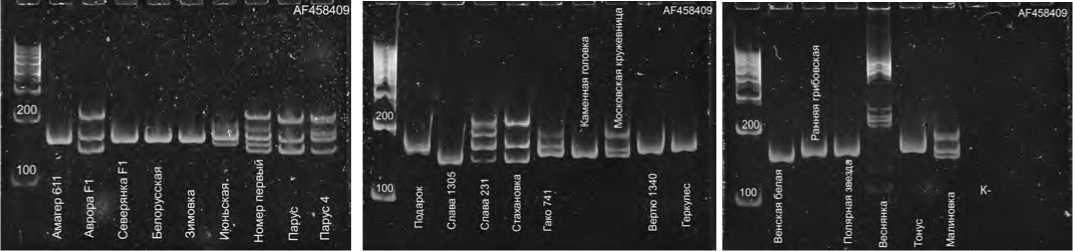
Рис. 1. Электрофореграммы образцов капусты B.oleracea L., полученные на основе амплификации локуса AF458409
Figure 1. Electrophoregram of cabbage accessions of B.oleracea L., obtained after amplification with locus AF458409
Веснянка
Венская белая 1390
Ранняя грибовская 1355 Полярная заезда Июньская 3200
Номер первый 147
Стахановка 1513
Аврора F1 Слава 1305 Спам 231 Гако 741
Каменная головка
Московская кружевница
Вертю 1340
Зимовка 1474
Псдаро*. 2500
АмагербП
Северяне F1 Белорусская 455
Малиновка
Геркулес 1342
Рис. 2. Результаты кластерного анализа вида B. oleracea L., полученного на основе полиморфизма 16 микросателлитных локусов. Figure 2. Results of cluster analysis in species B. oleracea L. obtained on the basis of polymorphism in 16 microsatellite loci.
No. Bands
■■No. Bands Freq. >= 5%
No. Private Bands
■■No. LComm Bands (<=25%)
■■ No. LComm Bands (<=50%)
---Mean He
Populations
Рис. 3. Распределение микросателлитных аллелей в популяциях B.oleracea L.
Pop 1 и Pop 2 – два подкластера капусты белокочанной, Pop 3 – подкластер капусты краснокочанной,
Pop 4 – подкластер капусты савойской, Pop 5 – другие разновидности.
Figure 3. Bands patterns across populations of B.oleracea L.
Pop 1 and Pop 2 – two subclusters of white head cabbage, Pop 3 – subcluster of red head cabbage,
Pop 4 – subcluster of Savoi cabbage, Pop 5 – other cabbage varieties.
Список литературы Классификация отечественных сортов капусты Brassica oleracea L. c использованием SSR маркеров
- Артемьева А.М., Клоке Э., Чесноков Ю.В. Анализ филогенетических связей вида Brassica oleracea L. (Капуста огородная)//Вестник ВОГиС. -2009. -Т. 13. -№ 4. -С. 759-771.
- Шаптуренко М.Н., Печковская Т.В., Вакула С.И., Якимович А.В., Забара Ю.М., Хотылева Л.В. Информативные EST-SSR-маркеры для типирования и внутривидовой дифференциации Brassica oleracea var. capitata L.//Вавиловский журнал генетики и селекции. -2016. -Т. 20. -№ 1. -С. 51-56. 10.18699/VJ16.133.
- Formisano G., Roig C., Esteras C., Ercolano M.R., Nuez F., Monforte A.J., Picof M.B. Genetic diversity of Spanish Cucurbita pepo landraces: an unexploited resource for summer squash breeding//Genet. Resour. Crop Evol. 2012. V. 59, N6. P. 1169-1184.
- Izzah N.K., Lee J., Perumal, S., Park J.Y., Ahn K., Fu D., Kim G-B., Nam Y-W., Yang T-J. Microsatellite-based analysis of genetic diversity in 91 commercial Brassica oleracea L. cultivars belonging to six varietal groups//Genetic Resources and Crop Evolution. 2013. V.60. 10.1007/s10722-013-9966-3.
- Jaccard P. Nouvelle recherches sur la distribution florale//Bull. Soc. Vandoise Sci. Nat. 1908. V.44, P. 223-270.
- Kumar A., Mishra P., Singh S.C., Sundaresan V. Efficiency of ISSR and RAPD markers in genetic divergence analysis and conservation management of Justicia adhatoda L., a medicinal plant//Plant Syst. Evol. -2014. -V. 300: 1409-1420. 10.1007/s00606-013-0970-z.
- Louarn S., Torp A.M., Holme I.B., Andersen S.B., Jensen B.D. Database derived microsatellite markers (SSRs) for cultivar differentiation in Brassica oleracea.//Genet. Resour. Crop. Evol. 2007. V. 54, P. 1717-1725.
- Lowe, A.J., C. Moule, M. Trick, and K.J. Edwards. Efficient large-scale development of microsatellites for marker and mapping applications in Brassica crop species//Theor. Appl. Genet. 2004. V. 108, P. 1103-1112.
- Paterson A.H., Lan T-h, Amasino R., Osborn T.C. Quiros C Brassica genomics: a complement to, and early beneficiary of the Arabidopsis sequence. Genome Biology 2001. V. 2, P. 1011. 1011-1011.1014.
- Powel W., Machray G.C., Povan J. Polymorphism revealed by simple sequence repeats//Trends Plant Sci. 1996. V. 1, P. 215-222.
- Tonguc M., Griffiths P.D. Genetic relationships of Brassica vegetables determined using database derived sequence repeats//Euphytica 2004. V. 137: 193-201.


