Компьютерное моделирование в разработке вакцин против COVID-19 на основе антигенов HLA-системы
Автор: Вологжанин Дмитрий Александрович, Голота Александр Сергеевич, Камилова Татьяна Аскаровна, Шнейдер Ольга Вадимовна, Щербак Сергей Григорьевич
Журнал: Клиническая практика @clinpractice
Рубрика: Обзоры
Статья в выпуске: 3 т.12, 2021 года.
Бесплатный доступ
Генетическая вариабельность популяции может объяснить различные индивидуальные иммунные реакции на вирус SARS-CoV-2. Использование технологий на основе анализа генома и протеома дает возможность разрабатывать вакцины путем оптимизации выбора антигенов-мишеней. Методология компьютерного моделирования предоставляет научному сообществу более полный список иммуногенных пептидов, включающий в себя ряд новых и перекрестно-реактивных кандидатов. Исследования, проводимые независимо друг от друга с различными подходами, дают высокую степень уверенности в воспроизводимости результатов. Бόльшая часть усилий по разработке вакцин и лекарств против SARS-CoV-2 направлена на гликопротеин шипа (белок S) - главный индуктор нейтрализующих антител. Несколько вакцин продемонстрировали эффективность в доклинических исследованиях и прошли клинические испытания по противодействию инфекции COVID-19. В обзоре представлен профиль предсказанных in silico[1] иммуногенных пептидов вируса SARS-CoV-2 для последующей функциональной валидации и разработки вакцин; освещаются текущие достижения в разработке субъединичных вакцин для борьбы с COVID-19 с учетом опыта, который был достигнут ранее с SARS-CoV и MERS-CoV. Методы иммуноинформатики сокращают время и затраты при разработке вакцин, которые вместе могут остановить эту новую вирусную инфекцию. [1] Термин, обозначающий компьютерное моделирование (симуляцию) эксперимента, чаще биологического.
Коронавирус, sars-cov-2, covid-19, иммуногенный пептид, антиген, hla, вакцина, эпитоп, компьютерное прогнозирование, компьютерное моделирование in silico, иммуноинформатика
Короткий адрес: https://sciup.org/143178081
IDR: 143178081 | DOI: 10.17816/clinpract76291
Текст обзорной статьи Компьютерное моделирование в разработке вакцин против COVID-19 на основе антигенов HLA-системы
В декабре 2019 года в городе Ухань китайской провинции Хубэй было одновременно зарегистрировано несколько тяжелых случаев пневмонии неизвестной этиологии. Вскоре после этого новый бета-коронавирус из семейства Coronaviridae рода Betacoronavirus отряда Nidovirales был идентифицирован как возбудитель тяжелого острого респираторного заболевания. У людей бета-коронавирусы могут вызывать легкие респираторные инфекции («простуда»), а также гораздо более серьезные заболевания (SARS и MERS).
11 февраля 2020 г. Всемирная организация здравоохранения присвоила новой коронавирусной инфекции официальное название — COVID-19 (CОronaVIrus Disease 2019), а Международный комитет по таксономии вирусов (International Committee of Taxonomy of Viruses, ICTV) — официальное на- звание возбудителю этой инфекции — SARS-CoV-2 (severe acute respiratory syndrome coronavirus 2) [1].
11 марта 2020 г. Всемирная организация здравоохранения объявила распространение коронавируса SARS-COV-2, вызывающего болезнь COVID-19, глобальной пандемией. Различные штаммы SARS-CoV-2 с секвенированным геномом имеют более 99,98% идентичности последовательности, и они близкородственны (88% идентичности) SARS-подобным коронавирусам летучих мышей, в то время как более отдалены от SARS-CoV и MERS-CoV человека [2].
У одних индивидов, зараженных коронавирусом SARS-CoV-2, развивается только легкая или бессимптомная форма COVID-19. Другие страдают тяжелой пневмонией с острым респираторным дистресс-синдромом и высокой летальностью. Согласно результатам анализа клинических
КТ ГА
COMPUTER SIMULATION IN THE DEVELOPMENT OF VACCINES
AGAINST COVID-19 BASED ON THE HLA-SYSTEM ANTIGENS
D.A. Vologzhanin1, 2, A.S. Golota1, T.A. Kamilova1, O.V. Shneider1, S.G. Sсherbak1, 2
-
1 Saint-Petersburg City Hospital No 40 of Kurortny District, Saint Petersburg, Russian Federation
-
2 Saint-Petersburg State University, Saint Petersburg, Russian Federation
The genetic variability of population may explain different individual immune responses to the SARS-CoV-2 virus. The use of genome- and peptidome-based technologies makes it possible to develop vaccines by optimizing the target antigens. The computer modeling methodology provides the scientific community with a more complete list of immunogenic peptides, including a number of new and cross-reactive candidates. Studies conducted independently of each other with different approaches provide a high degree of confidence in the reproducibility of results. Most of the effort in developing vaccines and drugs against SARS-CoV-2 is directed towards the thorn glycoprotein (protein S), a major inducer of neutralizing antibodies. Several vaccines have been shown to be effective in the preclinical studies and have been tested in the clinical trials to combat the COVID-19 infection. This review presents the profile of in silico predicted immunogenic peptides of the SARS-CoV-2 virus for the subsequent functional validation and vaccine development, and highlights the current advances in the development of subunit vaccines to combat COVID-19, taking into account the experience that has been previously achieved with SARS-CoV and MERS-CoV. The immunoinformatics techniques reduce the time and cost of developing vaccines that together can stop this new viral infection.
Submitted 20.07.2021
Revised 13.08.2021
Published 26.08.2021
характеристик пациентов из 73 научных статей, наиболее распространенные симптомы начала заболевания — повышение температуры (82,1%), кашель (45,8%), слабость (26,3%), головная боль (6,5%) и одышка (6,9%); из них тяжелых случаев с одышкой — 32,6%) [3]. Близкие по значению данные о клинических проявлениях COVID-19 получены в ретроспективном исследовании в Уханьской больнице Цзиньинтань [4] и в России [5].
Коронавирус SARS-CoV-2 обладает тропизмом к тканям легкого (альвеолоцитам, альвеолярным эпителиоцитам, легочным капиллярам, лимфоидной ткани и макрофагам легкого), вызывая вне-больничную пневмонию. У некоторых пациентов с пневмонией внезапно развивается тяжелая дыхательная недостаточность, требующая респираторной поддержки. Риск смерти этих пациентов достигает 60% [6]. У большинства пациентов с тяжелой дыхательной недостаточностью преобладает иммунная дисфункция с лимфопенией, чрезмерной секрецией провоспалительных цитокинов и белков острой фазы воспаления [7]. Результаты компьютерного моделирования (в экспериментах in silico) показывают, что существуют генетические различия, особенно в иммунной системе человека, которые могут объяснить различную способность реагировать на инфекцию SARS-CoV-2, различия в симптоматике и степени тяжести COVID-19.
Когда коронавирус инфицирует клетки человека, организм реагирует антивирусным сигналин-гом. Чтобы выяснить, могут ли разные аллели сигнальной системы объяснить диапазон иммунных ответов на SARS-CoV-2, многочисленные группы исследователей используют компьютерные алгоритмы для анализа всех белков коронавируса, чтобы предсказать, насколько хорошо различные версии антивирусной сигнальной системы обнаруживают вирусные белки. В частности, HLA-типирование предоставляет информацию о роли гаплотипа HLA (human leukocyte antigens) в вирусной инфекции для персонализированной профилактики, диагностики и лечения [3].
Ю1иническаяьп91
Том 122 №13
Результаты компьютерного моделирования предсказывают, что некоторые аллели HLA-антигенов связываются с большим количеством пептидов из белков вируса SARS-CoV-2, тогда как другие связываются с очень немногими [8], т.е. некоторые варианты HLA имеют большее сродство к SARS-CoV-2, чем другие, и, следовательно, специфические аллели HLA будут влиять на эффективность иммунного ответа при COVID-19. На этом основании достигнут консенсус о том, что различия в генах HLA являются частью объяснения широкого диапазона различий в степени тяжести COVID-19.
БИОЛОГИЯ И ГЕНЕТИКА SARS-COV-2
Геном SARS-CoV-2, как и геномы других коронавирусов, кодирует ряд структурных и неструктурных белков. Он состоит из одноцепочечной позитивносмысловой РНК длиной ~30 000 нуклеотидов и содержит две фланкирующие нетранслируемые области и одну длинную открытую рамку считывания (open reading frame, ORF), которая кодирует полипротеин, включающий в себя репликазный комплекс ORF1ab, и гены четырех структурных белков — гликопротеина шипа (S), гликопротеина мембраны (M), белка оболочки (E) и нуклеокапсидного фосфопротеина (N). Последовательность ORF1ab (>65% всей длины генома) кодирует 16 неструктурных белков. S-белок вируса SARS-CoV-2 обладает структурой, оптимальной для связывания с клеточным рецептором ACE2 (angiotensin-converting enzyme) [3, 9].
Все реплицирующиеся вирусы, включая коронавирус, накапливают мутации, которые сохраняются благодаря естественному отбору и способствуют уклонению от иммунитета. Исследование мутаций в 6420 последовательностях генома SARS-CoV-2, изолированных у пациентов из Азии, Северной Америки, Европы и Океании, идентифицировало в общей сложности 156 миссенс-мутаций, которые наблюдались с частотой >0,5%. Мутации OR-F1ab P4715L и S-белка D614G встречаются во всех четырех регионах, хотя в азиатских странах значительно реже, чем в других (15,4 против 52,6–73,7%; р=3,81х10-125). Мутации ORFIab P5828L и ORFIab Y5865C преобладают в Северной Америке (30,6 против 0-7,8%; р=3,26х10-237, и 31,4 против 0-8,1% в других регионах; р=5,54х10"246, соответственно). Мутация N-белка R203K/G204R встречается чаще других мутаций в Океании и Европе (14,7–27,6 против 3,6-5,3%; р=2,56х10"105) [9]. В исследовании по моделированию гомологии протестированы трансмембранные спиральные сегменты ORF1ab в неструктурных белках nsp2 и nsp3 (nonstructural protein) коронавируса. Результаты показали, что как стабилизирующая, так и дестабилизирующая мутации эндосомально-ассоциированного домена белков nsp2 и nsp3 могут объяснить высокую контагиозную способность SARS-CoV-2 [3].
Два белка SARS-CoV-2 — ORF-3a и ORF-7a — являются предполагаемыми детерминантами T-клеточного распознавания. Оба белка важны для репликации вируса и могут влиять на патогенез и распространение заболевания. Наибольшее снижение экспансии вируса отмечено после удаления белка ORF3a [10].
ЦИТОКИНОВЫЙ ШТОРМ ПРИ COVID-19
Как уже отмечалось выше, у пациентов с крайне тяжелым COVID-19 наблюдается лимфопения характеризующаяся прогрессивным снижением абсолютного и относительного числа CD4+, CD8+ и других субпопуляций лимфоцитов, при этом экспрессия маркеров T-клеточной активации — HLA-DR, CD45RO и PD-1 — увеличена у пациентов с тяжелой и особенно с крайне тяжелой формой по сравнению с пациентами с легкой формой заболевания. Уровни цитокинов IL-2, TNF-a, IL-6, IL-10, а также C-реактивного белка в плазме крови повышены, в то время как активация дендритных клеток (ДК) и B-клеток снижена у пациентов с критически тяжелой формой заболевания. Хотя процентное содержание IFN—Y—продуцирующих T-клеток CD4+ и CD8+ у пациентов с тяжелой и крайне тяжелой формой COVID-19 намного выше, чем у пациентов с COVID-19 легкой и средней тяжести, абсолютное их количество не различалось между этими тремя группами. Эти данные свидетельствуют о том, что патогенез крайне тяжелого COVID-19 обусловлен гиперфункцией T-клеток CD4+ и CD8+ [11].
На ранних стадиях заболевания миграция лимфоцитов из крови в легкие может быть причиной дефицита лимфоцитов в периферической крови, что обусловлено антигенной стимуляцией. Экспрессия маркера активации HLA-DR повышена, а костимулирующей молекулы CD28 — снижена у пациентов с тяжелым заболеванием, что предполагает активацию Т-клеток у пациентов с тяжелым течением заболевания. Гиперфункция T-клеток CD4+ инициирует синдром активации макрофагов, который приводит к цитокиновому шторму у пациентов с крайне тяжелым COVID-19. Непрерывные и избыточные воспалительные реакции в конечном

кт ГА
итоге вызывают апоптоз лимфоцитов, на более поздних стадиях инфекции наблюдается анергия лимфоцитов. Таким образом, функция лимфоцитов может быть совершенно разной на разных стадиях инфекции [11, 12].
Молекулы HLA-класса II, особенно HLA-DR, экспрессируются конститутивно главным образом антигенпрезентирующими клетками, В-клетками и некоторыми активированными T-клетками. Их экспрессия необходима для запуска адаптивного иммунного ответа и клиренса патогена. IFN-γ индуцирует экспрессию генов HLA-DR и воспалительных цитокинов TNF-α, IL-1 β и IL-6. Активация HLA класса II увеличивает HLA-рестриктированную антигенную презентацию и адаптивный иммунный ответ.
Вирусные инфекции, в том числе коронавирусы, способны индуцировать большое количество про-воспалительных цитокинов у предрасположенного хозяина, что в конечном итоге может вызвать ги-перферритинемический (гемофагоцитарный) синдром. Усиление секреции цитокинов лимфоцитами Th1 и Th17 формирует провоспалительную среду со сниженными уровнями противовоспалительных цитокинов (IL-4, IL-10, TGF-β) и последующим уменьшением количества общих и функциональных регуляторных Т-клеток (Treg CD4+, FoxP3+) и, соответственно, ограничением контроля аутотолерантности. Это связано с ролью клеток Treg в определении степени тяжести COVID-19. В тяжелых случаях наблюдается снижение пропорции наивных Treg, но повышение Treg-памяти, которые в свою очередь могут играть роль в формировании высокоинтенсивного аутоиммунного ответа с дисбалансом цитокинов и гиперэкспрессией молекул HLA-DR [12].
АНТИГЕНЫ HLA И SARS-COV-2
Антигенная презентацияпри коронавирусной инфекции
Локусы системы HLA являются одним из важных факторов генетической предрасположенности человека к инфекционным заболеваниям. Различные гаплотипы HLA связаны с различной подверженностью болезням, главным образом благодаря рецепторам антигенов Т-клеток, которые распознают конформационную структуру антигенсвя-зывающего домена молекулы HLA в комплексе с соответствующими антигенными пептидами. Соответственно, представляется преимуществом иметь молекулы HLA с повышенной специфично- стью связывания с пептидами вируса SARS-CoV-2 на поверхности антигенпрезентирующих и других иммунных клеток. Необходимо идентифицировать специфические аллели HLA класса I или II, связанные с развитием иммунитета против SARS-CoV-2. Такая информация важна для разработки тест-систем и оценки эффективности вакцинации у разных людей в общей популяции [3, 13].
Когда вирус проникает в клетку, его антигены будут представлены HLA-системой антигенпрезентирующих клеток и затем распознаются ви-рус-специфичными цитотоксическими Т-лимфоцитами (ЦТЛ). Например, презентация антигенов SARS-CoV в основном зависит от молекул HLA класса I, но HLA-II также участвуют в их презентации. Многочисленные полиморфизмы HLA-антигенов коррелируют с восприимчивостью к SARS-CoV, такие как HLA-B*46:01, HLA-B*07:03, HLA-DRB1*12:02 и HLA-Cw*08*01, тогда как аллели HLA-DR*03:01, HLA-Cw*15:02 и HLA-A*02:01 связаны с защитой от инфекции SARS-CoV [14]. Молекулы HLA-II, такие как HLA-DRB1*11:01 и HLA-DQB1*02:02, ассоциированы с восприимчивостью к инфекции MERS-CoV. Несмотря на то, что количество T-клеток CD4+ и CD8+ в периферической крови пациентов, инфицированных SARS-CoV-2, значительно снижено, в то время как их иммунный статус представляет собой чрезмерную активацию, о чем свидетельствует высокая доля фракций HLA-DR-позитивных Т-клеток CD4+ (3,47%) и Т-клеток CD38+ CD8+ (39,4%) [14].
Посредством комплексного биоинформаци-онного скрининга эпитопов из последовательностей SARS-CoV-2 для HLA идентифицированы 2013 и 1399 потенциальных пептидных эпитопов с высокой аффинностью к молекулам HLA класса I и II со- ответственно, которые могут индуцировать ответы Т-клеток CD8+ и CD4+. Эти эпитопы распределены по структурным (белки шипа, оболочки, мембраны и нуклеокапсида) и неструктурным белкам (соответствующим шести открытым рамкам считывания); при этом обнаружены несколько областей, обогащенных высокоаффинными эпитопами. Результаты исследования дают ценную информацию для разработки вакцин против SARS-CoV-2 и мониторинга Т-клеточных ответов [9].
Лимфопения заметно уменьшается по мере улучшения состояния [15]. У умерших пациентов с COVID-19 было значительно снижено количество CD4+ и CD8+ T-клеток, но они были гиперактиви-рованы [16]. Как теперь известно, гиперактивиро-
<линическая 2021 п эакти keu Том 12 №3
ванные иммунные клетки вовлечены в иммунное повреждение тканей и органов при COVID-19.
S. Zhang и соавт. [17] изучили динамику иммунологических показателей у 55-летнего пациента с критически тяжелой COVID-19. От критической стадии COVID-19 до стадии реконвалесценции низкие уровни T-клеток CD4+ и CD8+ постепенно повысились до нормальных уровней, когда определение вирусной РНК дало отрицательный результат.
Сигнальный путь HLA-G и его рецептора, экспрессируемого на поверхности иммунных клеток, негативно регулирует функции Т-, В- и NK-клеток и участвует в вирусной инфекции. Считается, что усиление экспрессии HLA-G является стратегией уклонения вируса от иммунитета. Основной механизм уклонения заключается в том, что HLA-G связывает иммуноингибирующие рецепторы цитотоксических иммунных клеток. Анализируя динамику периферических иммунных клеток, S. Zhang и соавт. [17] сравнили экспрессию HLA-G и его рецепторов на периферических иммунных клетках в день, когда был подтвержден положительный результат теста на РНК SARS-CoV-2, и в день, когда результат стал отрицательным. Анализ показал положительную связь экспрессии HLA-G на В-клетках с уровнями IFN-γ, экспрессия HLA-G на моноцитах негативно связана с уровнями IL-2. Процент Т-клеток HLA-G+ и экспрессия иммуноингибирующего рецептора ILT4 на B-клетках негативно коррелируют с уровнями TNF-α, на моноцитах — положительно коррелирует с уровнями цитокинов IL-6, IL-10 и IFN-γ. Паттерн экспрессии HLA-G на периферических иммунных клетках может отражать три стадии течения заболевания — инфицирования, репликации и клиренса SARS-CoV-2. При этом наблюдались различные корреляции между экспрессией HLA-G на В-клетках и моноцитах и продукцией IFN-γ и IL-2. Динамика экспрессии HLA-G на клетках периферических иммунных субпопуляций (высокий–низкий–высокий уровни) от SARS-CoV-2-позитивного статуса до SARS-CoV-2-негативного указывает на то, что статус инфекции SARS-CoV-2 связан с регуляцией экспрессии HLA-G цитокинами. Учитывая, что HLA-G является антигенпрезентирующей молекулой, подавление вирусом SARS-CoV-2 экспрессии HLA-G может нарушать распознавание вируса Т-клетками CD8+ и поддерживать уклонение от иммунитета [17].
Система HLA играет ключевую роль в иммунном ответе на патогены. Так, 82 пациента с COVID-19 были генотипированы по локусам HLA-A, HLA-B,
HLA-C, HLA-DRB1, HLA-DRB3/4/5, HLA-DQA1, HLA-DQB1, HLA-DPA1 и HLA-DPB1 с использованием технологии NGS (Next Generation Sequencing). Частоты аллелей HLA-C*07:29, C*08:01G, B*15:27, B*40:06, DRB1*04:06 и DPB1*36:01 повышены, в то время как частоты аллелей DRB1*12:02 и DPB1*04:01 снижены у пациентов с COVID-19 по сравнению с контрольной популяцией. При рассмотрении скорректированной статистической значимости различались частоты только HLA-C*07:29 и HLA-B*15:27. Эти данные свидетельствуют о том, что некоторые аллели HLA ассоциированы с развитием COVID-19 [18].
РАЗРАБОТКА ВАКЦИН ПРОТИВ SARS-COV-2
Для разработки вакцины против SARS-CoV-2 и оценки иммуногенности вакцин-кандидатов важно прогнозировать антигенные детерминанты SARS-CoV-2 и иммунные ответы на них.
C помощью прикладного математического анализа идентифицированы Т-клеточные эпитопы, способные вызывать стойкий иммунный ответ против SARS-CoV-2 в глобальной популяции людей. Прогнозирование В-клеточных эпитопов (как линейных, так и конформационных) считается ненадежным по сравнению с Т-клеточными эпитопами. Кроме того, B-клеточные эпитопы не вызывают сильного гуморального ответа. По этой причине в большинстве исследований анализировали in silico только эффективные Т-клеточные эпитопы, способные генерировать длительный ответ Т-клеток CD4+ и CD8+, чтобы выбрать среди них наиболее подходящие и достоверные эпитопы, которые можно в дальнейшем тестировать в «мокрой» лаборатории [10].
При вирусной инфекции белки вируса экспрессируются в инфицированных клетках и процессируются протеасомами в небольшие пептиды, которые презентируются молекулами HLA на поверхности инфицированных клеток и распознаются Т-клетка-ми посредством Т-клеточных рецепторов. Эпитопы, потенциально распознаваемые Т-клетками, могут быть получены из любых вирусных структурных и неструктурных белков. К. Kiyotani и соавт. [9] осуществили иммуноинформатический прогноз эпитопов белков S, E, N, M и ORF (соответствующих регистрационным номерам QHD43415-QHD43423, QHI42199) эталонного образца SARS-CoV-2_Wu-han-Hu-1 (регистрационный номер MN908947). Чтобы предсказать эпитопы HLA класса I, отобрали 7 аллелей HLA-A, 10 аллелей HLA-B и 8 аллелей
КТ ГА
HLA-C человека, которые присутствуют в японской популяции с частотой более 5%. Для предсказания эпитопов HLA класса II выбраны 5 гаплотипов HLA-DPA1-DPB1, 6 гаплотипов HLA-DQA1-DQB1 и 7 аллелей HLA-DRB1, которые встречаются в популяциях Японии с частотой 5–38%. Полноразмерные вирусные нуклеотидные последовательности SARS-CoV-2 (идентификационные номера в базе данных MN908947 и MN996527–MN996531), SARS-CoV (номера в базе данных AY274119, AY278488 и AY390556), SARS-подобного коронавируса летучей мыши (bat-SL-CoV) RaTG13 (номер MN996532) и MERS-CoV (номер JX869059) предоставляет NCBI GenBank [9].
Для выявления мутаций SARS-CoV-2 использовали 587 геномов, изолированных в Азии, 1918 — в Северной Америке, 3190 — в Европе и 726 — в Океании, депонированных в базе данных Global Initiative on Sharing Avian Influenza Data с 18 апреля 2020 года. Каждую из этих последовательностей SARS-CoV-2 выровняли относительно эталонной последовательности SARS-CoV-2_Wuhan-Hu-1, затем экстрагировали нуклеотидные последовательности, соответствующие отдельным белкам SARS-CoV-2, транслировали их в аминокислотные последовательности, которые сравнили с эталон- ными аминокислотными последовательностями SARS-CoV-2 Wuhan-Hu-1.
Аффинность связывания с молекулами HLA класса I рассчитана для всех 9- и 10-мерных пептидов из белков SARS-CoV-2 с использованием программ NetHLAv4.0 и NetMHРПanv4.0. Выбраны 0,5%-ные частотные эпитопы на основе прогноза наиболее сильного связывания. Аффинность связывания с молекулами HLA класса II рассчитывали для всех 15-мерных пептидов из белков SARS-CoV-2 с использованием программного обеспечения NetHLAIIpanv3.1. В качестве порога связывания эпитопов применен 2%-ный уровень.
Следующим этапом был скрининг эпитопов с вероятностью быть представленными на молекулах HLA класса I и II. Среди высокоаффинных пептидов из белков SARS-CoV-2 получены в общей сложности 2013 уникальных предсказанных эпитопов белков М и N для HLA класса I и 1399 эпитопов белков М, N, ORF3a и ORF6 для HLA класса II с высокой аффинностью к HLA-DPA1, DPB1, DQA1, DQB1 и DRB1.
Поскольку SARS-CoV-2 имеет 79%- и 50%-ную гомологию нуклеотидных последовательностей с вирусами SARS-CoV и MERS-CoV соответствен- но, предсказанные эпитопы SARS-CoV-2 сравнили с последовательностями SARS-CoV (BJ01, GZ02 и Tor2) и MERS-CoV для оценки их перекрестной реактивности. Обнаружено, что 780 (38,8%) эпитопов HLA класса I являются консервативными во всех трех последовательностях SARS-CoV, из них 633 (81,0%) расположены в белке ORF1ab, а 58 (7,4%), 15 (1,9%), 28 (3,6%) и 33 (4,2%) пептида находятся в белках S, E, M и N соответственно. При этом 36 (1,8%) эпитопов HLA класса I демонстрируют 100%-ную идентичность последовательности с пептидными последовательностями MERS-CoV, из них 33 расположены в белке ORF1ab и 3 — в белке S; 30 эпитопов в белке ORF1ab являются общими для SARS-CoV и MERS-CoV. Среди 1399 возможных эпитопов HLA-II 418 (29,9%) демонстрируют 100%-ную идентичность последовательности для всех трех коронавирусов; 362 (86,7%), 40 (11,0%), 4 (1,1%), 4 (1,1%) и 7 (1,9%) расположены в белках ORF1ab, S, E, M и N соответственно; 10 (2,4%) эпитопов (все в белке ORF1ab) также имеются у MERS-CoV.
Т-клеточные эпитопы, которые с наибольшей вероятностью представлены молекулами HLA, могут присутствовать у значительной части ин-дивидов/пациентов. Два эпитопа в белке OR-F1ab — ORF1ab2168-2176 и ORF1ab4089-4098, ко- торые, как предсказано, имеют сильное сродство к HLA-A*24:02, HLA-A*02:01 и HLA-A*02:06, показали самый широкий охват (83,8%) населения Японии. ORF1ab2168-2176, предсказанный как эпитоп, связывающий молекулы HLA-C (C*01:02, C*08:01, C*12: 02 и C*14:02), имеется у 76,5% японцев, эпитопы S268-277 и S448-457 в белке S — у 70% японцев. HLA-олигомеры с этими пептидами полезны для мониторинга ответов Т-клеток CD8+ у пациентов и бессимптомно инфицированных индивидов. Мутации в последовательностях описанных эпитопов не обнаружены [9].
Все пациенты с тяжелой дыхательной недостаточностью имели либо синдром активации макрофагов (macrophage activation syndrome, MAS), либо очень низкую экспрессию HLA-DR, сопровождающуюся глубоким истощением Т-лимфоцитов CD4+ и естественных клеток-киллеров (natural killer cells, NK). Усиленная продукция TNF-α и IL-6 циркулирующими моноцитами отличает COVID-19 от бактериального сепсиса или гриппа. Плазма пациентов с SARS-CoV-2 ингибирует экспрессию HLA-DR, которую частично восстанавливает блокатор IL-6 тоцилизумаб; лечение пациентов тоцилизумабом сопровождалось увеличением числа циркулирую- и ни не с кая ь™
пэактика -21
щих лимфоцитов. Таким образом, уникальный паттерн иммунной дисрегуляции при тяжелом течении COVID-19 характеризуется IL-6-опосредованным подавлением экспрессии HLA-DR и лимфопенией, связанной со стойким усилением продукции цитокинов и гипервоспалением [7].
Состояние иммунной системы при COVID-19 характеризуется значительным снижением количества молекул HLA-DR на моноцитах CD14. Хотя у пациентов с бактериальной пневмонией также снижена экспрессия HLA-DR на моноцитах CD14, их уровни циркулирующего ферритина значительно выше нормы. Эта особенность обнаружена только у пациентов с SARS-CoV-2 и синдромом активации макрофагов. Значения гемофагоцитоза более 169 являются высокодиагностическими. У пациентов с бактериальной пневмонией с промежуточным иммунным состоянием количество молекул HLA-DR на моноцитах CD14 было ниже нормы. Однако пациенты с пневмонией, вызванной SARS-CoV-2, сохраняли количество молекул HLA-DR на моноцитах CD14 гораздо ближе к здоровому состоянию. Когда оно внезапно упало, развилась тяжелая дыхательная недостаточность. Более того, абсолютные показатели нейтрофилов и моноцитов были выше у пациентов с нарушениями иммунной регуляции, чем у пациентов с MAS. Таким образом, значительное снижение HLA-DR на моноцитах CD14 связано с тяжелой дыхательной недостаточностью.
У всех пациентов, инфицированных SARS-CoV-2, циркулирующие концентрации IFN-γ ниже предела обнаружения, то есть иммунные ответы Th1-типа не способствуют чрезмерному воспалению. Напротив, концентрации IL-6 и C-реактивного белка значительно выше у пациентов с нарушенной регуляцией иммунитета, чем у пациентов с промежуточным состоянием иммунной активации. Известно, что IL-6 ингибирует экспрессию HLA-DR. В соответствии с этим обнаружена негативная корреляция между сывороточными уровнями IL-6 и абсолютным числом молекул HLA-DR на моноцитах CD14, а также между абсолютным числом лимфоцитов и абсолютным количеством молекул HLA-DR на моноцитах CD14 у пациентов с COVID-19 [7].
Роль IL-6 как фактора снижения HLA-DR на моноцитах CD14 подтверждается увеличением числа циркулирующих клеток HLA-DR+ во время выздоровления от COVID-19 [19]. Интересно, что блокада IL-6 с тоцилизумабом частично восстанавливает экспрессию HLA-DR на моноцитах CD14 и увеличивает количество циркулирующих лимфоцитов [20].
Уникальный паттерн нарушения регуляции иммунитета у пациентов с SARS-CoV-2 характеризуется, с одной стороны, повышением циркулирующих уровней провоспалительных цитокинов (особенно IL-6), с другой стороны — дефектом лимфоидной функции, связанным с IL-6-опосредованным снижением экспрессии HLA-DR [7]. Эти результаты подтверждают обоснованность начатых клинических испытаний эффективности анакинры, сари-лумаба, сильтуксимаба и тоцилизумаба (Clinicaltri-als.gov NCT04330638, NCT04317092, NCT04315298 и EudraCT № 2020-001039-29) для индукции противовоспалительных ответов у этих пациентов.
Комплексный анализ с помощью компьютерного моделирования ( in silico ) аффинности связывания пептид–HLA класса I для 145 генотипов HLA-A, -B и -C и полного протеома вируса SARS-CoV-2, а также перекрестного защитного иммунитета, возникающего в результате предварительного воздействия четырех распространенных коронавирусов человека, обнаружил, что антиген HLA-B*46:01 связывает наименьшее число предсказанных пептидов SARS-CoV-2. Это позволяет предположить, что индивиды с этим аллелем могут быть особенно уязвимы к COVID-19, как это ранее было показано относительно SARS-CoV, что соответствует клиническим данным, связывающим этот аллель с тяжелым заболеванием. И, наоборот, аллель HLA-B*15:03 продемонстрировал наибольшую способность представлять высококонсервативные пептиды SARS-CoV-2, которые являются общими для коронавирусов человека [8]. По мнению авторов, это означает, что он может обеспечить перекрестный Т-клеточный иммунитет, и, следовательно, отдельные генотипы HLA могут дифференциально индуцировать Т-клеточный противовирусный ответ, влияя на течение заболевания и его трансмиссию.
Среди 48 395 уникальных 8–12-мерных пептидов из протеома SARS-CoV-2 из дальнейшего рассмотрения исключили 16 138, относительно которых не было предсказано, что они пройдут путь процессинга через протеасомальное расщепление. Для оставшихся 32 257 пептидов показано SARS-CoV-2-специфичное распределение презентации молекулами HLA класса I, главными представителями которых являются аллели HLA-A*02:02, HLA-B*15:03 и HLA-C*12:03 (прогнозируемый порог аффинности связывания <500 нМ). Важно отметить, что предполагаемая способность пептидов SARS-CoV-2 к антигенной презентации не связана с частотой

кт ГА
аллелей HLA в популяции. Представляя глобальные карты частоты 145 изученных аллелей HLA, авторы особо выделяют глобальные распределения трех лучших (A*02:02, B*15:03, C*12:03) и трех худших (A*25:01, B*46:01, C*01:02) презентирующих HLA-аллелей по способности генерировать репертуар эпитопов SARS-CoV-2 для поддержки Т-кле-точного иммунного ответа. Эти различия остаются значимыми на уровне гаплотипов, с большой вариабельностью презентации среди различных гаплотипов. Хотя исследован только ограниченный набор из 145 хорошо изученных аллелей HLA, этот анализ может быть выполнен для более широкого разнообразия генотипов. Генотипическая гетерогенность и эволюция SARS-CoV-2 in vivo могут модифицировать репертуар презентированных вирусных эпитопов или модулировать вирулентность иным, HLA-независимым образом. Тем не менее авторы рекомендуют включить HLA-типирование в клинические испытания и объединить с тестированием COVID-19 для применения потенциального предиктора (предикторов) тяжести заболевания среди населения и, возможно, для адаптации будущих стратегий вакцинации к генотипическим группам риска.
А. Joshi и соавт. [10] также предприняли попытку разработать вакцину против SARS-COV-2 на основе эпитопа путем анализа протеома вируса с использованием средств иммуноинформатики. Выбраны антигенные нетоксичные неаллергенные пептиды из неаллергенных белков на основе их взаимодействия с наборами HLA-аллелей (табл. 1). Среди идентифицированных ими Т-клеточных эпитопов в результате изучения белковых последовательностей SARS-COV-2 кандидатом на вакцину против этого вируса является эпитоп ITLCFTLKR с лучшими показателями связывания в предполагаемых комплексах эпитоп–HLA и приемлемыми показателями стабильности (предполагаемый период полураспада этого пептида — 20 ч в ретикулоцитах млекопитающих), токсичности и охвата населения, который должен теперь пройти лабораторную верификацию [10].
Для получения иммуногенных эпитопов чаще всего анализируют гликопротеин шипа (белок S), который с высокой аффинностью связывается с рецептором ACE2 человека для проникновения в клетки. На этом основании гликопротеин шипа считается подходящей мишенью вакцины против коронавирусов. Работа М. Bhattacharya и соавт. [21] заключалась в поиске подходящих B- и T-клеточных эпитопов, которые могут генерировать иммунный ответ против инфекции SARS-COV-2 для усиления иммунных реакций ЦТЛ и Т-хелперов. На основании экспериментов in silico , используя методы иммуноинформатики и компьютерного моделирования, получены в общей сложности 34 последовательных линейных B-клеточных эпитопа различной длины (1–62 аминокислоты) в пределах гликопротеина шипа SARS-COV-2, из них идентифицированы 13 эпитопов HLA-I и 3 эпитопа HLA-II с антигенными свойствами гликопротеина шипа SARS-COV-2, связывающихся с аллелями HLA-I, и 3 эпитопа, связывающихся с аллелями HLA-II, которые распознаются Т-клеточным рецептором TLR5 (Toll-like receptor 5) [21].
Аналогичный иммуноинформатический подход для выявления значимых эпитопов в S-белке шипа вируса SARS-CoV-2 применили V. Baruah и S. Bose [22], которые изучали взаимодействие
Таблица 1 / Table 1
Вероятные взаимодействия антигенных эпитопов SARS-COV-2 и аллелей HLA для прогнозирования антигенности [10] /
Probable SARS-COV-2 antigenic epitopes and HLA allele interaction for the antigenicity prediction [10]
|
Аллель |
Пептид (белок) |
Аффинность |
|
HLA класса I |
||
|
HLA-A*68:01 |
FTIGTVTLK (ORF3a) |
4.9 |
|
HLA-A*31:01 |
RLWLCWKCR (ORF3a) |
16.59 |
|
HLA-A*11:01 |
GTITVEELK (М) |
20.98 |
|
HLA-A*11:01 |
ITLCFTLKR (ORF7a) |
22.97 |
|
HLA-A*68:01 |
ITLCFTLKR (ORF7a) |
27.24 |
|
HLA-A*23:01 |
VFITLCFTL (ORF7a) |
60.42 |
|
HLA класса II |
||
|
HLA-DRB1*04:01 |
WLLWPVTA (М) |
2375.78 |
|
HLA-DRB1*01:01 |
VYQLRARSV (ORF7a) |
267.05 |
|
HLA-DRB1*07:01 |
VYQLRARSV (ORF7a ) |
1235.08 |
<линическая 2021 п эакти keu Том 12 №3
Таблица 2 / Table 2
Эпитопы цитотоксических Т-лимфоцитов, идентифицированные в S-гликопротеине вируса
SARS-CoV-2, и соответствующие им аллели HLA [22] /
Probable interactions of antigenic epitopes of the
SARS-COV-2 spike protein and HLA alleles [22]
За исключением ЦТЛ-эпитопа VVNQNAQAL, который на 100% идентичен SARS-CoV, все идентифицированные эпитопы ЦТЛ и B-клеток по меньшей мере частично уникальны для SARS-CoV-2. Эпитоп ЦТЛ EPVLKGVKL не совпадает ни с одной из соответствующих последовательностей других коронавирусов.
D. Santoni и D. Vergni [23] применили биоинфор-мационную методологию, основанную на выборе вирусных пептидов, которые находятся на расстоянии более трех мутационных этапов от человека. Другими словами, выбраны те вирусные пептиды, которые отсутствуют у человека (нулломеры). Эти пептиды имеют более высокую вероятность связывания HLA, чем ожидалось, поэтому иденти- фикация наиболее далеких от человека пептидов может быть интересной с точки зрения дизайна вакцины по причинам перекрестной реактивности и большего разнообразия потенциальных антител, способных их распознавать, а также во избежание риска развития аутоиммунитета.
Общее число уникальных 9-мерных пептидов в протеоме человека — 11 224 527, в то время как у SARS-CoV-2 — 9591. При этом 25 из 27 найденных нулломеров являются общими для всех известных на момент публикации статьи штаммов SARS-CoV-2, в то время как два оставшихся (IMRLWLCWK и MRLWLCWKC) отсутствуют только в одном штамме MT039890. Это неудивительно, потому что доступные в настоящее время штаммы демонстрируют более 99,98% идентичности последовательностей. Эти пептиды (названные нул-ломерами третьего порядка, то есть относящиеся к классу W4 — самому отдаленному от человека) прошли три дополнительных этапа селекции, чтобы были выбраны те из них, которые имеют наибольшую вероятность экспозиции на клеточной поверхности:
-
1) вероятность быть продуктом протеасомального расщепления;
-
2) вероятность трансфера на клеточную поверхность транспортным комплексом TAP (transporter associated with antigen presentation);
-
3) вероятность сильного связывания по меньшей мере с одним аллелем HLA из 89 рассмотренных в этом исследовании.
Идентифицирован минимальный набор из 9 пептидов, заслуживающих дальнейшего экспериментального исследования. В частности, согласно предсказанию in silico , пептид YVMHANYIF из ORF1 (nsp16) прочно связывается с 27 различными HLA-антигенами и слабо — еще с 17, что свидетельствует о высокой вероятности расщепления протеасо-мальным комплексом и очень высокой вероятности TAP-зависимой транспортировки. Предполагается, что пептиды FLCWHTNCY и YIKWPWYIW прочно связывают 11 и 10 различных HLA соответственно с высокой вероятностью протеасомального расщепления и TAP-трансфера. Пептид YYHKNNKSW из S-белка шипа также демонстрирует сильную связь с 8 HLA-аллелями и значительные показатели протеасомального расщепления и TAP-трансфера. Компьютерный прогноз с помощью программного обеспечения NetSurf [24] показывает, что пептид YYHKNNKSW находится в доступной для растворителя области белка шипа и может
кт ГА
быть дополнительно исследован как потенциальный B-клеточный эпитоп [23].
Согласно прогнозу, четыре пептида из 8 выбранных (FLCWHTNCY, WHHSIGFDY, YIKWPWYIW и YVMHANYIF) прочно связывают антигены супертипов HLA-B15 и HLA-B40, каждый из которых встречается примерно у 15% индивидов. Пептиды YVMHANYIF, YYHKNNKSW и KLMGHFAWW, по прогнозу, прочно связывают антигены супертипа А24, встречающегося у 17% людей. Сравнительный анализ в поисках этих пептидов у близкородственных видов SARS, MERS и BatСorona показал, что все 8 пептидов встречаются только у двух близкородственных Bat-SARS-подобных вирусов. Ни один из них не встречается у MERS или SARS человека. Эти выбранные in silico пептиды SARS-CoV-2 представляют собой потенциальные мишени для иммунной системы, которые надлежит экспериментально проверить для подтверждения их иммуногенности. Эти пептиды выбраны как самые отдаленные от человека с точки зрения мутаций в расчете на то, что чем больше расстояние от человека, тем выше вероятность связывания с антигенами системы HLA. Авторы исследования убеждены, что использование нулломеров высокого порядка вместо простых нулломеров — шаг вперед в этом контексте [23].
Образование антител против иммуногенных белков SARS-CoV распространено у пациентов, инфицированных этим вирусом. Не все эти антитела эффективны (то есть не все являются нейтрализующими), кроме того, они имеют короткую продолжительность жизни после выздоровления пациентов. Ответы CD4+ и CD8+ обеспечивают более длительную защиту. Высокий уровень Т-клеточно-го ответа на белок N вируса SARS-CoV обнаруживается через 2 года после выздоровления, то есть клеточный иммунитет имеет решающее значение при этих инфекциях. Предполагается, что подобный феномен вызывает и инфекция SARS-CoV-2. Концепция мультиэпитопной вакцины заключается в идентификации и сборке В- и Т-клеточных эпитопов в единый иммуноген, который может индуцировать более эффективный ответ обоих звеньев иммунитета — гуморального и клеточного. Пептиды и эпитопы оказались желательными кандидатами для разработки вакцин из-за их относительно легкого производства, химической стабильности и отсутствия инфекционного потенциала. Многоэпитопные вакцины состоят из эпитопов для B-клеток, ЦТЛ CD8+ и T-хелперов CD4+.
М. Enayatkhani и соавт. [25] сконструировали, основываясь на анализе in silico , мультиэпитопную кандидатную NOM-вакцину (Nucleocapsid, ORF3a, Membrane protein). Основная стратегия исследования заключалась в конструировании рекомбинантного мультиэпитопного белка из протеома SARS-CoV-2. Три антигенных белка (нуклеокапсид-ный фосфопротеин, ORF3a и мембранный белок) вируса SARS-CoV-2 выбраны для прогнозирования иммуногенных эпитопов В- и Т-клеток, а затем валидированы с использованием инструментов биоинформатики. Чтобы генерировать более разнообразный и стойкий гуморальный и клеточный иммунный ответ, отобраны пять обогащенных эпитопами доменов, включая высокоэффективные общие для Т- и В-клеток эпитопы, соединенные с помощью линкера. Анализ на сервере Bepipred для предсказания линейного эпитопа выявил несколько непрерывных антигенных эпитопов белков N, ORF3a и М (табл. 3) [25].
Прогнозирование антигенности кандидата на роль вакцины представляет собой определение численного показателя способности вакцины связываться с В- и Т-клеточными рецепторами и усиливать иммунный ответ. Анализ сконструированного белка NOM показал сильную антигенность без адъюванта. Разработанная кандидатная вакцина не должна, согласно предсказанию in silico , вызывать аллергическую реакцию организма. После прогнозирования наиболее перспективная трехмерная модель использована для исследований молекулярного докинга с рецепторами TLR4 и HLA-A*11:01.
На следующем этапе моделирование молекулярной динамики показало стабильность соединения рекомбинантного белка с рецепторами TLR4 и HLA-A*11:01 (модели NOM–TLR4 и NOM–HLA-A*11:01). Оценка in silico показала, что сконструированный химерный белок может одновременно вызывать гуморальный и клеточный иммунный ответ. Индекс нестабильности NOM (35,75, то есть <40) указывает на то, что сконструированный белок обладает высокой стабильностью для инициирования иммунной реакции. Трехмерная структура рекомбинантного белка NOM смодели- рована и валидирована как удовлетворительная. Взаимодействие между рекомбинантным белком NOM и рецепторами TLR4 и HLA-A*11:01 индуцирует различные иммунные ответы. Чтобы достичь стабильного и релаксированного состояния белка NOM в комплексе с иммунными рецепторами, для
<линическая 2021 п эакти keu Том 12 №3
Таблица 3 / Table Header
Предсказанные антигенные высокоаффинные эпитопы, общие для B- и T-клеток [25] / Predicted antigenic high affinity SARS-CoV-2 epitopes common to B and T cells [25]
|
Белок |
Пептид |
Аллели и супертипы HLA-I |
Пептид |
Аллели и супертипы HLA-II |
|
Нуклеокапсид |
GMSRIGMEV KHWPQIAQF FPRGQGVPI |
HLA-A*02:01 HLA-A*02:04 HLA-B*07:02 |
YKHWPQIAQ NKHIDAYKT YYRRATRRI AQFAPSASA WFTALTQHG |
HLA-DRB1*01:01 |
|
DPNFKDQVI LPNNTASWF FPRGQGVPI |
HLA-B*51 |
WFTALTQHG YYRRATRRI HWPQIAQFA TASWFTALT |
HLA-DRB1*04:01 |
|
|
FPRGQGVPI LPNNTASWF |
HLA-B*54:01 HLA-B*53:01 |
FKDQVILLN HWPQIAQFA |
HLA-DRB1*07:01 |
|
|
RPQGLPNNT DQVILLNKH |
HLA-DRB1*08:01 |
|||
|
YRRATRRIR YYRRATRRI GARSKQRRP YKHWPQIAQ WPQIAQFAP |
HLA-DRB1*11:01 |
|||
|
ASWFTALTQ YKTFPPTEP |
HLA-DRB1*15:01 |
|||
|
ORF3a |
ASLPFGWLIV LIVGVALLAV TLKKRWQLAL FVCNLLLLFV |
HLA-A*02:01 |
YYQLYSTQL YQLYSTQLS LKKRWQLAL |
HLA-DRB1*01:01 |
|
ASLPFGWLI ALSKGVHFV STDTGVEHV |
HLA-A*02:04 |
YYQLYSTQL WLIVGVALL |
HLA-DRB1*04:01 |
|
|
WLIVGVALL ALSKGVHFV NLLLLFVTV |
HLA-A*02:06 |
WLIVGVALL YYQLYSTQL IITLKKRWQ LLFVTVYSH |
HLA-DRB1*04:02 |
|
|
IPIQASLPF |
HLA-B*07:02 |
ASKIITLKK FVTVYSHLL AVFQSASKI |
HLA-DRB*10:701 |
|
|
LPFGWLIVG IPIQASLPF |
HLA-B*51 |
IITLKKRWQ RWQLALSKG |
HLA-DRB1*11:01 |
|
|
VHFVCNLLL LLLFVTVYS |
HLA-DRB1*15:01 |
|||
|
Мембранный белок |
LLEQWNLVI KLLEQWNLV FVLAAVYRI |
HLA-A*02:01 |
ICLLQFAYA FVLAAVYRI ITGGIAIAM IAIAMACLV |
HLA-DRB1*01:01 |
|
LLWPVTLAC FIASFRLFA |
HLA-A*02:04 |
FLTWICLLQ WLLWPVTLA FVLAAVYRI YRINWITGG |
HLA-DRB1*04:01 |
|
|
NLVIGFLFL FVLAAVYRI GLMWLSYFI FIASFRLFA |
HLA-A*02:06 |
FLTWICLLQ IKLIFLWLL FAYANRNRF |
HLA-DRB1*04:02 |
|
|
WPVTLACFV |
HLA-B*07:02 |
FVLAAVYRI IIKLIFLWL |
HLA-DRB1*07:01 |


Таблица 3 Окончание / End of the Table 3
|
Белок |
Пептид |
Аллели и супертипы HLA-I |
Пептид |
Аллели и супертипы HLA-II |
|
Мембранный белок |
WPVTLACFV |
HLA-B*51 |
RFLYIIKLI WLLWPVTLA WPVTLACFV |
HLA-DRB1*08:01 |
|
WPVTLACFV |
HLA-B*54:01 |
WICLLQFAY YANRNRFLY LMWLSYFIA |
HLA-DRB1*11:01 |
каждой структуры выполнено моделирование молекулярной динамики, которое гарантирует, что структуры достаточно стабильны, чтобы их можно было использовать для белок-белкового молекулярного докинга. Все три структуры оказались достаточно стабильными для следующего шага проекта.
Чтобы найти лучшую ориентацию для оптимального взаимодействия белка NOM с иммунными рецепторами, использовали белок-белковый молекулярный докинг с помощью веб-сервера PatchDock, который специализируется на распознавании и сопоставлении поверхностей белков, чтобы расположить их в наилучших возможных положениях. Ранжирование состыкованных комплексов основано на энергии ближнего, дальнего, притягивающего и отталкивающего взаимодействия между двумя белками. С помощью этого критерия исследователи выбрали два лучших решения — комплексы NOM-TLR4 и NOM-HLA-A*11:01. Результаты показали сильное взаимодействие между аминокислотами рекомбинантного белка и рецепторами. Аминокислоты HLA-A*11:01 (GLU53, ASN174, GLY56, ASN174, GLU53, PRO57) участвовали во взаимодействии с аминокислотами ARG49, SER97, GLN91, MET102, ARG49 и GLN91 вакцины-кандидата. Кроме того, восемь аминокислот TLR4 (LYS123, ILE319, THR66, SER90, LYS124, LYS46, HIS85, LYS123) участвовали во взаимодействии с аминокислотами SER45, GLU199, GLN91, ALA84, MET328, GLN65, SER90, GLU68 вакцины-кандидата. Молекулярно-динамическое моделирование лучших решений показывает, что структура белка NOM оптимизирована для взаимодействия с иммунными рецепторами. Анализ флуктуации каждого аминокислотного остатка во время моделирования указывает, что рекомбинантный белок NOM намного более стабилен, когда он находится в комплексе с этими двумя иммунными рецепторами, особенно с HLA-A*11:01.
Таким образом, методами иммуноинформатики предсказан и валидирован рекомбинантный белок NOM в качестве мультиэпитопного кандидата на вакцину против COVID-19. Его оценка основана на структурном анализе, а также на моделировании молекулярного докинга и молекулярной динамики. Комплексы NOM–TLR4 и NOM–HLA-A*11:01 высокостабильны, проявляют сильные специфичные молекулярные взаимодействия и могут стимулировать как клеточный, так и гуморальный иммунитет, учитывая, что в окончательной конструкции выбраны В- и Т-клеточные эпитопы [25].
С.Н. Lee и H. Koohy [26] утверждают, что ими идентифицирован in silico полный список иммуногенных пептидов коронавируса SARS-CoV-2, которые могут быть использованы в качестве мишеней в разработке вакцины. Среди них 48 пептидов SARS-CoV-2, имеющих высокую степень сходства с иммуногенными пептидами, депонированными в базе данных иммунных эпитопов Immune Epitope Database (IEDB), и 28 пептидов SARS-CoV-2, идентичных пептидам коронавируса SARS-CoV, охарактеризованных ранее как иммуногенные для Т-клеток. Поиск de novo с помощью алгоритма моделирования иммуногенности iPred пептидов SARS-CoV-2, распознаваемых T-клеточными рецепторами (TCR), идентифицировал 63 пептида с высоким потенциалом иммуногенности. В поиске иммуногенных пептидов SARS-CoV-2 исследователи сосредоточились на гаплотипах, распространенных в популяциях Европы и Китая и включающих в себя аллели HLA-A*02:01, HLA-A*01:01, HLA-B*07:02, HLA-B*40:01 и HLA-C*07:02. Идентичные иммуногенным пептидам коронавируса SARS-CoV 28 пептидов SARS-CoV-2 связывают различные аллели HLA как из класса I (с наиболее высокой аффинностью к HLA-A*:02:01), так и из класса II и могут быть мишенью для T-клеток CD8+ и CD4+ соответственно. В табл. 4 представлен список наиболее перспективных кандидатов, который может ускорить процесс разработки вакцины против SARS-CoV-2 [26].
На основании HLА-презентации и прогноза иммуногенности отобраны 5 пептидов (VQMAPISAM, AMYTPHTVL, TLDSKTQSL, KVDGVVQQL, KVDGVD-VEL), которые, по прогнозу, будут связывать 4 различных аллеля HLA (табл. 5).
<линическая 2021 п эакти keu Том 12 №3
В совокупности этот анализ предлагает ряд 9-мерных иммуногенных кандидатов для дальнейшей экспериментальной валидации.
Для разработки субъединичных вакцин против вируса SARS-CoV-2 на основе эпитопов проведено исследование с использованием методов обратной вакцинологии и иммуноинформатики. В результате компьютерных экспериментов разработаны три конструкции потенциальной вак- цины и на основе исследования молекулярного докинга выбрана одна из них, которая должна эффективно действовать против SARS-CoV-2. После этого проведены молекулярно-динамическое моделирование и эксперименты in silico по проверке биологической стабильности и поиску эффективной стратегии массового производства выбранной вакцины. В обратной вакцинологии инструменты биоинформатики используются для
Таблица 4 / Table 4
Наиболее иммуногенные пептиды SARS-CoV-2, идентичные иммуногенным пептидам из базы данных Immune Epitope Database (IEDB) [26] /
Most immunogenic peptides of SARS-CoV-2, identical to the immunogenic peptides from the Immune
Epitope Database (IEDB) [26]
|
Белок |
Пептид |
Аллель HLA |
|
Мембранный гликопротеин |
TLACFVLAA |
HLA-A*02:01 |
|
Нуклеопротеин |
AFFGMSRIGMEVTPSGTW |
НИ |
|
Нуклеопротеин |
ALNTPKDHI |
HLA-A*02:01 |
|
Нуклеопротеин |
AQFAPSASAFFGMSR |
HLA class II |
|
Нуклеокапсидный фосфопротеин |
AQFAPSASAFFGMSRIGM |
НИ |
|
Нуклеопротеин |
GMSRIGMEV |
HLA-A*02:01 |
|
Нуклеопротеин |
ILLNKHIDA |
HLA-A*02:01 |
|
Нуклеопротеин |
IRQGTDYKHWPQIAQFA |
НИ |
|
Нуклеопротеин |
KHWPQIAQFAPSASAFF |
НИ |
|
Нуклеопротеин |
LALLLLDRL |
HLA-A*02:01 |
|
Нуклеопротеин |
LLLDRLNQL |
HLA-A*02:01 |
|
Нуклеопротеин |
LLNKHIDAYKTFPPTEPK |
НИ |
|
Нуклеопротеин |
LQLPQGTTL |
HLA-A*02:01 |
|
Нуклеокапсидный фосфопротеин |
RRPQGLPNNTASWFT |
HLA класс I |
|
Нуклеопротеин |
YKTFPPTEPKKDKKKK |
НИ |
|
Нуклеопротеин |
ILLNKHID |
HLA-A*02:01 |
|
Нуклеокапсидный фосфопротеин |
MEVTPSGTWL |
HLA-B*40:01 |
|
S-протеин |
ALNTLVKQL |
HLA-A*02:01 |
|
Предшественник гликопротеина шипа |
FIAGLIAIV |
HLA-A2 |
|
Предшественник гликопротеина шипа |
LITGRLQS |
HLA-A2 |
|
S-протеин |
NLNESLIDL |
HLA-A*02:01 |
|
S-протеин |
QALNTLVKQLSSNFGAI |
HLA-DRB1*04:01 |
|
Предшественник гликопротеина шипа |
RLNEVAKNL |
HLA-A*02:01 |
|
S-протеин |
VLNDILSRL |
HLA-A*02:01 |
|
Предшественник гликопротеина шипа |
VVFLHVTYV |
HLA-A*02:01 |
|
S-протеин |
GAALQIPFAMQMAYRF |
HLA-DRA*01:01/DRB1*07:01 |
|
S-протеин |
MAYRFNGIGVTQNVLY |
HLA-DRB1*04:01 |
|
S-протеин |
QLIRAAEIRASANLAATK |
HLA-DRB1*04:01 |
Примечание. НИ — не идентифицирован.
Note. НИ — not identified.
Девятимерные пептиды 2019-nCoV, связывающие 4 различных аллеля HLA [26] / SARS-CoV-2 9-meric peptides that bind 4 different HLA alleles [26]
Таблица 5 / Table 5
|
Эпитоп |
A*01:01 |
A*02:01 |
B*07:02 |
B*40:01 |
C*07:02 |
|
VQMAPISAM |
0 |
1 |
1 |
1 |
1 |
|
AMYTPHTVL |
0 |
1 |
1 |
1 |
1 |
|
TLDSKTQSL |
1 |
1 |
1 |
0 |
1 |
|
KVDGVVQQL |
1 |
1 |
1 |
0 |
1 |
|
KVDGVDVEL |
1 |
1 |
1 |
0 |
1 |
Примечание. 1 — связывание с аллелем HLA; 0 — отсутствие связывания.
Note. 1 — binding to the HLA allele; 0 — no binding.
анализа генома патогена, идентификации и анализа новых антигенов. Данный подход позволяет выбрать антигенные сегменты вируса, которым следует уделять внимание при разработке вакцин. Этот метод является быстрым, эффективным, простым и экономичным способом разработки вакцины (рис. 1) [27].
В. Sarkar и соавт. [27] провели эксперимент по созданию рекомбинантной субъединичной вакцины против четырех белков SARS-CoV-2 (нуклео-капсидного фосфопротеина, который отвечает за упаковку генома и сборку вируса; поверхностного гликопротеина, который отвечает за слияние мем- бран во время проникновения вируса; белка ORF3a, ответственного за репликацию вируса, вирулентность, распространение вируса и развитие инфекции; мембранного гликопротеина, ответственного за взаимодействие вирионов с клеточными рецепторами) с использованием подходов обратной вакцинологии. Сервер IEDB генерирует эпитопы путем анализа экспериментальных данных об антителах и Т-клеточных эпитопах из исследований, проведенных в контексте аллергии, инфекционных заболеваний, аутоиммунитета и трансплантации. Исходя из оценок антигенности и аффинности связывания, из 20 лучших эпитопов для создания вак-
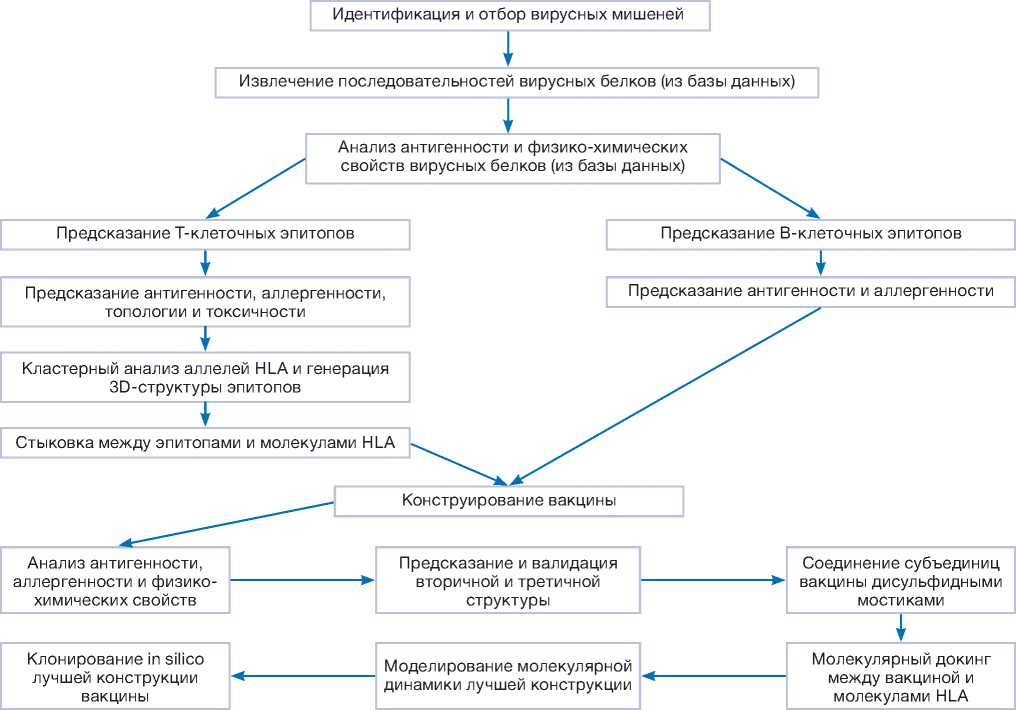
Рис. 1. Поэтапная стратегия обратной вакцинологии при разработке вакцин [27].
Fig. 1. Step-by-step strategies of the reverse vaccinology approach of vaccine development [27].
<линическая 2021 п эакти keu Том 12 №3
цины выбраны высокоантигенные, неаллергенные и нетоксичные Т-клеточные и В-клеточные эпитопы (табл. 6).
Для прогнозирования и кластерного анализа возможных аллелей HLA класса I и II, которые могут взаимодействовать с выбранными эпитопами, использован online-инструмент HLAcluster 2.0 . После прогнозирования трехмерной структуры выбранных эпитопов моделировали докинг пептид–белок, чтобы выяснить способность эпитопов связываться с молекулами HLA класса I и II. Аллель HLA-A*11:01 использовали в качестве рецептора для стыковки с эпитопами HLA класса I, а HLA-DRB1*04:01 — для стыковки с эпитопами HLA класса II. Лучшие результаты получены для пептидов из нуклеокапсид-ного фосфопротеина — QLESKMSGK с эпитопами HLA класса I и LIRQGTDYKHWP с эпитопами HLA класса II. Пептид GVLTESNKK из поверхностного гликопротеина был лучшим для эпитопов HLA класса I, а пептид TSNFRVQPTESI поверхностного гликопротеина — для эпитопов HLA класса II.
После успешной стыковки пептид–белок были сконструированы три вакцины против SARS-CoV-2 с использованием выбранных эпитопов. Для конструирования вакцин использовали три различных адъюванта (β-дефенсин, рибосомальный белок L7/L12 и белок HABA) и линкеры EAAAK, GGGS, GPGPG и KK. При конструировании вакцины использовалась последовательность PADRE, обладающая способностью увеличивать эффективность вакцин с минимальной токсичностью и усиливать ответ ЦТЛ. Созданные вакцины обозначены как CV-1, CV-2 и CV-3.
Лучшей считается конструкция вакцины с лучшим результатом по критерию молекулярного докинга. По этому критерию лучшей вакциной оказалась CV-1. Однако CV-2 продемонстрировала наибольшую аффинность связывания с HLA-DRB3*02:02 и HLA-DRB1*03:01. У вакцины CV-3 лучшие показатели энергии связывания с большинством HLA-аллелей (HLA-DRB5*01:01, HLA-DRB5*01:01, HLA-DRB1*01:01 и HLA-DRB3*01:01). Так как CV-1 показала наилучшие результаты стыковки белок–белок, она признана лучшей конструкцией из трех. Моделирование молекулярной динамики и адаптационные исследования in vitro проводились только на этой вакцине. Динамическое моделирование белков определило приемлемую относительную стабильность комплекса CV-1–TLR8. Если в испытаниях in vitro и in vivo будут получены удовлетворительные результаты, предложенные вакцинные конструкции могут быть использованы для вакцинации против SARS-CoV-2 и предотвращения распространения инфекции [27].
Несколько групп разработали субъединичные вакцины против SARS-CoV-2, однако для разработки вакцины они использовали либо один белок, либо только эпитопы ЦТЛ без В-клеточ-ных и ХТЛ-эпитопов (хелперный Т-лимфоцит). Конструкция новой рекомбинантной мультипеп-тидной субъединичной вакцины против COVID-19 содержит ЦТЛ-клеточные, ХТЛ- и В-клеточные эпи-
Таблица 6 / Table 6
Эпитопы SARS-CoV-2, соответствующие критериям отбора (высокая антигенность, неаллергенность и нетоксичность) и выбранные для создания вакцины [27] /
The SARS-CoV-2 epitopes that fit the selection criteria (high antigenicity, non-allergenicity and non-toxicity) and were selected for the vaccine creation [27]
|
Белок |
Эпитопы HLA класса I |
Эпитопы HLA класса II |
В-клеточные эпитопы |
|
Нуклеокапсидный фосфопротеин |
AGLPYGANK AADLDDFSK QLESKMSGK |
QELIRQGTDYKH LIRQGTDYKHWP RLNQLESKMSGK LNQLESKMSGKG LDRLNQLESKMS |
MSDNGPQNQRNAPRITFGGPSDSTGSNQNGERSGARSK QRRPQGLPNNTAS RIRGGDGKMKDL TGPEAGLPYGANK GTTLPKGFYAEGSRGGSQASSRSSSRSRNSSRNSTPGSS RGTSPARMAGNGGD SKMSGKGQQQQGQTVTKKSAAEASKKPRQKRTATKAYN KTFPPTEPKKDKKKKADETQALPQRQKKQQ |
|
Поверхностный гликопротеин |
SVLNDILSR GVLTESNKK RLFRKSNLK QIAPGQTGK |
TSNFRVQPTESI SNFRVQPTESIV LLIVNNATNVVI |
LTPGDSSSGWTAG VRQIAPGQTGKIAD YQAGSTPCNGV QTQTNSPRRARSV ILPDPSKPSKRS |
КТ ГА
топы, соединенные специфическими линкерами. Эта уникальная вакцина состоит из 33 высокоантигенных эпитопов из трех белков SARS-CoV-2, которые участвуют в распознавании клеточных рецепторов, проникновении вируса в клетку и патогенности (табл. 7). Все отобранные для вакцины ХТЛ-, ЦТЛ- и B-клеточные эпитопы нетоксичны. Расчетные данные позволяют предположить, что вакцина неаллергенна, термостабильна и обладает способностью вызывать гуморальный и клеточный иммунный ответ. Стабильность конструкции вакцины подтверждается исследованиями по моделированию молекулярной динамики [28].
Для конструирования субъединичной вакцины против SARS-CoV-2 использованы в общей сложности 6 ХТЛ-, 18 ЦТЛ- и 9 B-клеточных эпитопов из трех белков. Эпитопы ХТЛ протестированы сервером IFN-эпитопов ifnepitope/) на способность индуцировать иммунный ответ типа Th1, сопровождающийся продукцией IFN-γ. Для усиления иммуногенности вакцинной конструкции в качестве адъюванта добавлена последовательность из 68 аминокислотных остатков человеческого β-дефенсина — агониста рецептора TLR3. Эпитопы ЦТЛ необходимы для индукции клеточного иммунного ответа с целью нейтрализации
Таблица 7 / Table 7
ЦТЛ-клеточные, ХТЛ-клеточные и В-клеточные эпитопы в рекомбинантной мультипептидной субъединичной вакцине против COVID-19 [28] /
CTL-cell, HTL-cell and B-cell epitopes in the recombinant multi-epitope subunit COVID-19 vaccine [28]
|
Специфичность |
Белок |
Эпитоп |
Аллель HLA |
|
ХТЛ-специфичные эпитопы |
Нуклеокапсидный фосфопротеин |
GTWLTYTGAIKLDDK AALALLLLDRLNQLE |
DRB1*07:01 DRB4*01:01 |
|
Мембранный гликопротеин |
NRFLYIIKLIFLWLL |
DRB4*01:01 |
|
|
Гликопротеин шипа |
EFVFKNIDGYFKIYS ITRFQTLLALHRSYL ATRFASVYAWNRKRI |
DRB5*01:01 |
|
|
В-специфичные эпитопы |
Нуклеокапсидный фосфопротеин |
TRRIRGGDGKMKDLSP KSAAEASKKPRQKRTA EGALNTPKDHIGTRNP |
Нет данных |
|
Мембранный гликопротеин |
RSMWSFNPETNILLNV SFRLFARTRSMWSFNP |
||
|
Гликопротеин шипа |
YACWHHSIGFDYVYNP VVKIYCPACHNSEVGP TLKGGAPTKVTFGDDT TSRYWEPEFYEAMYTP |
||
|
ЦТЛ-специфичные эпитопы |
Нуклеокапсидный фосфопротеин |
LLLDRLNQL GMSRIGMEV |
Супертип A2 |
|
KSAAEASKK KTFPPTEPK |
Супертип A3 |
||
|
FPRGQGVPI KPRQKRTAT |
Супертип В7 |
||
|
Мембранный гликопротеин |
GLMWLSYFI FVLAAVYRI |
Супертип A2 |
|
|
LSYFIASFR RIAGHHLGR |
Супертип A3 |
||
|
LPKEITVAT RLFARTRSM |
Супертип В7 |
||
|
Гликопротеин шипа |
YLQPRTFLL KIADYNYKL |
Супертип A2 |
|
|
RLFRKSNLK GVYFASTEK |
Супертип A3 |
||
|
SPRRARSVA IPTNFTISV |
Супертип В7 |
Примечание. ЦТЛ — цитотоксический Т-лимфоцит; ХТЛ — хелперный Т-лимфоцит.
Note: ЦТЛ — cytotoxic T-lymphocyte, T-helper cell; ХТЛ — helper T-lymphocyte.
и ни не с кая ь™
пэактика -21
инфицированных вирусом и поврежденных клеток путем высвобождения цитотоксических белков, таких как гранзимы, перфорины и т.д. Для прогнозирования рассмотрены супертипы HLA A2, A3 и B7, поскольку они охватывают не менее 88,3% общей численности населения. Все выбранные ХТЛ-, ЦТЛ-и B-клеточные эпитопы протестированы in silico на токсичность с помощью модуля ToxinPred .
Иммунизация на основе белка может потребовать повторного введения вакцины для поддержания терапевтически эффективного уровня. Кроме того, результатом введения этих вакцин могут быть некоторые неспецифические эффекты, такие как перекрестно-реактивные TCR и антитела, активация ранее существовавших клеток-эффекторов или клеток памяти. Именно поэтому вакцины на основе мРНК SARS-CoV-2 потенциально имеют преимущества над вакцинами на основе белка: они безопаснее, эффективнее и проще в изготовлении. Производство вакцин на основе мРНК является новым перспективным направлением вакцинологии. Существуют два типа РНК-вакцин против инфекционных патогенов: нереплицирующиеся мРНК-вак-цины и самоамплифицирующиеся, или репликон-ные, РНК-вакцины. Проникновение через липидный мембранный барьер является первым шагом для попадания экзогенной мРНК в цитоплазму. Механизмы поглощения мРНК-вакцин клеточноспецифичны, а физико-химические свойства мРНК могут существенно влиять на ее клеточную доставку и распределение в органах. Все эти факторы необходимо учитывать при разработке эффективной вакцины на основе мРНК. Несмотря на это, мР-НК-вакцина считается наиболее перспективным кандидатом, поскольку ее можно быстро масштабировать, что может сэкономить время в условиях пандемии.
В марте 2020 года в США началось первое клиническое исследование фазы I новой вакцины на основе мРНК — mRNA-1273, инкапсулированной в липидные наночастицы [Safety and Immunogenicity Study of 2019-nCoV Vaccine (mRNA-1273) for Prophylaxis of SARS-CoV-2 Infection (COVID-19). Identifier: NCT04283461. ]. Вакцина mRNA-1273 кодирует S-белок шипа SARS-CoV-2. Клинические испытания вакцины mRNA-1273 на безопасность и иммуногенность провели среди здоровых волонтеров американская биотехнологическая компания Moderna Inc. и Центр исследований вакцин (Vaccine Research Center) Национального института аллергии и инфекционных заболеваний (National Institute of Allergy and Infectious Diseases). В ноябре 2020 г. Moderna опубликовала данные клинического исследования фазы III об 94%-ной эффективности в предотвращении заражения COVID-19. Побочные эффекты представляли собой гриппоподобные симптомы. В настоящее время вакцина Moderna (mRNA-1273) рекомендована Всемирной организацией здравоохранения и применяется для иммунизации лиц в возрасте 18 лет и старше.
В инфицированной коронавирусом SARS-CoV-2 клетке синтезируются полипротеин 1a/1ab (pp1a/ pp1ab) и 16 неструктурных белков репликаци-онно-транскрипционного комплекса для синтеза дискретных субгеномных РНК (sgRNA), которые служат матрицами для синтеза субгеномных мРНК. Два полипептида (pp1a и pp1ab) возникают из-за сдвига рамки считывания между ORF1a и ORF1b. Другие ORF (35% генома) кодируют структурные белки S, M, E и N. Все эти структурные и неструктурные белки транслируются с sgRNA. В настоящее время более 200 полных и частичных последовательностей генома SARS-CoV-2 депонированы в базе данных Global Initiative on Sharing All Influenza Data (GISAID; и в базе данных GenBank Национального института здоровья США (NIH; nuccore/?term=covid-19). Вакцины, основанные на цитоплазматической экспрессии химерных мРНК, содержащих специально подобранные вирусные последовательности ORF, обладают огромным потенциалом прямой трансляции в цитоплазму и блокирования интеграции вируса с хромосомами. После инъекции вакцинная мРНК может процессироваться иммунными клетками и продуцировать белок непосредственно в процессе трансляции, за которой следует активация других иммунных клеток для распознавания вновь продуцированного вирусного белка и образования антител [29].
Коронавирус SARS-CoV-2 обладает РНК, само-амплифицирующейся в цитозоле. Это обстоятельство является основой разработки мРНК-вакцины. Вакцины на основе мРНК используют последовательность мРНК рекомбинантного белка-мишени, а не антитела-мишени. Затем мРНК переносится липидными наночастицами в цитоплазму для трансляции белка-мишени. Когда белок-мишень высвобождается из клетки, антигенпрезентирующие клетки быстро захватывают и процессируют его.
КТ ГА
Затем происходит презентация молекулами HLA-I или HLA-II на поверхности антигенпрезентирующей клетки с последующей активацией В- и Т-клеток и, соответственно, гуморального и цитотоксического ответа [29].
Поиск наиболее подходящего сайта-мишени для разработки вакцины против SARS-CoV-2 чрезвычайно важен. Гликопротеин шипа (белок S) является ключевой мишенью для разработки вакцин и генерации терапевтических антител. Вирус SARS-CoV-2 проникает в клетку-хозяина путем слияния гомотримерного S-белка с клеточной мембраной, при этом субъединица S1 связывается с рецептором клетки, что вызывает нестабильность тримера, за которой следует отделение субъединицы S1 от субъединицы S2 с образованием высокостабильной структуры. Белок S помогает вирусу проникать в клетки-мишени, но этот эндоцитоз одновременно зависит как от связывания белка S с мембранным рецептором ACE2, так и от активации белка S клеточными протеазами. Вакцина может предотвратить первоначальную активацию S-белка, блокируя его связывание с ACE2. Если титр антител к S-белку достаточно высок, чтобы предотвратить попадание вируса в эндосомы или его слияние с клеточной поверхностью, то вирусная инфекция, связанная с активацией S-белка и внутриклеточным высвобождением вирусных частиц, будет эффективно ингибироваться.
Если в организме образуется достаточное количество антител к S-белку, то с учетом биодоступности ACE2 как ключевой детерминанты распространения и инфицирующей способности SARS-CoV-2 может возникнуть двойной терапевтический эффект. Во-первых, иммунная система удалит ком-лекс S-белок–антитело, что приведет к клиренсу самого вируса. Во-вторых, биодоступность ACE2 будет значительно снижена, что поможет ослабить распространение и контагиозность вируса. Эмпирические данные исследований SARS-CoV и SARS-CoV-2 определили ACE2 в качестве основного портала для проникновения этих вирусов в клетку. Функционально значимые мутации S-белка, по-ви-димому, обеспечивают более высокую аффинность связывания по сравнению с SARS-CoV [30]. Следовательно, нет гарантии, что нацеленная на S-белок вакцина может использоваться в течение длительного времени, так как существует вероятность того, что в ближайшем будущем она утратит эффективность из-за мутаций. Тем не менее мРНК-1273 индуцирует мощные ответы нейтрализующих антител как на SARS-CoV-2 дикого типа (D614), так и на мутантный D614G 2 SARS-CoV-2, а также Т-клеточные ответы CD8+ и защиту от инфекции SARS-CoV-2 в легких и носу in vivo без признаков иммунопатологии [31].
Существуют четыре основных преимущества использования противовирусных вакцин на основе мРНК по сравнению с традиционными подходами:
-
1) противовирусные вакцины на основе мРНК минимизируют потенциальный риск инфицирования и мутагенеза вследствие естественной деградации мРНК в клеточной микросреде;
-
2) высокая эффективность иммуногена благодаря структурным модификациям мРНК повышает его стабильность и эффективность трансляции;
-
3) высокоэффективные вакцины на основе мРНК способны генерировать противовирусные нейтрализующие иммуноглобулины только с одной или двумя низкими дозами иммунизации;
-
4) рекомбинантная мРНК способствует крупномасштабному производству достаточных доз вакцины, необходимых для лечения больших групп населения.
Эти факторы делают мРНК-вакцину более подходящей для быстрого реагирования на пандемию COVID-19 [29].
Гликозилирование является очень распространенным биологическим явлением, одной из форм посттрансляционной модификации белков и способом регуляции локализации и функции белков. У вирусов гликозилирование их структурных белков тесно связано с инвазией в клетки и репликацией, что помогает вирусу избежать обнаружения иммунной системой человека. Необычно высокая степень гликозилирования SARS-CoV-2 означает возможность быстро мутировать, что чрезвычайно затрудняет разработку вакцины. Однако использование технологии получения вакцины на основе мРНК и нацеливание только на белок S, а не на целую вирусную частицу, может привести к тому, что иммунная система человека будет продуцировать антитела к белку S, невзирая на сатус гликозилирования.
Принимая во внимание полезные свойства вакцин на основе мРНК, такие как отсутствие интеграции в геном, отсутствие индукции аутоантител, возможность продуцировать мРНК-вакцины в больших количествах и их высокую чистоту, вакцина на основе мРНК — перспективный выбор для борьбы с COVID-19. Однако для обеспечения стабильности мРНК-вакцины требются новые спосо-
<линическая 2021 п эакти keu Том 12 №3
бы упаковки вакцины в наночастицах и очистки или удаления компонентов реакции, связанных с мРНК-вакциной. Кроме того, требуют разработки новые способы доставки in vivo , чтобы максимально увеличить эффективность поглощения мРНК-вакцины. Наилучший способ должен заключаться в закреплении ингибитора РНКазы в единой структуре мРНК-вакцины. Необходимы углубленные исследования структуры и свойств S-белка и других структурных белков вируса SARS-CoV-2, которые используются в разработке вакцин [29].
Еще одна вакцина против COVID-19 на базе мРНК — вакцина Pfizer/BioNTech (BNT162b2), разработанная немецкой биотехнологической компанией BioNTech в сотрудничестве с американской Pfizer и китайской Fosun Pharma, прошла полный цикл клинических испытаний и уже широко применяется для вакцинации населения, однако мониторинг первичных результатов фазы III будет продолжаться до августа 2021 года, а мониторинг вторичных результатов — до января 2023 года [32]. Вакцина BNT162b2 от Pfizer/BioNTech, как и вакцина мРНК-1273 от Moderna, основана на стабилизированной мРНК, кодирующей S-белок вируса SARS-CoV-2 [33].
ЗАКЛЮЧЕНИЕ
В отсутствие специфического лечения вакцина является лучшим вариантом борьбы с COVID-19. Вычислительные исследования с последующими доклиническими испытаниями показывают, что субъединичная вакцина на основе нескольких эпитопов может быть безопасна и эффективна против инфекции SARS-CoV-2.
Индивидуальные генетические вариации системы HLA-антигенов могут объяснить различные иммунные реакции на вирус в популяции, поэтому в разработке вакцин важная роль принадлежит выявлению значимых эпитопов в протеоме SARS-CoV-2, распознаваемых антигенпрезентирующими клетками, ЦТЛ и В-клетками, с использованием методов биоинформационного анализа.
ДОПОЛНИТЕЛЬНАЯ ИНФОРМАЦИЯ
Вклад авторов. Щербак С.Г., Голота А.C. — написание текста статьи; Шнейдер О.В., Вологжанин Д.А. — редактирование текста статьи; Камилова Т.А. — поисково-аналитическая работа, написание, обсуждение и редактирование текста статьи. Авторы подтверждают соответствие своего авторства международным критериям ICMJE (все авторы внесли существенный вклад в разработку концепции, проведение исследования и подготовку статьи, прочли и одобрили финальную версию перед публикацией).
Author contribution. Shcherbak S.G., Golota A.S. — manuscript writing; Shneider О.V., Vologzhanin D.А. — manuscript revision; Kamilova T.A. — search and analytical work, manuscript writing and revision. The authors made a substantial contribution to the conception of the work, acquisition, analysis, interpretation of data for the work, drafting and revising the work, final approval of the version to be published and agree to be accountable for all aspects of the work.
Источник финансирования. Авторы заявляют об отсутствии внешнего финансирования при проведении исследования.
Funding source. This study was not supported by any external sources of funding.
Список литературы Компьютерное моделирование в разработке вакцин против COVID-19 на основе антигенов HLA-системы
- Gorbalenya AE, Baker SC, Baric RS, et al. Severe acute respiratory syndrome-related coronavirus: the species and its viruses, a statement of the Coronavirus Study Group. BioRxiv. 2020;1-15. doi: 10.1101/2020.02.07.937862
- Lu R, Zhao X, Li J, et al. Characterisation and epidemiology of 2019 novel coronavirus: implications for virus origins and receptor binding. Lancet. 2020;395(10224):565-574. doi: 10.1016/S0140-6736(20)30251-8
- Forouzesh M, Rahimi A, Valizadeh R, et al. Clinical display, diagnostics and genetic implication of novel Coronavirus (COVID-19) epidemic. Eur Rev Med Pharmacol Sci. 2020;24(8):4607-4615. doi: 10.26355/eurrev_202004_21047
- Tian X, Li C, Huang A, et al. Potent binding of 2019 novel coronavirus spike protein by a SARS coronavirus-specific human monoclonal antibody. Emerg Microb Infect. 2020;9(1):382-385. doi: 10.1080/22221751.2020.1729069
- Самсонова М.В., Черняев А.Л., Михалева Л.М., и др. Патологическая анатомия легких при COVID-19. Атлас / под общей ред. О.В. Зайратьянца. Москва; Рязань, 2020. 52 с. [Sam-sonova MV, Chernyaev AL, Mikhaleva LM, et al. Vitiatam anatomia pulmo in COVID-19. Atlas. Ed. by O.V. Zairatyants. Moscow; Ryazan; 2020. 52 p. (In Russ).]
- Arabi YM, Murthy S, Webb S. COVID-19: a novel coronavirus and a novel challenge for critical care. Intensive Care Med. 2020;46(5):833-836. doi: 10.1007/s00134-020-05955-1
- Giamarellos-Bourboulis EJ, Netea MG, Rovina N, et al. Complex immune dysregulation in COVID-19 patients with severe respiratory failure. Cell Host Microbe. 2020;27(6):992-1000.e3. doi: 10.1016/j.chom.2020.04.009
- Nguyen A, David JK, Maden SK, et al. Human leukocyte antigen susceptibility map for SARS-CoV-2. J Virol. 2020;94(13):e00510-e00520. doi: 10.1128/JVI.00510-20
- Kiyotani K, Toyoshima Y, Nemoto K, Nakamura Y. Bioinfor-matic prediction of potential T cell epitopes for SARS-Cov-2. J Hum Genet. 2020;65(7):569-575.doi: 10.1038/s10038-020-0771-5
- Joshi A, Joshi BC, Mannan MA, Kaushik V. Epitope based vaccine prediction for SARS-COV-2 by deploying immuno-in-formatics approach. Inform Med Unlocked. 2020;100338. doi: 10.1016/j.imu.2020.100338
- Wang F, Hou H, Luo Y, et al. The laboratory tests and host immunity of COVID-19 patients with different severity of illness. JCI Insight. 2020;5(10):e137799. doi: 10.1172/jci.insight.137799
- Alijotas-Reig J, Esteve-Valverde E, Belizna C, et al. Immunomodulatory therapy for the management of severe COVID-19. Beyond the anti-viral therapy: A comprehensive review. Autoimmun Rev. 2020;19(7):102569. doi: 10.1016/j.autrev.2020.102569
- Shi Y, Wang Y, Shao C, et al. COVID-19 infection: the perspectives on immune responses. Cell Death Differ. 2020;27(5):1451-1454. doi: 10.1038/s41418-020-0530-3
- Li X, Geng M, Peng Y, Meng L, Lu S. Molecular immune pathogenesis and diagnosis of COVID-19. J Pharm Anal. 2020; 10(2): 102-108. doi: 10.1016/j.jpha.2020.03.001
- Holshue ML, DeBolt C, Lindquist S, et al. First case of 2019 novel coronavirus in the United States. N Engl J Med. 2020;382(10):929-936. doi: 10.1056/NEJMoa2001191
- Xu Z, Shi L, Wang Y, et al. Pathological findings of COVID-19 associated with acute respiratory distress syndrome. Lancet Respir Med. 2020;8(4):420-422. doi.: 10.1016/S2213-2600(20)30076-X
- Zhang S, Gan J, Chen BG, et al. Dynamics of peripheral immune cells and their HLA-G and receptor expressions in a patient suffering from critical COVID-19 pneumonia to convalescence. Clin Transl Immunology. 2020;9(5):e1128. doi: 10.1002/cti2.1128
- Wang W, Zhang W, Zhang J, et al. Distribution of HLA allele frequencies in 82 chinese individuals with Coronavirus Disease-2019 (COVID-19). HLA. 2020;96(2):194-196. doi: 10.1111/tan.13941
- Thevarajan I, Nguyen HO, Koutsakos M, et al. Breadth of concomitant immune responses prior to patient recovery: a case-report of non-severe COVID-10. Nat Med. 2020;26(4):453-455. doi: 10.1038/s41591 -020-0819-2
- Jamilloux Y, Henryb T, Belot A, et al. Should we stimulate or suppress immune responses in COVID-19? Cytokine and anti-cytokine interventions. Autoimmun Rev. 2020;19(7):102567. doi: 10.1016/j.autrev.2020.102567
- Bhattacharya M, Sharma AR, Patra P, et al. Development of epitope-based peptide vaccine against novel coronavirus 2019 (SARS-COV-2): Immunoinformatics approach. J Med Virol. 2020;92(6):618-631. doi: 10.1002/jmv.25736
- Baruah V, Bose S. Immunoinformatics-aided identification of T cell and B cell epitopes in the surface glycoprotein of SARS-CoV-2. J Med Virol. 2020;92(5):495-500. doi: 10.1002/jmv.25698
- Santoni D, Vergni D. In the search of potential epitopes for Wuhan seafood market pneumonia virus using high order nullomers. J Immunol Methods. 2020;481 -482:112787. doi: 10.1016/j.jim.2020.112787
- Klausen MS, Jespersen MC, Nielsen H, et al. NetSurfP-2.0: Improved prediction of protein structural features by integrated deep learning. Proteins. 2019;87(6):520-527. doi: 10.1002/prot.25674
- Enayatkhani M, Hasaniazad M, Faezi S, et al. Reverse vacci-nology approach to design a novel multi-epitope vaccine candidate against COVID-19: an in silico study. J Biomol Struct Dyn. 2020;1-16. doi: 10.1080/07391102.2020.1756411
- Lee CH, Koohy H. In silico identification of vaccine targets for SARS-CoV-2. F1000Res. 2020;9:145. doi: 10.12688/f1000research.22507.2
- Sarkar B, Ullah A, Johora FT, et al. Immunoinformat-ics-guided designing of epitope-based subunit vaccine against the SARS Coronavirus-2 (SARS-CoV-2). Immunobiology. 2020;225(3):151955. doi: 10.1016/j.imbio.2020.151955
- Kalita P, Padhi AK, Zhang KY, Tripathi T. Design of a pep-tide-based subunit vaccine against novel coronavirus SARS-CoV-2. MicrobPathog. 2020;145:104236. doi: 10.1016/j.micpath.2020.104236
- Wang F, Kream RM, Stefano GB. An evidence based perspective on mRNA-SARS-CoV-2 vaccine development. Med Sci Monit. 2020;26:e924700. doi: 10.12659/MSM.924700
- Ortega JT, Serrano ML, Pujol FH, Rangel HR. Role of changes in SARS-CoV-2 spike protein in the interaction with the human ACE2 receptor: an in silico analysis. EXCLI J. 2020;19:410-417. doi: 10.17179/excli2020-1167
- Corbett KS, Edwards DK, Leist SR, et al. SARS-CoV-2 mRNA vaccine design enabled by prototype pathogen preparedness. Nature. 2020;586(7830):567-571. doi: 10.1038/s41586-020-2622-0
- Walsh EE, Frenck RW, Falsey AR, et al. Safety and immunogenicity of two RNA-based Covid-19 vaccine candidates. N Engl J Med. 2020;383(25):2439-2450. doi: 10.1056/N EJMoa2027906
- Xia X. Domains and functions of spike protein in SARS-Cov-2 in the context of vaccine design. Viruses. 2021;13(1):109. doi: 10.3390/v13010109


