Молекулярно-генетические аспекты типизации вируса лейкоза крупного рогатого скота
Автор: Шаева А.Ю., Вафин Р.Р., Хазипов Н.З., Камалов Б.В., Алимов А.М., Тагиров М.Ш.
Статья в выпуске: 1 т.205, 2011 года.
Бесплатный доступ
Целью нашего исследования являлось определение таксономической принадлежности изолятов ВЛКРС, выделенных в хозяйствах РТ, в контексте молекулярно-генетических аспектов типизации BLV. Системный мониторинг инфицированности стад ВЛКРС на генотипическом уровне идентификации возбудителя будет способствовать повышению эффективности противолейкозных мероприятий.
Влкрс, типизация, пцр, пдрф, филогенетический анализ
Короткий адрес: https://sciup.org/14287144
IDR: 14287144 | УДК: 619:57.065:578.828
Текст научной статьи Молекулярно-генетические аспекты типизации вируса лейкоза крупного рогатого скота
Лейкоз крупного рогатого скота – инфекционная болезнь опухолевой природы, этиологическим агентом которого является вирус лейкоза кр.рог.ск. (ВЛКРС, англ. Bovine leukemia virus, BLV), относящийся к роду Deltaretrovirus семейства Retroviridae [1, 2, 3, 4, 5].
Вопросы диагностики данного заболевания в контексте изучения генетического многообразия его этиологического агента весьма актуальны.
Ряд разработанных на основе интерпретации env -ПЦР-ПДРФ-профилей стратегий типизации BLV [1, 2, 3] имеют определенную идентификационную ценность, однако не способны охарактеризовать весь спектр генотипического разнообразия ВЛКРС [4, 5].
В 2009 году S.M. Rodriguez et al. [5] была предложена система классификации BLV на основе филогенетического анализа env -гена, предусматривающая наличие 7 генотипов ВЛКРС. Действенность данной концепции типизации BLV отражена в работе другой научной группы [4].
Целью нашего исследования являлось определение таксономической принадлежности изолятов ВЛКРС, выделенных в хозяйствах РТ, в контексте молекулярно-генетических аспектов типизации BLV.
Материалы и методы. В работе были использованы 2 изолята ВЛКРС: «N10» и «N28», выделенные от кр.рог.ск. из Мензелинского и Спасского районов Республики Татарстан, соответственно.
При постановке ПЦР с выделенными образцами провирусной ДНК BLV использовалась модификация «nested» ПЦР с применением внешних (env5032+env5608) и внутренних (env5099+env5521) праймеров, инициирующих на заключительном этапе реакции амплификацию фрагмента env -гена ВЛКРС длиной 444 п.н. [1, 2, 3].
На этапе ПДРФ-анализа амплификаты подвергались гидролизу с использованием эндонуклеаз рестрикции BamHI , PvuII, Ksp22I и HaeIII .
ПЦР-ПДРФ-продукты разгоняли в 2,5% агарозном геле (20 В/см, 40 мин) и визуализировали в УФ-трансиллюминаторе (λ=310 нм).
Секвенирование продуктов амплификации локуса env -гена BLV с внутренними праймерами «env5099» и «env5521» изолятов «N10» и «N28» ВЛКРС выполнено на приборе ABI-300 в лабораториях НПО «СибЭнзим».
Изоляты «N10» (GenBank A/N: HM102355) и «N28» (GenBank A/N: HM102356) были выравнены по длине 444 нуклеотидов локуса env -гена c соответствующими опубликованными в GenBank последовательностями представителей известных генотипов BLV, используя программы BLAST и CLUSTAL W (v. 1.83) с последующим филогенетическим анализом.
Результаты исследования и их обсуждение.
Интерпретация ПЦР - ПДРФ - профилей BLV по D. Beier et al. (2001)
В результате реализации стратегии типизации представителей ВЛКРС на основе интерпретации ПЦР-ПДРФ-профилей BLV по D. Beier et al. (2001) [1], установлено, что изолят «N10», выделенный от кр.рог.ск. из Мензелинского района, характеризуется признаком Бельгийского подтипа, а изолят «N28», выделенный от кр.рог.ск. из Спасского района, относится к Австралийскому подтипу. Детальная информация, отражающая характеристику заявленных ПДРФ-вариантов ВЛКРС по протоколу Beier et al. (2001) [1], представлена в табл. 1, а результаты ПЦР-ПДРФ-анализа исследованных нами изолятов «N10» и «N28» ВЛКРС отражены на рис. 1.
1. Env -ПЦР-ПДРФ-профили BLV (праймеры env5099-env5 521 )
|
ПДРФ-вариант ВЛКРС |
ПЦР-продукт (п.н.) |
ПДРФ-фрагменты (п.н.) |
||
|
PvuII |
BamHI |
BclI ( Ksp22I) |
||
|
Бельгийский подтип |
444 |
280/164 |
444 |
225/219 |
|
Австралийский подтип |
444 |
444 |
316/128 |
225/219 |
|
Японский подтип |
444 |
444 |
316/128 |
219/121/104 |
|
1.0 1.1 1.2 1.3 М |
2.0 2.1 |
2.2 2.3 |
||
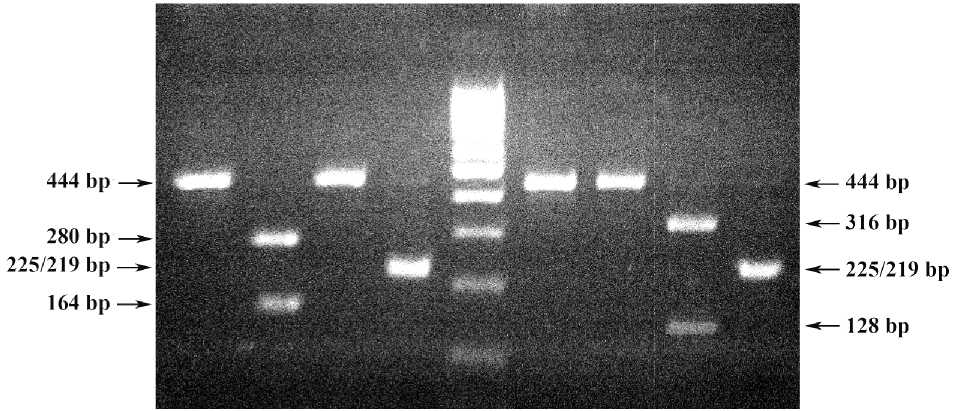
1. Электрофореграмма env -ПЦР-ПДРФ-профилей изолятов N10 и N28 BLV
Обозначения: 1.0-1.3) ПЦР-ПДРФ-профиль изолята N10 BLV: 1.0) ПЦР-продукт (444 bp); 1.1) PvuII -ПДРФ (280/164 bp); 1.2) BamHI -ПДРФ (444 bp); 1.3) Ksp22I -ПДРФ (225/219 bp). М) ДНК-маркеры 100-1000 bp (СибЭнзим). 2.0-2.3) ПЦР-ПДРФ-профиль изолята N28 BLV: 2.0) ПЦР-продукт (444 bp): 2.1) PvuII -ПДРФ (444 bp); 2.2) BamHI -ПДРФ (316/128 bp); 2.3) Ksp22I -ПДРФ (225/219 bp).
Однако, разработанная D. Beier et al. (2001) [1] типизация ВЛКРС, а также ранее предложенная H/ Fechner et al (1997) [2] классификация не позволяют охватить весь спектр генетического многообразия BLV [4, 5].
Интерпретация ПЦР - ПДРФ - профилей BLV по M. Licursi et al. (2002)
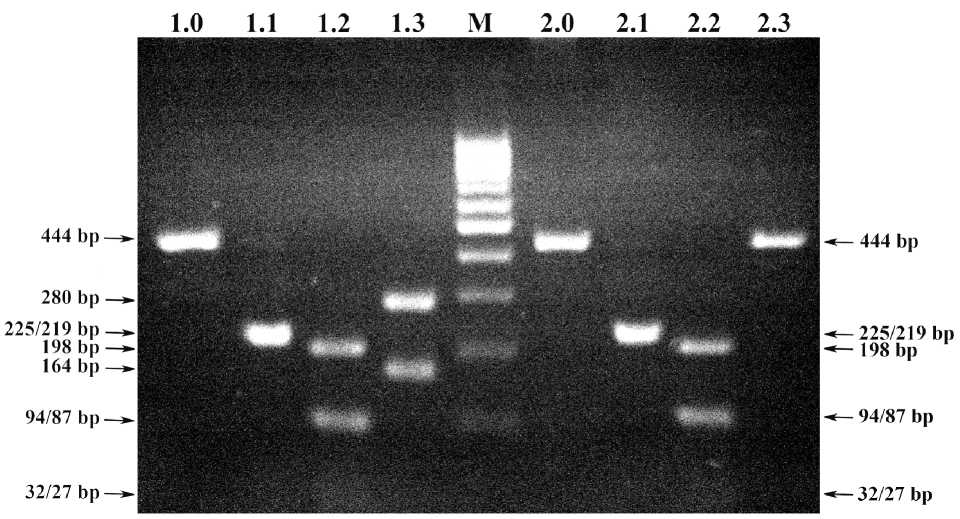
В результате реализации стратегии типизации представителей ВЛКРС на основе интерпретации ПЦР-ПДРФ-профилей BLV по M. Licursi et al. (2002) [3], установлено, что изолят «N10» относится к 6-му генотипу, а изолят «N28» – к 1-му генотипу.
Детальная информация, отражающая характеристику заявленных генотипов ВЛКРС по протоколу M. Licursi et al. (2002) [3], представлена в табл. 2, а результаты ПЦР-ПДРФ-анализа исследованных нами изолятов «N10» и «N28» ВЛКРС отражены на рис. 2.
2. Env -ПЦР-ПДРФ-профили BLV (праймеры env5099-env5521)
|
Генотип |
ПЦР-продукт (п.н.) |
ПДРФ-фрагменты (п.н.) |
||
|
BclI ( Ksp22I) |
HaeIII |
PvuII |
||
|
1 |
444 |
225/219 |
198/94/87/32/27/6 |
444 |
|
2 |
444 |
219/121/104 |
312/94/32/6 |
444 |
|
3 |
444 |
219/121/104 |
285/94/32/27/6 |
444 |
|
4 |
444 |
219/121/104 |
198/94/87/32/27/6 |
444 |
|
5 |
444 |
225/219 |
285/94/32/27/6 |
444 |
|
6 |
444 |
225/219 |
198/94/87/32/27/6 |
280/164 |
1.0 1.1 1.2 1.3 М 2.0 2.1 2.2 2.3
-— 444 bp
94/87 bp
32/27 bp
-—225/219 bp
— 198 bp
444 bp
280 bp
225/219 bp
198 bp
164 bp
94/87 bp
32/27 bp
2. Электрофореграмма env -ПЦР-ПДРФ-профилей изолятов N10 и N28 BLV
Обозначения: 1.0-1.3) ПЦР-ПДРФ-профиль изолята N10 BLV: 1.0) ПЦР-продукт (444 bp); 1.1) Ksp22I -ПДРФ (225/219 bp); 1.2) HaeIII -ПДРФ (198/94/87/32/27/6 bp); 1.3) PvuII -ПДРФ (280/164 bp). М) ДНК-маркеры 100-1000 bp (СибЭнзим). 2.0-2.3) ПЦР-ПДРФ-профиль изолята N28 BLV: 2.0) ПЦР-продукт (444 bp): 2.1) Ksp22I -ПДРФ (225/219 bp); 2.2) HaeIII -ПДРФ (198/94/87/32/27/6 bp); 2.3) PvuII -ПДРФ (444 bp).
Следует отметить, что разработанная M. Licursi et al. (2002) [3] система генотипирования представителей ВЛКРС также не позволяет охарактеризовать весь спектр генотипического разнообразия BLV, в отличие от филогенетической классификации, предложенной S.M. Rodriguez et al. (2009) [5], регламентирующей наличие 7 генотипов (рис. 3).
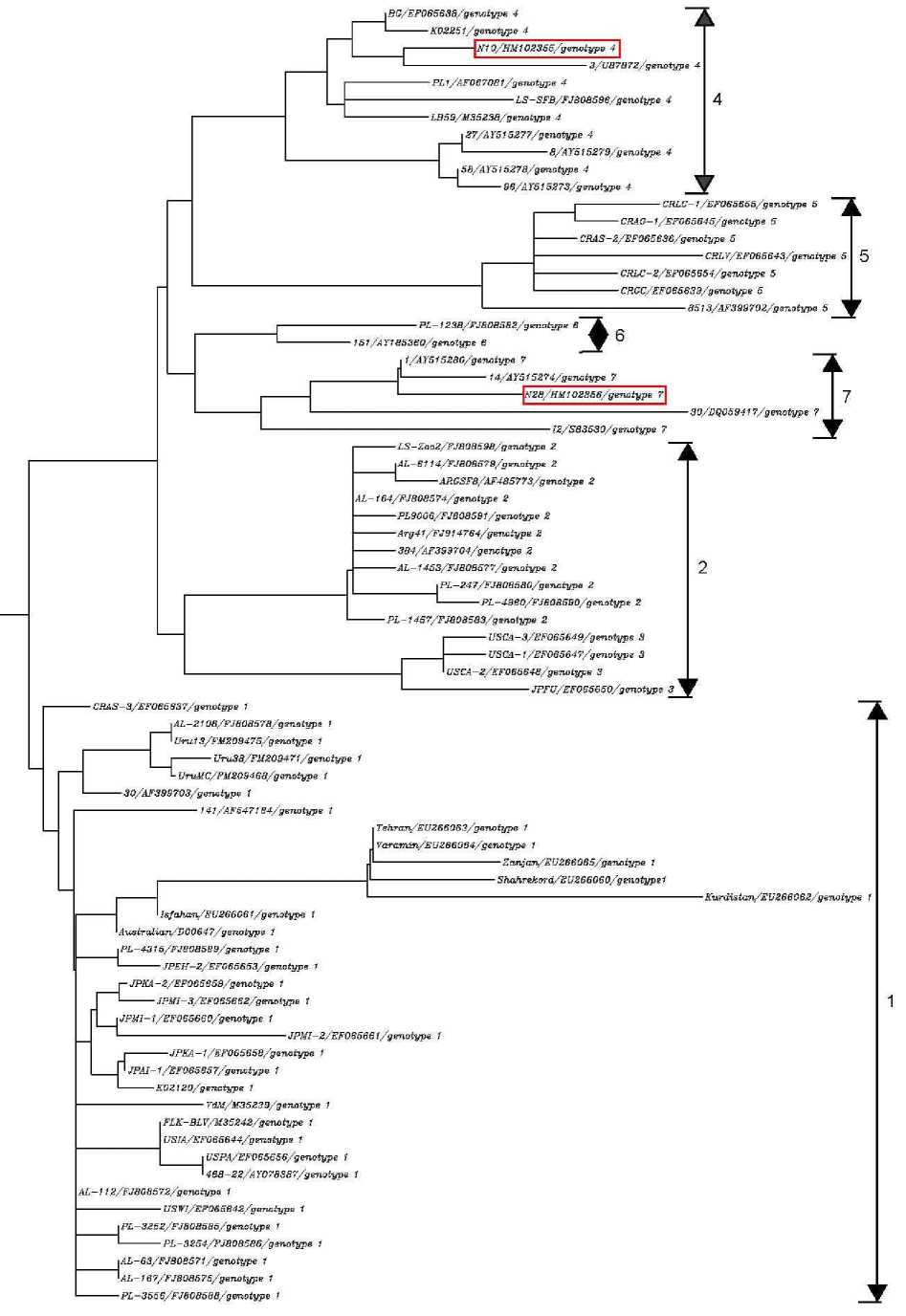
CHLC-VE^085655/gBTtofrype 5 j
JPFU/EFO6565O/g
■ Knr^vaton/EU 286062/genotype 1
-------CRAG—l/EFO65845/genot‘ype 5
CRAS-2/EF065636/genotype 5
---------------------CRLV/EF065643/geTiotype 5
------CRLC-2/EF065654/geTwtype 5
-------CRGC/EF065839/g enotyp e 5
7агат1п/ЕЦ2ббОб4/депо£уре 1
----------------------Еапзст/£и266й85/детго£уре 1
---------------------ShaArekiyrd/EU266060/genotypel
-------- ' Isfah>a.'n/EU26606f/g • AiustTalia7i/D00647/ge,Rotype 1 iPL—4315/FJ80B583/geTLotyye 1 ।-------JPEH-2/EFO65653/g®-raotype 1 ___^JPKA-2/EF0e5e59/genotype 1 | *------JPMI-3/EF065662/genotype Г I___tJPMI-t/EF065660/genotype 1 __ ।-----------------------------JPMI-2/EF065661 /genotype 1 j------JPKA-1/EFO6565B/genotype 1 ____П JPAI-l/EF065657/geiwtype 1 *------KQ 2120/genotype 1 ------------------------YdM/MS 52 ЗЭ/д antitype 1 FLK-BLV/M35Z42/gcmtype 1 USIA/EFO65644/genotype 1 ______I USPA/EF065656/genotype 1 I 468-22/A У075387/депо^уре 1 AL—1 12/FJB08572/genotype 1 --------------USWl/EF0e5e42/genofrype 1 ______iFL—3252/FJ8QB5a5/genetype 1 ।-------PL-3£54/FJ808586/gantitype 1 ______iAL-63/FJ808571/genotype 1 ML-?67/W80S575/ye notice 1 -------PL-3556/FJ8O8588/genotype 1 — CRAS-3/EFO65S37/ge-notype 1 ___I AL—2106/FJ808578/gantitype 1 | I Uru.13/FM209475/g antitype 1 | ।------Uru38/FM20947Vgenotype 1 ' L UruAIC/FM2O9468/genotype t ------30/AF399703/genQtype I ----------------------f 41/AF547184/genotype 1 -----------------------------------I2/S83530/genotyye 7 ।JL - 6114/FJB0B579/g enotype 2 *-------APGSF8/AF485773/gentitype 2 AL-t64/FJ808574/genotype 2 -------PL9006/FJ808591/genotype 2 -------Arg41 /FJ914764/yenatype 2 -------3B4/AF3997Q4/genot-ype 2 -------AL-1453/FJBQ8577/gB7i.oiype 2 >PL—247/FJB085BO/ge7iatypB 2 " I-------PL-4960/FJ808590/gentitype 2 ------PL-1457/FJ8085a3/genntype 2 j-------US CM—3/EF065649/g enotype S ____________USCA-t/EF065647/geno^pe 3 --------- । USCA-2/EFO65648/genotyp8 3 -----------PL-123B/FJ808583/g enofryp e в 15VAY18536Q/gentit-ype 6 ^ (i/AY5-f5280/genoiype 7 ----------------14/AY515274/geTbQiype 7 ----------------------------------------------------------30/EQ059417/genotype 7 iBQ/EF06563a/gerz)iype 4 *-------K02251/genotype 4 I-------[AW^2£ffff/5^53 I-------------------------------3/UB7872/genotype 4 -------PL 1 /AFO670B1 /g enotyp в 4 ----------------------LS-SFB/FJ8085d6/getu>type 4 -------LB59/M35238/geTiotype 4 ___|г7/аГ5?5^77/депо^е 4 __________I I----------------8/AY515279/genotype 4 | 15fl/kK515278/genotype 4 _ ।--------9 6/AY615273/genotype 4 4 3. Дендрограмма типовых представителей известных генотипов BLV (env-ген) [CLUSTAL W (v. 1.83); алгоритм NJ] По результатам филогенетического анализа выравненных нуклеотидных последовательностей локуса env-гена типовых представителей известных генотипов ВЛРКС установлено, что изолят «N10» принадлежит к кластеру 4-го генотипа, а изолят «N28» – 7-го генотипа BLV (рис. 3). Заключение. Системный мониторинг инфицированности стад ВЛКРС на генотипическом уровне идентификации возбудителя будет способствовать повышению эффективности противолейкозных мероприятий. ЛИТЕРАТУРА: 1. Beier, D. Identification of different BLV provirus isolates by PCR, RFLPA and DNA sequencing / D. Beier, P. Blankenstein, O. Marquardt, J. Kuzmak // Berl Munch Tierarztl Wochenschr – 2001. – V. 114. – № 7-8. – P. 252-256. 2. Fechner, H. Provirus variants of the bovine leukemia virus and their relation to the serological status of naturally infected cattle / H. Fechner, P. Blankenstein, A.C. Looman, J. Elwert, L. Geue, C. Albrecht, A. Kurg, D. Beier, O. Marquardt, D. Ebner // Virology –1997. – V. 237. – № 2. – P. 261-269. 3. Licursi, M. Genetic heterogeneity among bovine leukemia virus genotypes and its relation to humoral responses in hosts / M. Licursi, Y. Inoshima, D. Wu, T. Yokoyama, E.T. González, H. Sentsui // Virus Res. – 2002. – V. 86. – № 1-2. – P. 101-110. 4. Moratorio, G. Phylogenetic analysis of bovine leukemia viruses isolated in South America reveals diversification in seven distinct genotypes / G. Moratorio, G. Obal, A. Dubra, A. Correa, S. Bianchi, A. Buschiazzo, J. Cristina, O. Pritsch // Arch. Virol. – 2010. – V. 155. – № 4. – P. 481-489. 5. Rodriguez, S.M. Bovine leukemia virus can be classified into seven genotypes: evidence for the existence of two novel clades / S.M. Rodriguez, M.D. Golemba, R.H. Campos, K. Trono, L.R. Jones //J Gen Virol. – 2009. – V. 90. – № 11. – P. 2788-2797. МОЛЕКУЛЯРНО-ГЕНЕТИЧЕСКИЕ АСПЕКТЫ ТИПИЗАЦИИ ВИРУСА ЛЕЙКОЗА КРУПНОГО РОГАТОГО СКОТА Шаева А.Ю., Вафин Р.Р., Хазипов Н.З., Камалов Б.В., Алимов А.М., Тагиров М.Ш. Резюме Целью нашего исследования являлось определение таксономической принадлежности изолятов ВЛКРС, выделенных в хозяйствах РТ, в контексте молекулярно-генетических аспектов типизации BLV. Системный мониторинг инфицированности стад ВЛКРС на генотипическом уровне идентификации возбудителя будет способствовать повышению эффективности противолейкозных мероприятий. MOLECULAR-GENETIC ASPECTS OF BOVINE LEUKEMIA VIRUS TYPING Shaeva A.Y., Vafin R.R., Khazipov N.Z., Kamalov B.V., Alimov A.M., Tagirov M.Sh.


