Молекулярно-генетический анализ геномов вируса парагриппа-3 крупного рогатого скота
Автор: Галиуллин А.К., Гумеров В.Г., Гильмутдинов Р.Я., Шаева А.Ю., Гериш Ашуак, Магдеева Э.А., Софронов П.В.
Статья в выпуске: 3 т.255, 2023 года.
Бесплатный доступ
В статье представлена актуальность проблемы парагриппа-3 (111 -3) крупного рогатого скота для ветеринарной службы, в том числе распространенность возбудителя в различных регионах Российской Федерации. Показана степень изученности вопроса генотипирования ПГ-3 крупного рогатого скота в мире. Продемонстрированы перспективы молекулярногенетического анализа геномов вируса с целью более точной его идентификации и разработки соответствующих вакцин с повышенными иммуногенными свойствами. Авторы впервые в Российской Федерации провели генотипирование вируса ПГ-3 крупного рогатого скота в животноводческих хозяйствах Республики Татарстан.
Парагрипп-3, вирус парагриппа-3, крупный рогатый скот, генотип, филогенетический анализ
Короткий адрес: https://sciup.org/142238931
IDR: 142238931 | УДК: 619:616.9:616.24 | DOI: 10.31588/2413_4201_1883_2_255_92
Текст научной статьи Молекулярно-генетический анализ геномов вируса парагриппа-3 крупного рогатого скота
Парагрипп-3 (ПГ-3) крупного рогатого скота - острая контагиозная вирусная болезнь, характеризующаяся поражением органов дыхания у животного. Впервые заболевание описано в США Форлеем и Скотом в 1932 году, а вирус, являющийся РНК-содержащим представителем семейства Paramixoviridae, впервые выделен Рейзенгером в 1959 году. Взаимодействие вируса с животным организмом может протекать в виде как моноинфекции, так и смешанных вирусных, вирусно-бактериальных инфекций, которые наносят значительный экономический ущерб из-за гибели и выбраковки животных, снижения их продуктивности и нарушения воспроизводства.
Об актуальности проблемы для Российской Федерации свидетельствует регулярной обновление Министерством сельского хозяйства приказов, связанных с осуществлением ветеринарных мероприятий, направленных на предотвращение распространения и ликвидацию очагов ПГ-3. Так, например, Приказом № 659 от 24 сентября 2021 г. был заменен соответствующий Приказ № 334 от 17 июня 2019 г.
Проанализировано распространение ПГ-3 крупного рогатого скота в животноводческих хозяйствах Восточной Сибири (Строганова И.Я., 2011), Краснодарскаого края (Гречаный В.С., 2011), Свердловской области (Петрова О.Г., Барашкин М.И., 2014), достигавшее в отдельные годы до 29 %.
В Свердловской области ежегодно переболевают, согласно официальным данным ветеринарной отчетности по болезням легких, в среднем более 85 тыс. голов молодняка крупного рогатого скота. За 2002-2012 гг. было зарегистрировано 6897 случаев болезней легких с диагнозом инфекционной патологии, а по диагностическим материалам 857 случаев. Из этого количества 3,36 % принадлежит ПГ-3 (Петрова О.Г., Барашкин М.И., 2014).
Целью данного исследования был молекулярно-генетический анализ парагриппа-3 крупного рогатого скота, выделяемого в различных регионах РФ.
Материал и методы исследований. В работе представлены материалы вирусологического исследования вируса парагриппа-3 крупного рогатого скота на территории РТ. Вирусный изолят ЛД-9 выделен из патологического материала от больного теленка из Лаишевского района РТ (на основании изучения физикохимических и биологических свойств идентифицирован как вирус ПГ-3), инфекционная активность на перевиваемой линии культуры клеток ЛЭК – 6,25 lg ТЦД 50/мл.
Выделение нуклеиновой кислоты (РНК) вируса было проведено из образцов тканей (носовая слизь, легочная ткань и лимфатические узлы) с использованием коммерческого набора РИБО-преп (AmpliSens, Россия), согласно инструкции производителя. Все выделенные пробы перед тестированием хранили при температуре минус 20 °C. Вирусная РНК подвергалась обратной транскрипции с помощью комплекта REVERТA-L (AmpliSens, Россия) в соответствии с инструкцией изготовителя. Образцы ДНК изолята ЛД-9, вакцинного штамма ПТК-45/86 и референтного штамма SF-4 были исследованы методом ПЦР со специфическими праймерами, кодирующими фрагмент матричного гена (М гена) вируса. В таблице 1 указаны область и размеры ампликона. Амплификацию проводили в твердотельном амплификаторе фирмы «Терцик» ДНК-технология (Россия).
Результат исследований.
Идентификацию патогена проводят в основном с использованием серологических реакций: РТГАд (наиболее быстрый), РИФ, РТГА, PH, ИФА. Серологическими исследованиями, проведенными в различных странах мира и нашей стране, антитела к вирусу ПГ-3 обнаружены у 95-97 % обследованных животных (Гусев А.А. и др., 2002). Высокий уровень серопозитивности к BPIV3 у молочного скота указывает на высокий уровень заражения BPIV3. Между тем, большие возможности в изучении патогена открывает использование методов генотипирования. Так, многофакторный эпидемиологический анализ заболеваемости парагриппом-3 включает генотипирование всех изолятов вируса. Серологически вирус может быть монотипическим, а по анализу последовательностей РНК изолятов – возможно существование нескольких его вариантов, циркулирующих на определенных территориях (Думова В.В. и соавт., 2012). Кроме того, генотипирование имееет большие перспективы при разработке соответствующих вакцин, поскольку отмечается низкая иммуногенность известных вакцин, изготовленных на основе штаммов VR-ATCC-739, SF-4, ПТК-45/86 вируса bPIV3 и др. В результате множественного выравнивания 17 нуклеотидных последовательностей, депонированных в GenBank, с последовательностями, полученными нами, российский изолят ЛД-9 показал 98,3 % идентичность с референтным штаммом SF-4, относящимся к генотипу А, тогда как российский вакцинный штамм ПТК 45/86 был на 100 % идентичен референтному штамму SF-4 (Таблица 1).
Нуклеотидная последовательность фрагмента М-гена изолята ЛД-9 вируса ПГ-3 была размещена в базе данных GenBank под регистрационным номером MW524841. Результаты филогенетического анализа показали, что российский изолят ЛД-9 (MW52481) и вакцинный штамм ПTK45/86 относятся к генотипу А вируса парагриппа-3, как и референтный штамм SF-4 (Рисунок 1).
Современные варианты генотипирования включают идентификацию полиморфизма длины рестриционного фрагмента геномной ДНК (RFLPI), случайную амплифицированную полиморфную детекцию геномной ДНК (RAPD), детекцию полиморфизма длины амплифицированного фрагмента (AFLPD), полимеразную цепную реакцию (ПЦР), спеквенирование ДНК, аллельспецифичные олигонуклеотидные зонды (ASO) и гиборидизацию с ДНК-микрочипами или шариками. Генотипирование важно при исследовании генов и их вариантов, связанных с заболеванием. Из-за современных технологических ограничений почти все генотипирование является частичным. То есть определяется только небольшая часть генотипа индивидуума, например, с помощью (epi) GBS (генотипирование и путем секвенирования) или RADseq. Новые технологии массового секвенирования обещают обеспечить полногеномное генотипирование в будущем.
Таблица 1 – Сравнительный анализ идентичности в нуклеотидных последовательностях области М гена (328 п.н.) изолята ЛД-9, вакцинного штамма ПTK45/86 и 17 штаммов вируса ПГ-3 крупного рогатого скота
|
Штаммы |
Идентичность, % |
||
|
SF-4 |
ПТК45/86 |
ЛД-9 |
|
|
ПТК45/86 |
100 |
/ |
98,25 |
|
ЛД-9 |
98,3 |
98,25 |
/ |
|
SF-4 |
/ |
100 |
98,3 |
|
NM09 |
94,3 |
94,3 |
92,98 |
|
JCU |
92,5 |
92,5 |
90,8 |
|
BN-1 |
93,42 |
93,42 |
91,67 |
|
ISU* |
91,67 |
91,67 |
89,91 |
|
910N |
93,42 |
93,42 |
91,67 |
|
Kаnsas* |
99,12 |
99,12 |
97,35 |
|
BP4158 |
87,28 |
87,28 |
85,53 |
|
Texas |
100 |
100 |
98,25 |
|
TVMDL15 |
86,84 |
86,84 |
85,09 |
|
SD0835 |
87,72 |
87,72 |
86,4 |
|
NX49 |
87,72 |
87,72 |
84,4 |
|
TVMDL20 |
87,28 |
87,28 |
85,96 |
|
HS9 |
88,16 |
88,16 |
86,84 |
|
12Q061 |
87,28 |
87,28 |
85,96 |
|
LE017 |
99,12 |
99,12 |
97,37 |
|
GP** |
81,58 |
81,58 |
81,4 |
Примечание: *swine parainfluenza virus type 3 (SPIV-3), **Human parainfluenza type 3 (HPIV-3)
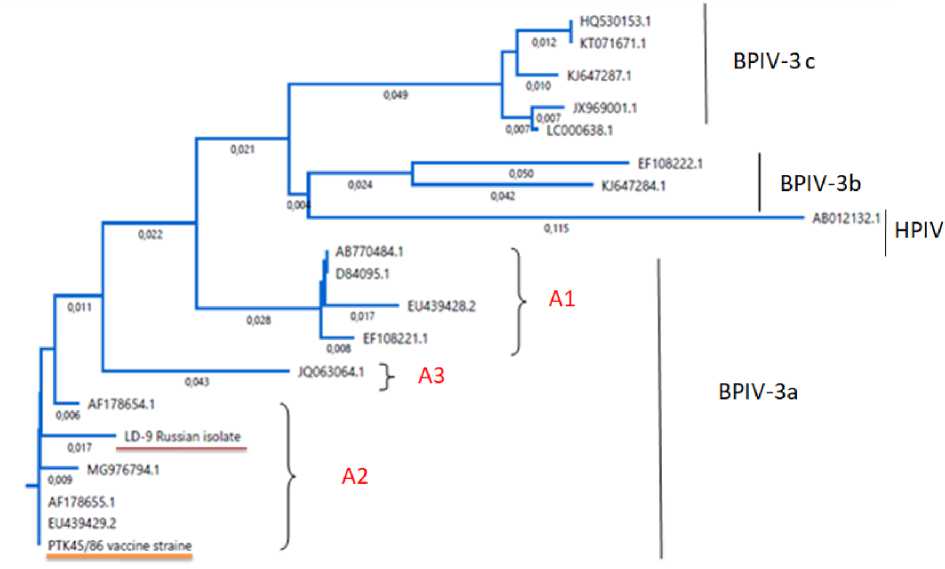
Рисунок 1 – Филогенетический анализ нуклеотидных последовательностей изолятов вируса ПГ-3 на основе фрагмента М гена
На сегодня с использованием генетического и филогенетического анализа описаны три генотипа bPIV3: A или bBPIV-3a (классический), B или bBPIV-3b и C или bPIV-3c. Генотип A впервые был выделен в США, а позже - в Египте, Китае и Японии. Генотип B впервые описан в Австралии. Выделение генотипа C проведено в Китае, Южной Корее и Японии. Между тем, все три генотипа зарегистрированы в Аргентине (Neill J. et al., 2015). Между тем, в целом исследования этих генотипов и инфекций, связанных с ними, в том числе в условиях эксперимента, достаточно ограничены.
BPIV-3 является РНК-вирусом с высокой генетической изменчивостью, тем не менее, вклад событий рекомбинации в его изменчивость до сих пор не оценен. T. Alatorre-García et al. (2021) проанализировали 25 полных последовательностей генома (CGS), о которых сообщалось на данный момент, и 215 частичных последовательностей различных вирусных генов BPIV-3 для определения их генотипов и субгенотипов, распределения и существования потенциальных событий рекомбинации. На основе анализа генов HN, M, N и P был обнаружен один гипотетический субгенотип (субгенотип A4). Четыре события рекомбинации между последовательностями свиней и крупного рогатого скота были обнаружены с помощью анализа RDP4 в сочетании с филогенетическими несоответствиями в гене L. Кроме того, было обнаружено, что 9 последовательностей, о которых сообщалось из Аргентины, были классифицированы как пропущенные. Эти результаты показывают, что события гомологичной рекомбинации играют важную роль в эволюции BPIV-3, и подчеркивают важность внедрения усовершенствованной молекулярной характеристики для лучшего понимания изменчивости и эволюции BPIV-3 как компонента BRDC.
В Турции M. Timurkan et al. (2019) осуществили молекулярный анализ bPIV-3, в частности, усовершенствовал метод секвенирования Сэнгера. В усовершенствованном варианте они использовали матричный ген (М ген) для BPIV-3 (328 bp). Образцы были протестированы методом ПЦР с использованием пар геноспецифичных праймеров. Были использованы праймеры, специфичные для матричного гена bPIV3.
H. Albayrak et al. (2019), продолжая исследования M. Timurkan et al., для секвенирования генома BPIV-3 использовал метод нового поколения Illumina с использованием гликопротеина (F) размером 400 bp. Филогенетический анализ первого изолята из образца легкого умершего теленка показал, что генотип С (номер доступа в GenBank MH357343) обнаруживается и в других регионах, который до сих пор описывался, как циркулирующий только в части Азии и Южной Америки. Дальнейшие исследования были направлены на определение молекулярной характеристики изолятов вирусов ПГ-3 из носовых слизей коров в районе Эрзурум на востоке Турции. Всего было изолировано 155 образцов: 92 пробы от быков и 63 – коров. Возраст животных составлял от 9 месяцев до 5 лет. Большинство быков были на откорме и содержались в открытых помещениях; коровы находились в период лактации и содержались в свободных стойлах. Все отобранные образцы были проверены на наличие вирусного генома с использованием ПЦР. Ген-специфические праймеры в молекулярном методе идентифицировали штаммы ПГ-3 (М-ген) на уровне рода. В результате РНК из ПГ-3 был обнаружен в трех (1,93 %) образцах, соответственно, один из которых был секвенирован, и последовательности были сопоставлены с эталонными штаммами вируса. Филогенетический анализ позволил отнести вирус ПГ-3 к генотипу С.
N. Sobhy et al. (2017) выделил BPIV-3 из носовой слизи египетского крупного рогатого скота с легкой формой пневмонии. Вирус, выделенный в клетках MBDK, был подтвержден методом ОТ ПЦР. Полный геном египетского штамма BPIV-3 был секвенирован с использованием метода Illumina.
В 2008 году четыре штамма BPIV3
были выделены с использованием клеток MDBK крупного рогатого скота в Китае и охарактеризованы с помощью ОТ-ПЦР, анализа нуклеотидной последовательности, наблюдения под просвечивающим электронным микроскопом, тестов на гемадсорбцию и гемагглютинацию. Нуклеотидный филогенетический анализ частичного гена гемагглютинин-нейраминидазы (HN) для четырех изолятов и полного генома для изолята SD0835 показал, что четыре китайских штамма BPIV3 отличаются от ранее зарегистрированных генотипов A (BPIV3a) и B (BPIV3b) и могут быть потенциально новым генотипом, который был предварительно классифицирован как генотип C (BPIV3c). Y. Zhu et al. (2011) сделали первое сообщение о выделении и генетической характеристике BPIV3 у крупного рогатого скота в Китае. Нуклеотидный филогенетический анализ частичного гена гемагглютинин-нейраминидазы (HN) для четырех изолятов и полного генома для изолята SD0835 показал, что четыре китайских штамма BPIV3 отличаются от ранее зарегистрированных генотипа A (BPIV3a) и генотипа B (BPIV3b) и могут быть потенциально новым генотипом, который был предварительно классифицирован как генотип C (BPIV3c).
BPIV3, выделенный в 2009 году с использованием культуры клеток MDBK из мазков из носа нормального крупного рогатого скота в Китае и названный NM09, был охарактеризован Y. Wen et al. (2012) с помощью ОТ-ПЦР и анализа нуклеотидной последовательности. Длина его полного генома составляла 15 456 нуклеотидов. Подобно другим секвенированным PIV-штаммам, вирус NM09 состоял из шести непересекающихся генов, которые, как было предсказано, кодируют девять белков с консервативными и комплементарными 3'-лидерными и 5'-концевыми участками, консервативными генными стартами, генными остановками и тринуклеотидными межгенными последовательностями. Филогенетический анализ нуклеотидов матрикса и гена гемагглютинин-нейраминидазы показал, что этот изолят
NM09 принадлежал к генотипу BPIV3 A вместо ранее зарегистрированного в Китае генотипа BPIV3 C. Предполагается, что различные генотипы A и C могут сосуществовать в Китае в течение длительного времени.
Позже É. Leal et al. (2019) выделили в Китае новый генотип bPIV-3 и назвали его bPIV-3 - BJ. Филогенетический анализ показал, что изолят родственен BPIV3 генотипа A. Показано значительное различие между BPIV3-BJ и эталонным китайским изолятом NM09. Обнаружено много различий в аминокислотном составе белка нуклеокапсида (NP) у этих штаммов генотипа А. Поскольку NP вовлечен в исследования по иммунизации, изолят BPIV3 будет полезен для разработки иммунных анализов и исследований вакцин. Обнаруженное в Китае разнообразие линий BPIV3 указывает на продолжающуюся эволюцию иммунного ответа. Авторы подчеркивают важность генетического надзора для определения влияния вариабельности BPIV3 на эволюцию патогена и популяционный иммунитет.
-
A. Kumagai et al. (2020) изучили генетические и антигенные характеристики BPIV3, циркулирующего в Японии. Филогенетический анализ генов фосфопротеина и гемагглютинин-нейраминидазы показал, что изоляты сгруппировались в два генотипа: BPIV3a (49 %) и BPIV3c (51 %). Штаммы BPIV3a имели более широкую генетическую вариабельность, чем остальные генотипы. Кроме того, были получены новые варианты, и их предварительно обозначили как подгруппу 4 BPIV3a. Первый японский штамм BPIV3c был выделен в 2012 году, но здесь штамм BPIV3c NM2 был выделен из образца, собранного на четыре года раньше, чем в предыдущем отчете. Антигенность десяти штаммов BPIV3, включающих все три генотипа, оценивали с помощью теста на перекрестную нейтрализацию вирусов.
-
R. Vaucher et al. (2019) в Бразилии амплифицировали методом ОТ-ПЦР, секвенировали с использованием специфических дегенеративных праймеров
и проанализировали путем филогенетического сравнения с эталонными штаммами bPI3V геномные фрагменты генов HN и L вируса bPIV-3, выделенные в качестве контаминантов из клеточных культур и клинических образцов, были амплифицированы с помощью ОТ ПЦР. Секвенированные бразильские изоляты показали высокую степень геномного сходства по сравнению со штаммом-прототипом SF4/32 в рамках недавно предложенного генотипа A bPIV-3.
У нас же в стране исследования по молекулярно-генетическому анализу геномов (генотипированию) вируса парагриппа-3 крупного рогатого скота только начинаются. Первые данные получены по Республике Татарстан (Гериш Ашуак, 2021). Генетическая идентификация выделенного изолята ЛД-9 вируса парагриппа 3 крупного рогатого скота проведена на основе секвенирования фрагментов М гена возбудителя. Показана принадлежность изолята ЛД-9 и вакцинного штамма ПТК-45/86 вируса bPIV3 к одному и тому же генотипу А и к тому же кластеру, что и эталонный штамм SF-4.
Заключение. Таким образом, исходя из анализа основных достоинств и недостатков, приоритетным методом диагностики респираторных вирусных болезней является ПЦР, в частности ОТ-ПЦР, для быстрого выявления и профилактики болезни молодняка крупного рогатого скота. Матричный белок вируса парагриппа-3 играет важную роль в сборке вирионов, установлении персистирующей инфекции и патогенезе вируса. Ген (М-ген), кодирующий этот белок, является подходящей мишенью для создания информативной филогенетической реконструкции вируса парагриппа-3 крупного рогатого скота.
Вакцины, используемые в России против вируса ПК-3 крупного рогатого скота, были разработаны на основе референтного штамма, принадлежащего генотипу А. Перекрестная защита таких вакцин от ПГ-3, вызванного генотипами В и С, остается неясной, поэтому следует оценить необходимость разработки альтернативных диагностических тестов и вакцин для профилактики. Генетическая характеристика циркулирующих штаммов возбудителя может способствовать разработке более эффективных методов диагностики и профилактики данной инфекции.
предотвращение распространения и ликвидацию очагов парагриппа-3"
Резюме
В статье представлена актуальность проблемы парагриппа-3 (ПГ-3) крупного рогатого скота для ветеринарной службы, в том числе распространенность возбудителя в различных регионах Российской Федерации. Показана степень изученности вопроса генотипирования ПГ-3 крупного рогатого скота в мире. Продемонстрированы перспективы молекулярногенетического анализа геномов вируса с целью более точной его идентификации и разработки соответствующих вакцин с повышенными иммуногенными свойствами. Авторы впервые в Российской Федерации провели генотипирование вируса ПГ-3 крупного рогатого скота в животноводческих хозяйствах Республики Татарстан.
MOLECULAR GENETIC ANALYSIS OF GENOMES OF BOVINE PARAINFLUENZA-3
Список литературы Молекулярно-генетический анализ геномов вируса парагриппа-3 крупного рогатого скота
- Гериш, А. Генотипизация вируса парагриппа-3 крупного рогатого скота, выделенного в Республике Татарстан / А. Гериш. – Дисс. … канд. вет. наук – Казань, 2021.
- Гречаный, В. С. Морфофункциональная характеристика и терапия бронхопневмоний, вызванных ассоциацией вируса парагриппа-3, Mycoplasma bovis и haemophilus somnus, у телят / В. С. Гречаный / Дис. … канд. вет. наук. - пос. Персиановский, 2011. – 180 с.
- Госманов, Р. Г. Иммунология / Р. Г. Госманов, Н. М. Колычев, Р. Х. Равилов [и др.]. – Санкт-Петербург, 2018. – С. 188.
- Гусев, А. А. Штамм вируса парагриппа-3 крупного рогатого скота для изготовления диагностических и вакцинных препаратов / А. А. Гусев, В. А. Мищенко, О. И. Сухарев [и др.] / Патент A61K39/155. – 2002.
- Думова, В. В. Распространение вируса парагриппа-3 крупного рогатого скота у жвачных животных / В. В. Думова, В. А. Мищенко, А. В. Мищенко, М. Ю. Киселев, О. Ю. Черных // Ветеринария Кубани. – 2012. – № 6. – С. 10-12.
- Петрова, О. Г. Острые респираторные заболевания крупного рогатого скота и проблемы профилактики на региональном уровне / О. Г. Петрова, М. И. Барашкин // Аграрный вестник Урала. – 2014. – № 6 (124). – С. 40-42.
- Приказ Министерства сельского хозяйства РФ от 24 сентября 2021г. N 659 "Об утверждении Ветеринарных правил осуществления профилактических, диагностических, лечебных, ограничительных и иных мероприятий, установления и отмены карантина и иных ограничений, направленных на предотвращение распространения и ликвидацию очагов парагриппа-3"
- Приказ Минсельхоза России от 17 июня 2019 г. N 334 "Об утверждении Ветеринарных правил осуществления профилактических, диагностических, лечебных, ограничительных и иных мероприятий, установления и отмены карантина и иных ограничений, направленных на предотвращение распространения и ликвидацию очагов парагриппа-3".
- Равилов, Р. Х. Хламидиоз собак и кошек / Р. Х. Равилов // Сер. Практика ветеринарного врача. Москва, 2006. – 126 c.
- Строганова, И. Я. Распространение парагриппа-3 крупного рогатого скота в животноводческих хозяйствах Восточной Сибири / И. Я. Строганова // Вестник Красноярского государственного аграрного университета. – 2011. – №. 6. – С. 115-117.
- Albayrak, H. Characterisation of the first bovine parainfluenza virus 3 isolate detected in cattle in Turkey / H. Albayrak, Z. Yazici, E. Ozan [et al.] // Vet. Sci. – 2019. – Vol. 6 (2). – P. 56.
- Alatorre-García, T. Homologous recombination as a mechanism of genetic changes in bovine parainfluenza-3 virus / T. Alatorre-García, S. Fonseca- Coronado, F. González-Candelas // Vet. Microbiol. – 2021. – Vol. 261. – P. 109185.
- Kumagai, A. Phylogenetic and antigenic analysis of bovine parainfluenza virus type 3 isolated in Japan between 2002 and 2019 / A. Kumagai, T. Kanno, K. Kawauchi [et al.] // Vet. Microbiol. – 2020. – Vol. 247. – P. 108774.
- Leal, É. Isolation of a divergent strain of bovine parainfluenza virus type 3 (BPIV3) infecting cattle in China / É. Leal, C. Liu, Z. Zhao, Y. Deng [et al.] // Viruses. – 2019. – Vol. 11 (6). – P. 489.
- Neill, J. Identification and genome characterization of genotype B and genotype C of bovine parainfluenza type 3 viruses isolated in the United States / J. Neill, J. F. Ridpath, B. T. Valayudhan // BMC Vet. Res. – 2015. – Vol. 11. – P. 112.
- Sobhy, N. Surveillance, isolation and complete genome sequence of bovine parainfluenza virus type 3 in Egyptian cattle / N. Sobhy, S. Mor, I. Bastawecy [et al.] // Intern. J. Vet. Sci. Med. – 2017. – Vol. 5 (1). – P. 8-13.
- Timurkan M. Identification and molecular characterisation of bovine parainfluenza virus-3 and bovine respiratory syncytial virus-first report from Turkey / M. Timurkan, H. Aydin, A. Sait // J. Vet. Res. – 2019. – Vol. 63 (2). – P. 167.


