Молекулярно-генетический анализ популяций Pinus sylvestris L. и Pinus sibirica du Tour в Пермском крае на основании полиморфизма ISSR-PCR маркеров
Автор: Нечаева Юлия Игоревна, Пыстогова Нина Александровна, Чертов Никита Валерьевич, Боронникова Светлана Витальевна
Журнал: Бюллетень науки и практики @bulletennauki
Рубрика: Биологические науки
Статья в выпуске: 4 т.7, 2021 года.
Бесплатный доступ
Изучен полиморфизм ДНК, определены показатели генетического разнообразия и генетической структуры 3 популяций Pinus sylvestris L. и 3 популяций Pinus sibirica Du Tour в Пермском крае. В популяциях P. sibirica обнаружено 102 ISSR-PCR маркера, из которых 88 были полиморфными ( P95 = 0,863), а в популяциях P. sylvestris - 113 ISSR-PCR маркеров, при этом 100 из них являлись полиморфными ( P95 = 0,885). Популяции двух исследуемых видов древесных растений характеризуются высоким генетическим разнообразием. При этом у P. sibirica показатели генетического разнообразия оказались незначительно выше ( HE = 0,195; ne = 1,335; na = 1,330), чем у P. sylvestris ( HE = 0,166; ne = 1,268; na = 1,212). Анализ генетической структуры показал, что коэффициенты генетической подразделенности ( GST ) у двух изученных видов рода Pinus близки и составляют 0,320 у P. sibirica и 0,303 у P. sylvestris. Популяции сосны сибирской и сосны обыкновенной характеризуются средней степенью генетической дифференциации, поскольку на долю межпопуляционной компоненты приходится 32,0% и 30,3% генетического разнообразия этих видов соответственно. С помощью теста Мантела установлена высокая корреляция (R2 = 0,6871) между генетическими и географическими расстояниями у популяций P. sibirica . Полученные данные актуальны для сохранения генофондов изученных двух видов рода Pinus в Пермском крае.
Генетическое разнообразие, генетическая структура, issr-pcr маркеры, пермский край
Короткий адрес: https://sciup.org/14120934
IDR: 14120934 | УДК: 575.2:575.22:574.3 | DOI: 10.33619/2414-2948/65/01
Текст научной статьи Молекулярно-генетический анализ популяций Pinus sylvestris L. и Pinus sibirica du Tour в Пермском крае на основании полиморфизма ISSR-PCR маркеров
Бюллетень науки и практики / Bulletin of Science and Practice
УДК 575.2:575.22:574.3
Генетическое разнообразие и внутривидовая дифференциация имеют важное биосферное и ресурсное значение. Для решения современных проблем сохранения и возобновления лесов необходима оценка биоразнообразия лесных экосистем, важным элементом которой является изучение генетического разнообразия популяций основных лесообразующих видов растений [1]. Разработка и обоснование комплекса мероприятий, направленных на максимальное сохранение генетического разнообразия лесообразующих видов в различных условиях должна основываться на данных о генетической структуре и состоянии генофондов их популяций [2].
Сосна сибирская ( Pinus sibirica Du Tour) и сосна обыкновенная ( Pinus sylvestris L.) являются ценными хозяйственными видами, а также одними из основных эдификаторов лесных экосистем бореальной зоны Евразии. Высокая экологическая пластичность и хозяйственная ценность сосны сибирской и сосны обыкновенной давно привлекают внимание генетиков и селекционеров к проблеме изучения, сохранения и воспроизводства генофондов этих видов.
Цель работы — сравнительный анализ генетического разнообразия и генетической структуры популяций P. sylvestris и P. sibirica в Пермском крае на основании полиморфизма межмикросателлитных маркеров.
Материалы и методы исследований
В качестве объектов для сравнительного анализа генетического разнообразия и генетической структуры избраны популяции двух видов древесных растений (Pinus sylvestris L. и Pinus sibirica Du Tour; Pinaceae), расположенные в центральной и северной частях Пермского края. Исследованы 3 популяции сосны сибирской (P. sibirica), расположенные в Красновишерском лесничестве (Ps_Kr), Кочевском лесничестве (Ps_Kh) и на территории ФГБУ «Государственный заповедник «Басеги»» (Ps_Bs) (Таблица 1). Среди изученных популяций сосны сибирской на наибольшем географическом расстоянии (268 км) находятся популяции Ps_Kh и Ps_Bs, а на наименьшем — Ps_Kr и Ps_Bs (176 км). Исследованные три популяции P. sylvestris находятся на территории Березниковского лесничества (Psl_Br), Закамского лесничества (Psl_Zc) и Кишертского (Psl_Ks) лесничества (Таблица 1). При этом наиболее географически удаленными являются популяции Psl_Br и Psl_Ks (260 км), а на наименьшем географическом расстоянии находятся популяции Psl_Ks и Psl_Zc (105 км).
Таблица 1.
ИЗУЧЕННЫЕ ПОПУЛЯЦИИ P. SIBIRICA И P. SYLVESTRIS
|
Обозначение популяций |
Расположение популяций |
Объем выборки, шт. |
Координаты (с. ш.; в. д.) |
|
Ps_Kr |
Красновишерский район, Красновишерское лесничество |
17 |
N: 60.11 E: 57.44 |
|
Ps_Kh |
Кочевский район, Кочевское лесничество |
30 |
N: 59.39 E: 54.39 |
|
Ps_Bs |
Горнозаводский район, Государственный заповедник «Басеги», Горнозаводское лесничество |
29 |
N: 58.56 E: 58.30 |
|
Psl_Ks |
Кишертский район, Кишертское лесничество |
28 |
N:57.08 E:57.23 |
|
Psl_Br |
г. Березники, Березниковское лесничество |
28 |
N:59.40 E:56.70 |
|
Psl_Zc |
Пермский район, Закамское лесничество, |
28 |
N:57.96 E:56.16 |
Примечание : популяции P. sibirica : Ps_Kr — расположена в Красновишерском лесничестве; Ps_Kh — в Кочевском лесничестве; Ps_Bs — в Горнозаводском лесничестве; популяции P. sylvestris: Psl_Ks — из Кишертского лесничества; Psl_Br — из Березниковского лесничества; Psl_Zc — из Закамского лесничества; с. ш. — северная широта; в. д. — восточная долгота.
Для проведения молекулярно-генетических исследований были собраны образцы хвои в 6 популяциях P. sibirica и P. sylvestris с 106 деревьев. Сбор образцов осуществлялся со случайно выбранных деревьев, расположенных на расстоянии не менее 100 метром друг от друга. ДНК из хвои выделяли по методике С. Роджерса [3], модифицированной с использованием в качестве сорбента PVPP (polyvinylpolypyrrolidone) [4]. Качество и характеристики ДНК определяли на приборе Spectrofotometr TM NanoDrop 2000 (Thermo scientific, USA). Для оценки генетического разнообразия и генетической структура популяций был применен ISSR- (Inter Simple Sequence Repeats) метод анализа полиморфизма ДНК [5]. Для проведения ПЦР были использованы эффективные для P. sibirica ISSR-PCR праймеры, которые были подобраны ранее [6]: ISSR-9 [(ACG)7G]; CR-217 [(GT) 6 GG]; CR-215 [(CA) 6 GT]; M1 [(AC) 8 CG]; X11 [(AGC) 6 G)]. Для ПЦР с пробами ДНК P. sylvestris также использовались ранее подобранные эффективные праймеры [7]: ISSR-1 [(АС) 8 T]; CR-212 [(CT) 8 TG]; CR-215 [(CA) 6 GT]; M27 [(GA) 8 C]; X10 [(AGC) 6 C)].
Для ПЦР использовали реакционную смесь объемом 25 мкл, содержащую: 0,4 мкл Tag-полимеразы; 2,5 мкл стандартного 10× буфера для ПЦР; 0,25 мкл праймера; 2,5 мкл Mg 2+ ;
0,25 мкл dNTP; 5 мкл тотальной ДНК. В качестве отрицательного контроля (К-) для проверки чистоты реактивов к реакционной смеси вместо ДНК добавляли 5 мкл деионизированной воды. Амплификацию проводили в амплификаторе GeneAmp PCR System 9700 (Applied Biosystems, USA) по типичной для ISSR-PCR метода программе: предварительная денатурация 94 °C, 2 мин.; первые пять циклов 94 °С, 20 сек.; t° отжига, 10 сек.; 72 °С, 10 сек.; в последующих тридцати пяти циклах 94°С, 5 сек.; t° отжига, 5 сек.; 72 °С, 5 сек. Последний цикл элонгации длился 2 мин при 72 °С. Температура отжига в зависимости от G/С-состава праймеров варьировала от 52 °С до 64 °С. Продукты амплификации разделяли с помощью электрофореза в 2% агарозном геле в 1× ТВЕ буфере. Гели окрашивали бромистым этидием и фотографировали в проходящем ультрафиолетовом свете в системе Gel-Doc XR (Bio-Rad, USA). Для определения длины фрагментов ДНК использовали маркер молекулярной массы (100 + bp+DNA Ladder; ЗАО «Евроген», Москва). Определение длин фрагментов проводилось с использованием программы QuantityOne в системе гель-документации Gel-Doc XR (Bio-Rad, USA). Изучен полиморфизм 102 ISSR-PCR маркеров в 3 популяциях P. sibirica и 113 ISSR-PCR маркеров в 3 популяциях P. sylvestris.
Компьютерная обработка данных проведена с помощью программы POPGENE 1.31 [8] и с помощью специализированного макроса GenAlEx6 [9] для MS-Excel с определением: доли полиморфных локусов ( P 95 ) [10], абсолютного числа аллелей ( n a ), эффективного числа аллелей ( n e ) [11], ожидаемой гетерозиготности ( H E ) [12]. Анализ генетической структуры проведен в соответствии с методикой М. Нея [13]. Генетическое расстояние между популяциями определяли по формуле M. Нея и В. Ли [14]. Была рассчитана матрица генетических различий, на основании которой невзвешенным парно-групповым методом UPGMA (unweighted pair-group method using arithmetic average) была построена дендрограмма, отражающая степень сходства исследуемых популяций по ISSR-спектрам при помощи компьютерных программ Treecon 1.3b. Для определения корреляции между генетическими и географическими расстояниями был применен общепринятый тест Мантела [15].
Результаты и их обсуждение
В результате молекулярно-генетического анализа популяций P. sibirica выявлено 102 ISSR-PCR маркера, из которых 88 были полиморфными ( P 95 =0,863). В ходе анализа полиморфизма ДНК популяций P. sylvestris было обнаружено 113 ISSR-PCR маркеров (Таблица 2), из которых 100 являлись полиморфными ( P 95 =0,885). Соответственно, доля полиморфных локусов незначительно выше у изученных популяций P. sylvestris.
Число амплифицированных ISSR-PCR маркеров у P. sibirica варьировало в зависимости от праймера от 17 (M1 [(AC) 8 CG]) до 25 (X11 [(AGC) 6 G]). В популяциях P. sylvestris максимальное число ISSR-PCR маркеров, равное 27, выявлено в ПЦР с праймером CR-212 [(CT) 8 TG], а минимальное (20) с праймерами ISSR-1 [(АС) 8 T] и M27 [(GA) 8 C]. Размеры ISSR-PCR маркеров в исследованных популяциях P. sibirica изменялись (Таблица 2) в зависимости от праймера в пределах от 190 п.н. (M1 [(AC) 8 CG]) до 1570 п.н. (CR-217 [(GT) 6 GG]). В популяциях P. sylvestris размеры амплифицированных ISSR-PCR маркеров варьировали в зависимости от праймера от 210 п.н. (X10 [(AGC) 6 C]) до 1400 п.н. (CR-212 [(CT) 8 TG]). Ожидаемая гетерозиготность ( H E ) у P. sibirica составила 0,195, что незначительно превышает значения этого показателя у P. sylvestris (H E = 0,166).
Таблица 2.
ХАРАКТЕРИСТИКА ISSR-PCR-МАРКЕРОВ P. sibirica И P. sylvestris
Однако, эффективное число аллелей ( n e ) значительно (Таблица 3) выше у P. sibirica ( n e = 1,335 ) по сравнению с P. sylvestris ( n e = 1,268).
СРАВНЕНИЕ ПОКАЗАТЕЛЕЙ ГЕНЕТИЧЕСКОГО РАЗНООБРАЗИЯ
ДВУХ ВИДОВ РОДА PINUS
Таблица 3.
|
Выборка |
P 95 |
H E |
n e |
n a |
|
Популяции P. sibirica |
0,863 |
0,195 (0,011) |
1,335 (0,021) |
1,330 (0,048) |
|
Популяции P. sylvestris |
0,885 |
0,166 (0,010) |
1,268 (0,018) |
1,212 (0,051) |
|
Критерий |
Фишера (F) |
Критерий Стьюдента (t) |
||
|
Значение критерия |
F=0,419 |
F=0,477 |
t=2,420 |
t=1,680 |
|
Сравнение с F st или t st |
0,419 < 1,960 |
0,477 < 1,960 |
2,420 > 1,977 |
1,680 < 1,977 |
Примечание: P 95 — доля полиморфных локусов, H E — ожидаемая гетерозиготность; n e — эффективное число аллелей на локус; у H E и n e в скобках даны стандартные отклонения.
Число редких аллелей выше у P. sylvestris (R=29) по сравнению с P. sibirica (R=18). Анализ генетической структуры трех популяций P. sibirica показал (Таблица 4), что ожидаемая доля гетерозиготных генотипов на общую выборку ( H T ) составила 0,287. Этот показатель выше, чем ожидаемая доля гетерозиготных генотипов в отдельной популяции ( H S =0,196). Наибольшая дифференциация популяций сосны сибирской установлена с использованием праймера X-11 [(AGC) 6 G]. Установлено, что доля межпопуляционного генетического разнообразия в общем разнообразии или коэффициент подразделенности ( G ST ) составил 0,320.
У P. sylvestris доля гетерозиготных генотипов на общую выборку ( H T ) составила 0,238, а ожидаемая доля гетерозиготных генотипов в отдельной популяции ( H S =0,196). Наибольшая дифференциация популяций сосны обыкновенной выявлена с использованием праймера CR-215 [(CA) 6 GT]. Коэффициент подразделенности ( G ST ) у P. sylvestris равен 0,303 (Таблица 4).
Таблица 4.
ГЕНЕТИЧЕСКАЯ СТРУКТУРА И ДИФФЕРЕНЦИАЦИЯ
ИЗУЧЕННЫХ ПОПУЛЯЦИЙ ДВУХ ВИДОВ РОДА PINUS
|
ISSR- PCR праймер |
Нуклеотидная последовательность (5'→3') |
H T |
H S |
G ST |
|
P. sibirica |
||||
|
CR-217 |
(GT) 6 GG |
0,312 (0,017) |
0,227 (0,013) |
0,272 |
|
CR-215 |
(CA) 6 GT |
0,260 (0,028) |
0,192 (0,020) |
0,264 |
|
ISSR-9 |
(ACG) 7 G |
0,260 (0,028) |
0,192 (0,020) |
0,264 |
|
M-1 |
(AC) 8 CG |
0,303 (0,031) |
0,231 (0,022) |
0,239 |
|
X-11 |
(AGC) 6 G |
0,274 (0,030) |
0,167 (0,017) |
0,390 |
|
Среднее |
0,287 (0,027) |
0,196 (0,018) |
0,320 |
|
|
P. sylvestris |
||||
|
ISSR-1 |
(АС) 8 T |
0,281 (0,028) |
0,171 (0,010) |
0,391 |
|
CR-212 |
(CT) 8 TG |
0,261 (0,020) |
0,211 (0,015) |
0,190 |
|
CR-215 |
(CA) 6 GT |
0,251 (0,027) |
0,156 (0,009) |
0,379 |
|
M27 |
(GA) 8 C |
0,177(0,022) |
0,133 (0,010) |
0,248 |
|
X10 |
(AGC) 6 C |
0,209(0,021) |
0,145 (0,007) |
0,307 |
|
Среднее |
0,238(0,024) |
0,166 (0,011) |
0,303 |
|
Примечание: H T — ожидаемая доля гетерозиготных генотипов как мера общего генного разнообразия во всей популяции; H S — ожидаемая доля гетерозиготных генотипов в отдельной популяции, как мера ее внутрипопуляционного разнообразия или среднее выборочное генное разнообразие по всем локусам; G ST — доля межпопуляционного генетического разнообразия в общем разнообразии или показатель подразделенности популяций; в скобках даны стандартные отклонения.
Установлено, что доля межпопуляционного генетического разнообразия ( G ST ) близка у обоих исследованных видов рода Pinus . При этом значение данного показателя (G ST = 0,320 ) у P. sibirica незначительно выше, чем у P. sylvestris ( G ST = 0,303). В целом, можно сказать, что популяции исследованных видов рода Pinus дифференцированы в средней степени, поскольку на межпопуляционную компоненту у P. sibirica приходится около 32,0% всей генетической изменчивости, а у P. sylvestris – 30,3%.
При подсчете попарных генетических расстояний было установлено, что у P. sibirica на наименьшем генетическом расстоянии (D =0,157) находятся популяции Ps_Kr и Ps_Bs , а на наибольшем ( D =0,213) популяции Ps_Kh и Ps_Bs . У P. sylvestris наиболее генетически удаленными являются популяции Psl_Ks и Psl_Br ( D =0,187), а на наименьшем генетическом расстоянии расположены популяции Psl_Br и Psl_Zc ( D =0,042) .
На основании матриц попарных генетических расстояний ( D ) был проведен кластерный анализ невзвешенным парно-групповым методом (UPGMA) и построены дендрограммы, отражающие степень сходства по ISSR-спектрам исследуемых популяций каждого вида. На дендрограмме популяции P. sibirica Ps_Kr и Ps_Bs сформировали 1 кластер, к ним примыкает популяция Ps_Kh (Рисунок 1А).
Бюллетень науки и практики / Bulletin of Science and Practice Т. 7. №4. 2021
0.4 0.3 0.2 0.1
A-------------------I--------------------!-------------------1--------------------
1001--------------------------------------------------------- Ps_Kr
I--Ps_Bs
_______________________________________________________________ Ps Kb
A
M #5 0.4 U 02 0.1
4-------------------1-------------------1-------------------1-------------------1-------------------1-------------------
100 I Psi Br
---------------1____________________________________________________________________________ Psi Zc _______________________________________________________________________________ Psi Ks
Б
Рисунок 1. UPGMA-дендрограмма генетического сходства исследуемых популяций P. sibirica (А) и популяций P. sylvestris (Б); шкала сверху — генетическое расстояние; на дендрограмме цифрами указаны значения бутстрепа (в %)
На дендрограмме P. sylvestris 1 кластер сформировали выборки Psl_Zc и Psl_Br, а к ним примыкает популяция Psl_Ks (рис. 1Б). О достоверности межкластерных различий можно судить по высокому индексу бутстрепа (>50%) в узлах ветвления.
При выявлении зависимости между генетическими и географическими расстояниями с помощью теста Мантела у P. sibirica был получен высокий коэффициент детерминации (R² = 0,6871), что свидетельствует о высокой корреляции между географическим и генетическим расстояниями у популяций у P. sibirica (Рисунок 2А).
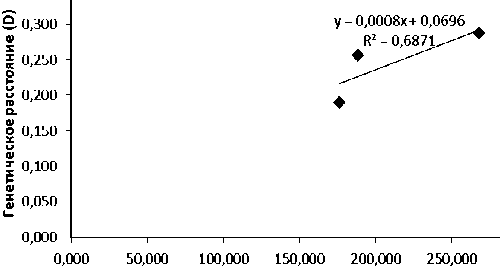
Географическое расстояние (км)
А
Рисунок 2. График зависимости генетических и географических расстояний у изученных популяций: А — P. sibirica ; Б — P. sylvestris
y = 0,0003x + 0,0839

0,037
Географическое расстояние (км)
Б
У P. sylvestris с использованием теста Мантела был выявлен низкий коэффициент детерминации (R 2 = 0,0649), что указывает на низкую степень корреляции между географическим и генетическим расстояниями популяций этого вида (Рисунок 2Б).
Заключение
При молекулярно-генетическом анализе у P. sylvestris выявлено 113 ISSR-PCR маркеров, а у P. sibirica меньше — 102 ISSR-PCR маркера. Доля полиморфных локусов незначительно выше у P. sylvestris (P95=0,885) по сравнению с этим показателем у P. sibirica (P95=0,863). Другие показатели генетического разнообразия незначительно выше у P. sibirica (HE=0,195; na=1,330) в сравнении с P. sylvestris (HE=0,166; na=1,212;). Интересен тот факт, что достоверно отличается у двух изученных видов только один показатель генетического разнообразия, а именно — число эффективных аллелей: ne=1,335 у P. sibirica и ne=1,268 у P. sylvestris. Вместе с тем наибольшее число редких аллелей отмечено у P. sylvestris (R=29). Наибольшим генетическим разнообразием характеризуются популяция Ps_Kh у P. sibirica (HE=0,247; na=1,578; ne=1,427) и популяция Psl_Br у P. sylvestris (HE=0,227; na=1,558; ne=1,375), которые рекомендуются для сохранения генетического разнообразия изученных видов на популяционном уровне. Анализ генетической структуры показал, что коэффициенты генетической подразделенности (GST) у двух изученных видов рода Pinus близки и составляют 0,320 у P. sibirica и 0,303 у P. sylvestris. В связи с этим, популяции сосны сибирской и сосны обыкновенной характеризуются средней степенью генетической дифференциации. На наибольшем генетическом расстоянии находятся популяции Ps_Kh и Ps_Bs у P. sibirica и популяции Psl_Ks и Psl_Br у P. sylvestris, а на наименьшем — популяции Ps_Kr и Ps_Bs P. sibirica и популяции Psl_Br и Psl_ Zc у P. sylvestris. Полученные результаты подтверждаются на UPGMA-дендрограммах генетического сходства. С использованием теста Мантела установлено, что между генетическими и географическими расстояниями у популяций P. sibirica степень корреляции высокая (R2 = 0,6871), а у популяций P. sylvestris — низкая (R2 = 0,0649). Результаты данного исследования важны для разработки рекомендаций по сохранению генофондов двух видов рода Pinus в Пермском крае.
Исследование выполнено при финансовой поддержке Правительства Пермского края в рамках научного проекта №С-26/174.3 от 31.01.2019.
Список литературы Молекулярно-генетический анализ популяций Pinus sylvestris L. и Pinus sibirica du Tour в Пермском крае на основании полиморфизма ISSR-PCR маркеров
- Видякин А. И., Кантор Г. Я. Пространственная организация и факторы формирования групп популяций сосны обыкновенной в Южном Зауралье // Вестник Оренбургского государственного университета. 2013. Т. 159. №10. С. 34-39.
- Yanbaev Y., Sultanova R., Blonskaya L., Bakhtina S., Tagirova A., Tagirov V., Kulagin A. Gene pool of scots pine (Pinus sylvestris L.) Under reforestation in extreme environment // Wood Research. 2020. V. 65. №3. P. 459-470. https://doi.org/10.37763/wr.1336-4561/65.3.459470
- Rogers S. O., Bendich A. J. Extraction of DNA from milligram amounts of fresh, herbarium and mummified plant tissues // Plant molecular biology. 1985. V. 5. №2. P. 69-76. https://doi.org/10.1007/BF00020088
- Нечаева Ю. С., Бельтюкова Н. Н., Пришнивская Я. В., Тайман К. Е. Оптимизация методики выделения ДНК некоторых хвойных видов растений Пермского края // Синтез знаний в естественных науках. Рудник будущего: проекты, технологии, оборудование: Материалы междунар. конф. Пермь, 2011. С. 278-282.
- Zietkiewicz E., Rafalski A., Labuda D. Genome fingerprinting by simple sequence repeat (SSR)-anchored polymerase chain reaction amplification // Genomics. 1994. V. 20. №2. P. 176-183. https://doi.org/10.1006/geno.1994.1151
- Мазунина Ж. И., Нечаева Ю. С. Анализ эффективности ISSR-праймеров для изучения полиморфизма ДНК кедра сибирского Pinus sibirica Du Tour // Научные исследования: теоретико-методологические подходы и практические результаты: материалы Международной научно-практической конференции. Самара, 2017. С. 376-378.
- Бобошина И. В., Нечаева Ю. С., Видякин А. И., Боронникова С. В. Подбор праймеров для проведения ISSR-анализа полиморфизма ДНК Pinus sylvestris L. // Молекулярно-генетические подходы в таксономии и экологии: материалы научной конференции. Ростов-на-Дону, 2013. С. 17-20.
- Yeh F. C., Yang R. C., Mao J., Ye Z., Boyle T. J. POPGENE, the Microsoft Windows-based user-friendly software for population genetic analysis of co-dominant and dominant markers and quantitative traits // Dept. Renewable Resources, University of Alberta, Edmonton, Canada. 1996. V. 238.
- Peakall R. O. D., Smouse P. E. GenALEX 6: genetic analysis in Excel. Population genetic software for teaching and research // Molecular ecology notes. 2006. V. 6. №1. P. 288-295. https://doi.org/10.1111/j.1471-8286.2005.01155.x
- Williams J. G., Kubelik A. R., Livak K. J., Rafalski J. A., Tingey S. V. DNA polymorphisms amplified by arbitrary primers are useful as genetic markers // Nucleic acids research. 1990. V. 18. №22. P. 6531-6535. https://doi.org/10.1093/nar/18.22.6531
- Kimura M., Crow J. F. The number of alleles that can be maintained in a finite population // Genetics. 1964. V. 49. №4. P. 725. https://doi.org/10.1093/genetics/49A725
- Nei M. Molecular evolutionary genetics. Columbia university press, 1987.
- Nei M. Molecular population genetics and evolution. North-Holland Publishing Company, 1975.
- Nei M., Li W. H. Mathematical model for studying genetic variation in terms of restriction endonucleases // Proceedings of the National Academy of Sciences. 1979. V. 76. №10. P. 52695273. https://doi.org/10.1073/pnas.76.10.5269
- Mantel N. The detection of disease clustering and a generalized regression approach // Cancer research. 1967. V. 27. №2. Part 1. P. 209-220.


