Оценка генетического полиморфизма сортов, линий и мутантов гороха посевного (Pisum sativum L.) с помощью ДНК-маркеров на основе ретротранспозонов
Автор: Кокаева З.Г., Гостимский С.А.
Журнал: Сельскохозяйственная биология @agrobiology
Рубрика: ДНК-технологии в селекции растений
Статья в выпуске: 3 т.42, 2007 года.
Бесплатный доступ
Резюме. IRAP-метод использовали для изучения инсерционного полиморфизма ретротранспозо-нов группы Ту1-copia у 16 сортов, линий и мутантов гороха посевного (Pisum sativum L.). Оценили эффективность десяти комбинаций праймеров при выявлении полиморфизма у анализируемых образцов, получили полиморфные маркеры генома гороха. На основании IRAP-анализа определили генетические дистанции и построили дендрограмму, отражающую генетическое сходство и родство между исследованными образцами. Для каждого из анализируемых сортов и линий получили индивидуальный IRAP-спектр с целью выявления сортоспецифичных фрагментов, пригодных для паспортизации и маркирования изученных форм.
Горох, филогенетический анализ, ретротранспозон
Короткий адрес: https://sciup.org/142134816
IDR: 142134816 | УДК: 635.656:575.17.015.3:577.088
Текст краткого сообщения Оценка генетического полиморфизма сортов, линий и мутантов гороха посевного (Pisum sativum L.) с помощью ДНК-маркеров на основе ретротранспозонов
Ранее для паспортизации и идентификации сортов и линий гороха посевного мы применили RAPD-метод (9).
Целью настоящей работы было изучение генетического полиморфизма сортов, линий и мутантов гороха Pisum sativim L. с использованием ДНК-маркеров на основе ретротранспозонов группы Ty1- copia .
Методика . Анализировали растения 16 образцов гороха посевного: сорта Демон, Филби, Флагман, Виола, Немчиновский, Ранний Зеленый, Торсдаг, Finale, Rondo из коллекции кафедры генетики биологического факультета МГУ им. М.В. Ломоносова, линии L-131, L-851, L-1132, L-1238, L-102 из Института генетики сельскохозяйственных растений (Швеция) и хлорофильные мутанты Хл 42 и Хл 15, полученные обработкой семян соответственно сортов Немчиновский и Торсдаг этилметансульфона-том. Тотальную ДНК выделяли из молодых листьев методом CTAB с изменениями (10).
В работе использовали следующие праймеры (11):
Tps 3 5'-CCTTTGGGATATTAACCACAC-3'
Tps 6 5'-GTGAGATAGTTATATGTC-3'
Tps 7 5'-CTATAATACATAACAAGC-3'
Tps 10 5'-GGAATGATAGGCCTTGCC-3'
*RNaseH 1 5'-MGNACNAARCAYATHGA-3'
*RNaseH 2 5'-GCNGAYATNYTNACNAA-3'
* Последовательности вырожденных праймеров.
Амплификацию проводили в термоциклере «Терцик» («ДНК-технология», Россия) по следующей программе: предварительная денатурация при 95 оС в течение 2 мин; 30 циклов при 95 оС в течение 60 с; при температуре отжига в течение 60 с; при 72 оС в течение 2 мин; конечная элонгация при 72 оС в течение 10 мин. Реакционная смесь объемом 25 мкл содержала 50 нг ДНК, 2 ед. Taq-полимеразы («Силекс М», Россия), 2,5 мкл стандартного 10-кратного буфера для ПЦР («Силекс М», Россия), 25 пМ каждого праймера, 2,5 мМ Mg2+ и 0,25 мМ dNTP; на смесь наслаивали две капли минерального масла.
Работа выполнена при финансовой поддержке РФФИ (грант № 07-04-00652) и программы президиума РАН «Динамика генофондов растений, животных и человека».
Продукты амплификации разделяли электрофорезом в 2 % агарозном геле с однократным ТВЕ-буфером и окрашивали бромистым этидием. Длину фрагментов определяли по маркерам молекулярной массы 100 bp + 1,5+3 Kb DNA Ladder («Сибэнзим», Россия). Гели просматривали в УФ-свете ( λ = 312 нм); результаты документировали с использованием цифровой фотокамеры. Э ксперименты проводили в нескольких повторностях.
Для количественной оценки полиморфизма и определения генетического расстояния между исследованными формами данные IRAP-анализа были представлены в виде матрицы состояний, в которой наличие или отсутствие в IRAP-спектрах фрагментов одинаковой длины обозначили соответственно 1 или 0. По матрице состояний была составлена матрица различий, генетические расстояния в которой рассчитывались по формуле Nei и Li (12) с помощью компьютерной программы Treecon (13).
На основании полученной матрицы невзвешенным парно-групповым методом (UPGMA — Unweighted Pair-Group Method) (13) была построена генеалогическая дендрограмма, отражающая степень различий между IRAP-спектрами исследуемых форм. Для построения дендрограммы использовали компьютерную программу Treecon (13). Оценку значимости генеалогических реконструкций проводили методом бутстрепа (14). Топология дерева в участках со значениями больше 50 % может считаться надежно установленной.
Результаты . Из 21 протестированной комбинации праймеров десять эффективно выявляли полиморфизм, а три могли быть использованы при паспортизации сортов и линий гороха (табл. 1). Для оценки полиморфизма генома в каждой комбинации праймеров определяли отношение числа полиморфных фрагментов к общему числу амплифицированных. Было показано, что у проанализированных сортов, линий и мутантов гороха отдельные праймеры выявляли от 7 до 25 фрагментов размером 1501500 п.н., из которых от 4 до 13 были полиморфными (рис. 1). Всего получили 130 ДНК-фрагментов, из них 72 были полиморфными. Общий уровень полиморфизма с десятью комбинациями IRAP-праймеров составил 55,38 %. При оценке внутрилинейной изменчивости у линии L-131 показали, что IRAP-спектры растений гороха, полученные с парой праймеров (Tps 7 - Tps 10), идентичны (рис. 2).
1. Эффективность комбинаций праймеров при выявлении полиморфизма IRAP-фрагментов ДНК
|
Праймер Праймер |
Tps 3 |
Tps 6 |
Tps 7 |
Tps 10 |
RNaseH 1 |
RNaseH 2 |
Для количественной оценки полиморфизма и определения степени дивергенции между изученными формами гороха данные, полученные при анализе IRAP-полиморфизма, представили в виде матрицы состояний 130 бинарных признаков, которую использовали для расчета генетических расстояний и построения дендрограммы.
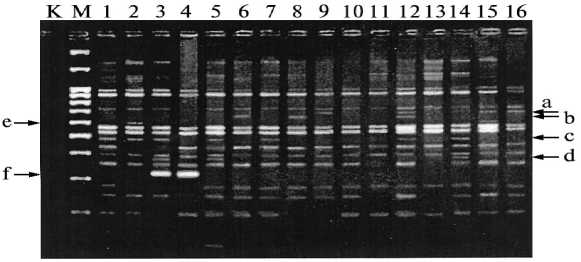
Рис. 1 . Продукты IRAP-амплификации ДНК сортов (1-7, 10, 11), линий (8, 9, 12, 15, 16) и мутантов гороха (13, 14) с парой праймеров (Tps 7 - Tps 10): 1 — Демон, 2 — Филби, 3 — Флагман, 4 — Виола, 5 — Немчиновский, 6 — Rondo, 7 — Finale, 8 — L-1238, 9 — L-102, 10 — Ранний Зеленый, 11 — Торсдаг, 12 — L-851, 13 — Хл 15, 14 — Хл 42, 15 — L-131, 16 — L-1132. К — отрицательный контроль; М — маркер молекулярной массы 100 bp + 1,5 + 3 Kb DNA Ladder. Стрелками отмечены полиморфные фрагменты: a — 690 п.н., b — 680 п.н., с — 490 п.н., d — 380 п.н., e — 600 п.н., f — 320 п.н.
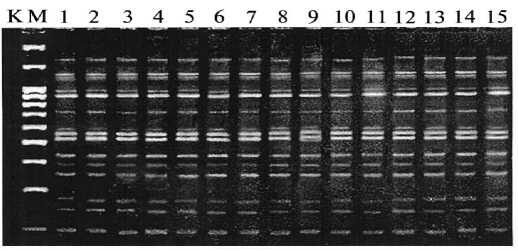
Рис. 2. IRAP-спектры ДНК-фрагментов у растений гороха линии L-131, полученные с парой праймеров ( Tps 7 - Tps 10): 1-15 — индивидуальные растения гороха линии L-131. К — отрицательный контроль; М — маркер молекулярной массы 100 bp + 1,5 + 3 Kb DNA Ladder.
Уровень различий между мутантами Хл 42, Хл 15 и исходными сортами составил 0 %, средний уровень полиморфизма между формами — 23,74 %, что согласуется с ранее полученными данными (9).
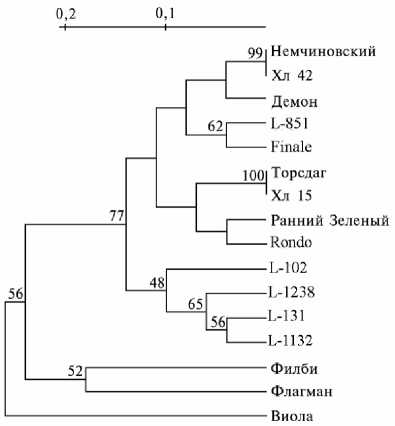
Рис. 3. Дендрограмма исследованных линий, сортов и мутантов гороха (по методу UPGMA). Верхняя шкала — генетическое расстояние по Nei и Li (12). Цифрами указаны значения бут-стрепа, %.
Дендрограмма, отражающая степень различий между IRAP-спектрами образцов гороха, представлена на рисунке 3. Исходя из матрицы различий, наибольшие генетические расстояния были установлены между сортом Флагман и маркерными линиями (37,77 %), наименьшие — между мутантами и исходными сортами (0 %). Дендрограмма показывает, что сорт Немчиновский и полученный из него мутант Хл 42, а также сорт Демон кластеризуются в одну группу; сорт Торсдаг и полученный из него мутант Хл 15, сорта Ранний Зеленый и Rondo — в другую (уровень различий между кластерами составил 0,03). Вместе кластеризуются маркерные линии шведской селекции L-1132, L-131, L-1238 и линия L-102 (уровень различий между маркерными линиями и сортами — 0,02-0,1). Сорт Виола оказался обособленным от остальных, что, возможно, объясняется их разным происхождением. Эти результаты также согласуются с полученными нами ранее (9).
-
2. Матрица ДНК-фрагментов для паспортизации сортов, линий и мутантов гороха посевного ( Pisum sativum L.) с помощью IRAP- маркеров
Образец
Длина ДНК-фрагментов, п.н.
710
1 690
680
600 490
440
380
320
280
250
200
Демон
+
–
–
Сорта – +
+
–
–
+
+
+
Филби
+
–
–
+ +
–
–
–
–
+
+
Флагман
+
–
–
– +
–
+
+
–
+
-
Виола
–
–
–
– –
+
–
+
–
–
+
Немчиновский
+
–
–
– +
+
+
–
+
+
+
Rondo
+
–
+
– –
+
–
–
+
+
+
Finale
+
+
–
– +
+
–
–
+
+
+
Ранний Зеленый
+
–
–
–
+
–
–
+
+
+
Торсдаг
+
–
–
– –
+
–
–
+
–
+
L-851
+
+
+
Линии – +
+
–
–
+
+
+
L-131
+
+
–
+ –
+
–
–
+
+
+
L-1132
+
+
+
+ –
+
–
–
+
+
+
L-1238
+
+
+
+ –
+
–
–
+
+
–
L-102
+
+
–
– –
+
–
–
+
+
–
Хл 15
+
–
Му –
тантные формы – – +
–
–
+
–
+
Хл 42
+
–
–
– +
+
+
–
+
+
+
П р и м е ч а н и
е. «+»
и «–»
означает
наличие и отсутствие
маркера.
ДНК-фрагменты
получены с
комбинацией праймеров (Tps 7 - Tps 10).
Для однозначной идентификации изученных форм гороха посевного методом IRAP-PCR оказалось достаточно использовать несколько пар праймеров: (Tps 7 - Tps 10), (Tps 3 - Tps 6), (Tps 3 - Tps 7). IRAP-маркирование с этими праймерами позволяло амплифицировать уникальные для каждого образца IRAP-спектры у всех исследованных сортов и линий. Так, в спектре, полученном с комбинацией праймеров (Tps7 - Tps 10), фрагмент размером 680 п.н. присутствовал только у сорта Rondo и трех маркерных линий — L-1238, L-102, L-1132; фрагмент 690 п.н. — только у маркерных линий (кроме L-1238); фрагмент 600 п.н. обнаружили у сорта Филби и у двух маркерных линий — L-1238, L-1132; фрагмент 320 п.н. — только у сортов Флагман и Виола, а фрагмент 380 п.н. — у сортов Флагман и Немчиновский, а также у мутанта сорта Немчиновский Хл 42 (см. рис. 1). Фрагмент 280 п.н. отсутствовал только у трех сортов — Филби, Флагман и Виола.
Выявленная специфичность спектров позволяет проводить быструю идентификацию образцов. Набор ДНК-фрагментов, полученный в результате амплификации, может служить молекулярногенетическим паспортом сорта или линии гороха (табл. 2).
Таким образом, использование IRAP-метода позволяет достаточно эффективно дифференцировать разные генетические формы гороха. Полученные IRAP- маркеры можно использовать не только для идентификации и паспортизации сортов и линий гороха, но также для филогенетических исследований и в работе по картированию хромосом. По сравнению с другими маркерными системами выявления генетического полиморфизма IRAP-метод чувствительнее и специфичнее, при этом он прост в исполнении, не требует использования радиоактивных изотопов и эндонуклеаз рестрикции, полученные результаты хорошо воспроизводимы. Единственное ограничение применения метода состоит в том, что необходимо знать нуклеотидную последовательность терминальных участков мобильных элементов.
Л И Т Е Р А Т У Р А
-
1. Г о с т и м с к и й С.А., К о к а е в а З.Г., К о н о в а л о в Ф.А. Изучение организации и изменчивости генома растений с помощью молекулярных маркеров. Генетика, 2005, 41, 4: 1-15.
-
2. P e a r c e S.R., K n o x M., E l l i s T.H.N. e.a. Pea Ty1- copia group retrotransposons: transpositional activity and use as markers to study genetic diversity in Pisum . Mol. Gen. Genet., 2000, 263: 898-907.
-
3. K u m a r A., B e n n e t z e n J. Plant retrotransposons. Annu. Rev. Genet., 1999, 33: 479-532.
-
4. T a t o u t C., W a r w i c k S., L e n o i r A. e.a. Sine insertions as clad markers for wild Crucifer species. Mol. Biol. Evol., 1999,
16: 1614-1621.
-
5. W a u g h R., M c L e a n K., F l a v e l l A.J. e.a. Genetic distribution of BARE-1-like retrotransposable elements in the barley genome revealed by sequence-specific amplification polymorphisms (S-SAP). Mol. Gen. Genet., 1997, 253: 687-694.
-
6. I w a m o t o M., N a g a s h i m a H., N a g a m i n e T. e.a. p-SINE-like intron of the CatA catalase homologs and phylogenetic relationships among AA-genome Oryza and related species. Theor. Appl. Genet., 1999, 98: 853-861.
-
7. T e o C.H., T a n S.H., H o C.L. e.a. Genome constitution and classification using retrotransposon-based markers in the Orphan Crop Banana. J. of Plant Biology, 2005, 48: 96-105.
-
8. K a l e n d a r R., G r o b T., R e g i n a M. e.a. IRAP and REMAP: two new retrotransposon-based DNA fingerprinting techniques. Theor. Appl. Genet., 1999, 98: 704-711.
-
9. Д р и б н о х о д о в а О.П., К о к а е в а З.Г., Г о с т и м с к и й С.А. Идентификация сортов, линий и мутантов гороха посевного с помощью RAPD-маркеров. С.-х. биол., 2005, 5: 61-66.
-
10. T o r r e s A.M., W e e d e n N.F., M a r t i n A. Linkage among sozyme, RFLP and RAPD markers in Vicia faba. Theor. Appl. Genet., 1993, 5: P.937-945.
-
11. P e a r c e S.R., S t u a r t - R o g e r s C., K n o x M.R. e.a. Rapid isolation of plant Ty1-copia group retrotransposon LTR sequences for molecular marker studies. The Plant Journal, 1999, 19: 711-717.
-
12. N e i M., L i W.-H. Mathematical model for studying genetic variation in terms of restriction endonucleases. Proc. Nat. Acad. Sci. USA, 1979, 76: 5269-5273.
-
13. V a n d e P e e r Y. TREECON for Windows: a software package for the construction and drawing of evolutionary trees for the Mi-
crosoft Windows environment. Computer Application in the Biosciences, 1994, 10, 5: 569-570.
-
14. F e l s e n s t e i n J. Confidence limits in phylogenies: an approach using bootstrap. Evolution, 1985, 39, 4: 783-791.
Московский государственный университет им. М.В. Ломоносова ,


