Определение консенсусных генотипов по микросателлитам у музейных краниологических образцов крупного рогатого скота (Вos taurus)
Автор: Абдельманова А.С., Волкова В.В., Харзинова В.Р., Форнара М.С., Чинаров Р.Ю., Боронецкая О.И., Трухачев В.И., Brem G., Зиновьева Н.А.
Журнал: Сельскохозяйственная биология @agrobiology
Рубрика: Генетика и геномика
Статья в выпуске: 6 т.58, 2023 года.
Бесплатный доступ
Анализ ДНК древних и исторических образцов, в том числе экземпляров из музейных и краниологических коллекций, неоценим как способ получения генетической информации для реконструкции происхождения локальных пород животных. Принимая во внимание высокую степень деградации ДНК в большинстве таких образцов, исследования проводят, как правило, на митохондриальном геноме, поскольку он присутствует в сотнях или даже тысячах копий в одной клетке. Однако в некоторых случаях исследование митохондриальной ДНК (мтДНК) не позволяет в полной мере проследить демографическую историю видов и пород, особенно при вовлечении в скрещивания производителей, представляющих улучшающие породы. Информативным инструментом для анализа такого рода демографических событий служат микросателлиты, или короткие тандемные повторы (STR). Однако генотипирование микросателлитов при использовании ДНК, выделенной из музейных образцов, сопряжено с повышенным риском ошибок амплификации. Цель нашей работы - усовершенствование алгоритма определения консенсусных генотипов STR-маркеров для проб, содержащих сильно деградированную ДНК, и оценка его эффективности на музейных образцах костной ткани крупного рогатого скота. Материалом для исследования служили музейные экспонаты черепов крупного рогатого скота, датированные концом XIX-первой половиной XX века, хранящиеся в краниологической коллекции Музея животноводства им. Е.Ф. Лискуна (РГАУ - МСХА им. К.А. Тимирязева, г. Москва). Образцы генотипировали с помощью мультиплексной панели, включавшей 11 микросателлитных локусов, рекомендованных Международным обществом генетики животных (ISAG), по протоколам, принятым в ФГБНУ ФИЦ ВИЖ им. академика Л.К. Эрнста. Успешность амплификации для каждого локуса в образце оценивали на основании расчета индексов качества генотипирования (quality indices, QI). Наиболее часто встречающийся генотип кодировали как 1, а отличающиеся от него за счет выпадения аллелей (allelic drop-out, ADO) или ложных аллелей (false alleles, FA) генотипы - как 0. Далее рассчитывали долю генотипов со значением 1 по отношению к общему числу повторов. Пороговое значение для QI было установлено на уровне 0,75. В консенсусный генотип включали генотипы, показавшие частоту встречаемости выше порогового значения для каждого локуса. Тестирование алгоритма выполнено на 144 музейных образцах черно-пестрого, турано-монгольского, палево-пестрого и бурого крупного рогатого скота. Полный профиль (11 микросателлитных локусов) удалось получить для 60,42 % случаев. Качество генотипирования в большинстве локусов (9 из 11 исследованных) было выше 0,950, варьируя от 0,951±0,011 в локусе TGLA122 до 0,995±0,003 в локусе BM2113. По результативности наименее успешным оказалось генотипирование локусов TGLA53 и BM1818 (соответственно 74,86 % и 61,45 %). Выявлена положительная корреляция на уровне тенденции ( r 2 = 0,53; p = 0.09) между размером аллелей в локусе и долей ошибок генотипирования. Поскольку изучение аллелофонда популяций невозможно без получения корректных генотипов, предложенный нами алгоритм, обеспечивающий вероятность корректного генотипирования p function show_abstract() { $('#abstract1').hide(); $('#abstract2').show(); $('#abstract_expand').hide(); }
Микросателлиты, ошибки генотипирования, консенсусный генотип, крупный рогатый скот, музейные образцы
Короткий адрес: https://sciup.org/142240675
IDR: 142240675 | УДК: 636.2:57.082.133:577.2 | DOI: 10.15389/agrobiology.2023.6.1035rus
Текст научной статьи Определение консенсусных генотипов по микросателлитам у музейных краниологических образцов крупного рогатого скота (Вos taurus)
Анализ ДНК древних и исторических образцов — неоценимый источник генетической информации для реконструкции происхождения локальных пород сельскохозяйственных животных (1-3). Особую ценность для таких исследований представляют экземпляры музейных (4, 5) и краниологических (6) коллекций. Принимая во внимание высокую степень деградации ДНК в большинстве таких образцов, исследования, как правило, проводят на митохондриальном геноме (7-9), поскольку он присутствует в сотнях
∗ При выполнении исследований использовали оборудование ЦКП «Биоресурсы и биоинженерия сельскохозяйственных животных» ФГБНУ ФИЦ ВИЖ им. академика Л.К. Эрнста. Работа выполнена при поддержке Российского научного фонда, проект ¹ 19-76-20012.
или даже тысячах копий в одной клетке. Однако в некоторых случаях анализ митохондриальной ДНК (мтДНК) не позволяет в полной мере проследить демографическую историю видов и пород животных (10, 11). Так, при создании некоторых отечественных пород крупного рогатого скота практиковалось вводное скрещивание с использованием быков-производителей зарубежных пород (6). Анализ митохондриального генома не позволит оценить такие события в демографической истории пород и проследить участие зарубежных пород в формировании аллелофонда современных популяций, поскольку у мтДНК материнский тип наследования. Информативным инструментом для анализа такого рода демографических событий служит исследование микросателлитов, или коротких тандемных повторов (short tandem repeats, STR) (12). Микросателлитный анализ признается золотым стандартом контроля происхождения в программах разведения крупного рогатого скота (13), что позволило получить большой объем информации о генотипах современных представителей локальных и трансграничных пород. Наличие больших наборов генетических данных, полученных на современных популяциях, имеет важное значение для изучения исторических образцов, поскольку может помочь проследить сохранение исторических аллелей в современных популяциях.
Основная проблема, связанная с генотипированием микросателлитов, в случае ДНК, выделенной из археологических и музейных образцов, заключается в ошибках амплификации. Это, в частности, ложные гомозиготы, или выпадение аллелей (allelic drop-out, ADO), и ложные аллели (false alleles, FA) (14, 15), которые представляют собой артефакты, полученные при проведении полимеразной цепной реакции (ПЦР) (16). Для повышения достоверности результатов генотипирования сильно деградированной ДНК или ДНК с низкой концентрацией был предложен мультиплексный подход, основанный на повторных независимых амплификациях каждого образца ДНК (15, 17). Такой подход по сравнению со стандартной процедурой позволяет количественно оценить степень ошибок для каждого возможного генотипа (17).
Несмотря на многочисленные работы, посвященные изучению ал-лелофонда крупного рогатого скота в нашей стране, до недавнего времени отсутствовали протоколы получения консенсусных генотипов с помощью микросателлитных маркеров для образцов, сохраняемых в краниологических коллекциях, поэтому ретроспективное изучение генетического пула предковых популяций локальных пород не представлялось возможным. Отметим, что в мире уже проводились исследования филогенетических связей между образцами, обнаруженными в ходе археологических изысканий, и современными породами с помощью STR-маркеров. Однако при этом использовались очень трудоемкие и дорогостоящие методы с большим числом повторов экстракции ДНК и амплификации одиночных локусов для получения корректных генотипов (2).
В настоящем сообщении впервые приведено описание и результаты оценки разработанного нами подхода к анализу музейных образцов, позволяющего наиболее эффективно получать корректные генетические данные по микросателлитам.
Нашей целью было усовершенствование алгоритма определения консенсусных генотипов STR-маркеров для образцов, содержащих сильно деградированную ДНК, и оценка его эффективности на музейных образцах крупного рогатого скота.
Методика. Источником ДНК служили музейные экспонаты черепов крупного рогатого скота, датированные концом XIX—первой половиной XX 1036
века, из краниологической коллекции Музея животноводства им. Е.Ф. Лискуна РГАУ — МСХА им. К.А. Тимирязева (г. Москва, 1950 год). Исследуемая выборка включала 144 музейных образца биоматериала от черно-пестрого, ту-рано-монгольского, палево-пестрого и бурого крупного рогатого скота, ге-нотипированных в период с 2019 по 2021 год.
Подготовку образцов и выделение ДНК проводили, как описано нами ранее (18). Извлеченные из черепов зубы промывали детергентом и дистиллированной водой для удаления возможного загрязнения современной ДНК. Зуб продольно распиливали и с помощью мини-дрели Dremel 3000-15 («Dremel», США) с алмазным бором на самых малых оборотах из внутренней части высверливали мелкодисперсный порошок. Далее следовали этапы растворения костного порошка в лизирующем растворе, отмывания от примесей, ингибирующих ПЦР, и получения очищенного экстракта ДНК в соответствии с протоколами, рекомендованными изготовителями коммерческих наборов Prep Filer™ BTA Forensic DNA Extraction Kit («Thermo Fisher Scientific, Inc.», США), COrDIS Экстракт декальцин (ООО «ГОРДИЗ», Россия), М-сорб-кость (ООО «Синтол», Россия), QIAamp DNA Investigator Kit («Qiagen», США). Качество полученных препаратов ДНК оценивали, измеряя концентрации двухцепочечной ДНК (дцДНК) (флуориметр Qubit™, «Invitrogen, Life Technologies», США) и определяя соотношение OD 260/280 (спектрофотометр NanoDrop™ 8000 («Thermo Fisher Scientific, Inc.», США). Учитывая, что при увеличении количества ДНК хорошего качества снижается риск ошибочного генотипирования (19), при микросателлитном анализе в качестве пороговых значений были выбраны концентрация дцДНК не менее 1 нг/мкл и соотношение OD 260/280 в пределах 1,6-2,0. Препараты ДНК, не отвечающие таким требованиям, из исследований исключали.
Образцы генотипировали с помощью мультиплексной панели с 11 микросателлитными локусами (TGLA227, BM2113, TGLA53, ETH10, SPS115, TGLA122, INRA23, TGLA126, BM1818, ETH225, BM1824; рекомендованы Международным обществом генетики животных ISAG) (20) по протоколам, принятым в ФГБНУ ФИЦ ВИЖ им. академика Л.К. Эрнста (21).
Мультиплексные ПЦР проводили в конечном объеме 10 мкл в ПЦР-буфере с 200 мМ dNTP, 1,0 мМ MgCl2, 0,5 мМ смеси праймеров (последовательности приведены на , 1 ед. Taq-полимеразы (ООО «Диалат», Россия) и 1 мкл геномной ДНК (концентрации выше 1 нг/мкл). ПЦР выполняли в следующем температурновременном режиме: начальная денатурация при 95 °С, 4 мин; 95 °С, 20 с; 63 °С, 30 с (35 циклов); 72 °С, 1 мин. Завершающую элонгацию проводили при 72 °С в течение 10 мин. Определяли размер полученных фрагментов (генетический анализатор ABI3130xl со стандартом длин фрагментов GeneScan™ 350 ET ROX™, программное обеспечение GeneMapper™ v. 4; «Applied Biosystems», США). Размеры аллелей стандартизировали в соответствии со сравнительным тестированием STR-типирования ISAG для вида Bos taurus 2018-2019 годов.
В качестве прототипа для определения консенсусных генотипов использовали модифицированный мультиплексный подход, предложенный ранее (22, 23). Расчет индексов качества генотипирования (quality indices, QI) для каждого образца/локуса проводили согласно С. Miquel с соавт. (24).
Наиболее часто встречающемуся генотипу в каждом локусе присваивали код 1. Генотипы, отличающиеся от наиболее часто встречающегося генотипа за счет выпадения аллелей (ADO) или ложных аллелей (FA), считали ошибками генотипирования и обозначали как 0. Далее рассчитывали долю генотипов со значением 1 по отношению к общему числу повторов. Пороговое значение для QI было установлено на уровне 0,75.
Результативность амплификации (долю +ПЦР) рассчитывали как число успешных ПЦР (+ПЦР), деленное на число повторов ПЦР для каждого локуса, выраженное в процентах. Для расчета доли неудачных амплификаций из 100 % вычитали долю +ПЦР в процентах. Для расчета доли утерянных аллелей (ADO) и доли ложных аллелей (FA) использовали протокол, предложенный T. Broquet и E. Petit (25). Долю ADO для каждого локуса рассчитывали для гетерозиготных генотипов (согласно соответствующим консенсусным генотипам) как число повторов, в которых был утерян один аллель, деленное на общее число +ПЦР. Показатель FA рассчитывали для гомозиготных или гетерозиготных генотипов как число ПЦР с ложными аллелями, деленное на общее число +ПЦР.
Анализируя данные генотипирования на наличие ложных результатов, влияющих на оценку параметров популяции (26, 27), рассчитывали долю ошибок (error rates, ER) как число генотипов, отличающихся от консенсусного, поделенное на общее число +ПЦР. Вероятность корректного генотипирования (p) для каждого локуса вычисляли в соответствии с описанием G. He с соавт. (28). Кроме того, рассчитывали средние значения и стандартные ошибки средних значений индекса качества генотипирования QI ( M ±SEM) для каждого локуса. По результатам оценки качества генотипирования вычисляли коэффициенты корреляции Пирсона ( r2 ), иллюстрирующие связь между длиной аллелей и долей ошибок генотипирования в локусах.
Результаты. Данные об исследуемых микросателлитных локусах обобщены в таблице 1.
1. Характеристика микросателлитных локусов, использованных при мультиплексном генотипировании краниологических музейных образцов крупного рогатого скота (Музей животноводства им. Е.Ф. Лискуна РГАУ — МСХА им. К.А. Тимирязева, г. Москва, 1950 год)
|
Локус |
BTA |
Число повторов |
Длина аллелей, bp |
||
|
min |
max |
min |
max |
||
|
BM1818 |
23 |
13 |
25 |
256 |
280 |
|
BM1824 |
1 |
10 |
18 |
174 |
190 |
|
BM2113 |
2 |
12 |
23 |
121 |
143 |
|
ETH10 |
5 |
14 |
22 |
209 |
225 |
|
ETH225 |
9 |
19 |
29 |
140 |
160 |
|
INRA023 |
3 |
10 |
25 |
192 |
222 |
|
SPS115 |
15 |
17 |
28 |
240 |
262 |
|
TGLA122 |
21 |
14 |
38 |
137 |
185 |
|
TGLA126 |
20 |
12 |
22 |
105 |
125 |
|
TGLA227 |
18 |
9 |
25 |
71 |
103 |
|
TGLA53 |
16 |
17 |
40 |
150 |
196 |
Пр и м еч ани е. BTA — аутосома крупного рогатого скота.
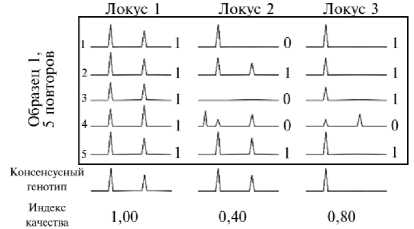
Рис. 1. Схема определения индекса качества генотипирования (QI) при мультиплексном генотипировании краниологических образцов крупного рогатого скота (Музей животноводства им. Е.Ф. Лискуна РГАУ — МСХА им. К.А. Тимирязева, г. Москва, 1950 год) .
Схема определения индексов качества для локусов приведена на рисунке 1. В локусе 1 профили всех повторов четко показывают наличие двух аллелей
(с разной интенсивностью). В локусе 2 наиболее часто встречающийся генотип визуализируется в повторах 2 и 5 (оцениваются как 1). В повторе 1 для этого локуса наблюдается выпадение аллеля (ADO), в повторе 4 — появление ложного аллеля (FA), повтор 3 показывает отсутствие амплификации, поэтому все эти повторы оцениваются как 0. В локусе 3 в повторе 4 наблюдается ложный аллель (оценка повтора 0), во всех остальных повторах виден четкий гомозиготный профиль (оценка 1). Таким образом, значения QI для трех вышеназванных локусов составили соответственно 1,00; 0,40 и 0,80.
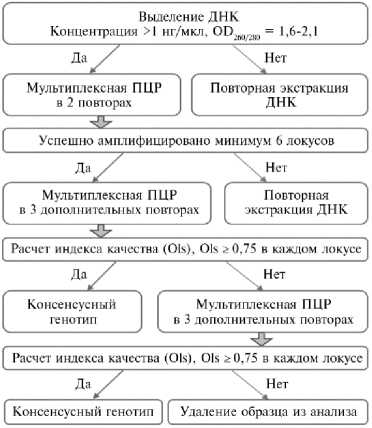
Рис. 2. Схема определения консенсусных генотипов по микросателлитам при мультиплексном генотипировании краниологических образцов крупного рогатого скота (Музей животноводства им. Е.Ф. Лискуна РГАУ — МСХА им. К.А. Тимирязева, г. Москва, 1950 год).
Нами предложен модифицированный алгоритм определения консенсусных генотипов при работе со сложными образцами на примере музейных образцов черепов крупного рогатого скота (рис. 2). После первоначальной мультиплексной амплификации микросателлитных локусов в двух повторах для дальнейшего анализа отбирали только те образцы, в которых успешно амплифицирова-ли не менее шести локусов (+ПЦР). Для таких образцов выполняли три дополнительных независимых повтора ПЦР с использованием тех же препаратов ДНК. Таким образом, каждый образец ДНК анализировали не менее чем в пяти повторах. Для образцов, в которых было амплифицировано менее шести локусов или получено менее четырех положительных ПЦР в каждом из локусов, проводили повторные выделение ДНК и постановку ПЦР, как описано выше.
Как представлено на рисунке 2, образцы, имеющие QI = 0,75 и выше в каждом из локусов, считались корректно генотипированными и отбирались для дальнейшего анализа. Для образцов со значением QI меньше установленного порога (QI < 0,75) в любом из локусов были проведены три дополнительных мультиплексных ПЦР с использованием тех же препаратов ДНК, после чего повторно рассчитывали индексы качества. Если вновь получали низкие значения индексов качества (QI < 0,75), для таких образцов повторно выделяли ДНК и проводили ПЦР с использованием описанного выше мультиплексного подхода (при чрезвычайной ценности конкретного образца, например в случае, когда исследуемая порода представлена всего 1-2 особями) или исключали из дальнейшего анализа.
2. Результативность генотипирования краниологических образцов крупного рогатого скота по микросателлитам ( n = 144, Музей животноводства им. Е.Ф. Лискуна РГАУ — МСХА им. К.А. Тимирязева, г. Москва, 1950 год)
|
Число успешно генотипированных локусов |
Число образцов, n |
Доля образцов, % |
|
11 |
87 |
60,42 |
|
10 |
21 |
14,58 |
|
9 |
9 |
6,25 |
|
8 |
3 |
2,08 |
|
7 |
3 |
2,08 |
|
6 |
3 |
2,08 |
|
< 6 |
27 |
18,75 |
Концентрация двухцепочечной ДНК в исследуемой выборке варьировала от 1,01 до 63,40 нг/мкл, величина OD 260/280 — от 1,64 до 2,00. Как показано в таблице 2, для 60,42 % из 144 генотипированных образцов удалось получить полный профиль (11 локусов микросателлитов), 14,58 и 6,25 % образцов были успешно генотипированы соответственно по 10 и 9 локусам микросателлитов. Для 18,75 % образцов успешно генотипировали 5 и менее локусов, вследствие чего эти образцы исключили из дальнейшего анализа.
-
3. Оценка качества генотипирования краниологических образцов крупного рогатого скота по локусам микросателлитов на основании индекса QI ( n = 144, Музей животноводства им. Е.Ф. Лискуна РГАУ — МСХА им. К.А. Тимирязева, г. Москва, 1950 год)
-
4. Результативность и распределение ошибок генотипирования краниологических образцов крупного рогатого скота по локусам микросателлитов ( n = 144, Музей животноводства им. Е.Ф. Лискуна РГАУ — МСХА им. К.А. Тимирязева, г. Москва, 1950 год)
|
Локус |
Среднее значение QI ( M ±SEM) |
QI = 1 |
0,75 ≤ QI < 1,00 |
QI < 0,75 |
р |
|||
|
1 |
2 |
1 |
2 |
1 |
2 |
|||
|
TGLA227 |
0,966±0,009 |
110 |
87,30 |
11 |
8,73 |
5 |
3,97 |
p < 0,001 |
|
BM2113 |
0,995±0,003 |
122 |
96,83 |
3 |
2,38 |
0 |
0,00 |
p < 0,001 |
|
TGLA53 |
0,759±0,030 |
68 |
53,97 |
20 |
15,87 |
38 |
30,16 |
p < 0,001 |
|
ETH10 |
0,972±0,008 |
112 |
88,89 |
10 |
7,94 |
4 |
3,17 |
p < 0,001 |
|
SPS115 |
0,953±0,011 |
104 |
82,54 |
18 |
14,29 |
4 |
3,17 |
p < 0,001 |
|
TGLA122 |
0,951±0,011 |
107 |
84,92 |
10 |
7,94 |
9 |
7,14 |
p < 0,001 |
|
INRA23 |
0,952±0,013 |
107 |
84,92 |
14 |
11,11 |
5 |
3,97 |
p < 0,001 |
|
TGLA126 |
0,960±0,009 |
106 |
84,13 |
15 |
11,90 |
5 |
3,97 |
p < 0,001 |
|
BM1818 |
0,700±0,040 |
82 |
65,08 |
5 |
3,97 |
39 |
30,95 |
p < 0,001 |
|
ETH225 |
0,953±0,017 |
115 |
91,27 |
5 |
3,97 |
6 |
4,76 |
p < 0,001 |
|
BM1824 |
0,961±0,013 |
113 |
89,68 |
7 |
5,56 |
6 |
4,76 |
p < 0,001 |
Примечани е. 1 — число образцов, n , 2 — доля образцов, %; QI — индекс качества; р — вероятность корректного генотипирования согласно G. He с соавт. (27).
Как свидетельствует таблица 3, показатель качества генотипирования, оцененного на основании средних значений индекса качества QI, в большинстве локусов (9 из 11 исследованных) был выше 0,950, варьируя от 0,951±0,011 в локусе TGLA122 до 0,995±0,003 в локусе BM2113. В двух локусах (TGLA53 и BM1818) показатель качества генотипирования был значительно ниже (соответственно QI = 0,759±0,030 и QI = 0,700±0,040). Кроме того, доля образцов с качеством генотипирования ниже порогового значения 0,75, для указанных локусов была максимальной и составила 30,16 % для TGLA53 и 30,95 % для BM1818. Вероятность корректного генотипирования для всех исследованных локусов составила p < 0,001.
Оценка результативности генотипирования, проведенная по 11 микросателлитным локусам (табл. 4), показала, что большая доля образцов с отсутствием амплификации выявлена для локусов BM1818 и TGLA53
(соответственно 38,55 % и 25,14 %). Локус BM1818 был успешно генотипи-рован всего у 94 образцов. В другом наименее успешно генотипированном локусе — TGLA53 консенсусные генотипы были получены для 110 образцов, однако общая доля ошибок генотипирования (ER) в этом локусе составила 14,37 %, что более чем в 3 раза превышало среднее значение этого показателя для 11 исследованных локусов. Число повторных ПЦР для успешного получения консенсусного генотипа либо для принятия решения об исключении препарата ДНК из дальнейшего анализа варьировало от 5 до 8, что сопоставимо с результатами аналогичных исследований на образцах с низкой концентрацией или сильной деградацией ДНК (29-31).
Мы предположили, что высокая доля ошибок генотипирования, выявленная в локусах TGLA53 и BM1818, может быть связана с длиной ам-плифицируемых фрагментов, что служит ограничивающим фактором при работе с деградированной ДНК. Расчет коэффициентов корреляции показал положительную корреляцию между длиной аллелей и долей ошибок амплификации, значимую на уровне тенденции ( r 2 = 0,53, p = 0,09). В исследованиях других авторов при анализе ДНК, выделенной из музейных образцов перьев (32) или фрагментов кожи от чучел (33), также выявлена взаимосвязь между размером амплифицируемого фрагмента и успехом амплификации.
В настоящее время основная часть работ зарубежных авторов по изучению полиморфизмов древней ДНК выполняется с применением секвенирования фрагментов мтДНК (34, 35) или полного генома (36), однако STR-маркеры продолжают использоваться для исследования сложных образцов (37). В этом сообщении не представлены результаты проведенного нами анализа мтДНК и полногеномного секвенирования музейных образцов, так как его цель состояла в описании протокола формирования консенсусных генотипов по микросателлитам — типу ДНК-маркеров, который остается наиболее используемым и доступным для большого числа лабораторий.
Поскольку изучение аллелофонда популяций невозможно без получения корректных генотипов, описанный нами алгоритм, обеспечивающий вероятность корректного генотипирования p < 0,001, может быть использован при работе с музейными и другими образцами, содержащими сильно деградированную ДНК. Также отметим, что при исследовании сложных образцов (неинвазивные образцы от диких животных, музейные образцы) важно проводить пилотные исследования, позволяющие предварительно рассчитать вероятность возникновения ошибок генотипирования и их возможное влияние на оценку параметров популяции (26, 27).
Таким образом, предложенная нами модификация метода генотипирования музейных краниологических образцов, в которых ДНК сильно деградирована вследствие длительного хранения, заключалась в использовании мультиплексной панели из 11 микросателлитных маркеров для анализа в одной пробирке с расчетом качества генотипирования и определения консенсусного генотипа для каждого локуса в отдельности вне зависимости от успешности генотипирования других локусов в одном и том же повторе амплификации. Дополнительно оценивалась успешность генотипирования каждого образца на основании среднего индекса качества в первых двух повторах амплификации и принималось решение о целесообразности дальнейшей работы с этим образцом. Описанные модификации позволили снизить стоимость и трудоемкость анализа, поскольку, благодаря количественной оценке успешности генотипирования отдельных локусов и расчету среднего индекса качества для каждого индивидуума, образцы низкого качества удалялись из анализа на начальных этапах, что позволило минимизировать число повторов ПЦР для получения точных данных генотипирования.
Итак, с использованием предложенного нами модифицированного мультиплексного метода удалось получить корректные консенсусные генотипы по STR-маркерам для краниологических образцов крупного рогатого скота, датированных концом XIX—первой половиной ХХ века. Предполагается, что изложенный в статье подход поможет снизить трудоемкость и стоимость анализа исторической ДНК и повысить вовлеченность музейных образцов в исследования взаимосвязей между породами сельскохозяйственных животных.
Список литературы Определение консенсусных генотипов по микросателлитам у музейных краниологических образцов крупного рогатого скота (Вos taurus)
- McHugo G.P., Dover M.J., MacHugh D.E. Unlocking the origins and biology of domestic ani-mals using ancient DNA and paleogenomics. BMC Biol., 2019, 17(1): 98 (doi: 10.1186/s12915-019-0724-7).
- Edwards C.J., Connellan J., Wallace P.F., Park S.D.E., McCormick F.M., Olsaker I., Eythórsdót-tir E., MacHugh D.E., Bailey J.F., Bradley D.G. Feasibility and utility of microsatellite markers in archaeological cattle remains from a Viking Age settlement in Dublin. Animal Genetics, 2003, 34: 410-416 (doi: 10.1046/j.0268-9146.2003.01043.x).
- Chen N., Cai Y., Chen Q., Li R., Wang K., Huang Y., Hu S., Huang S., Zhang H., Zheng Z., Song W., Ma Z., Ma Y., Dang R., Zhang Z., Xu L., Jia Y., Liu S., Yue X., Deng W., Zhang X., Sun Z., Lan X., Han J., Chen H., Bradley D.G., Jiang Y., Lei C. Whole-genome resequencing reveals world-wide ancestry and adaptive introgression events of domesticated cattle in East Asia. Nature Communications, 2018, 9(1): 2337 (doi: 10.1038/s41467-018-04737-0).
- Rowe K.C., Singhal S., Macmanes M.D., Ayroles J.F., Morelli T.L., Rubidge E.M., Bi K., Moritz C.C. Museum genomics: low-cost and high-accuracy genetic data from historical specimens. Molecular Ecology Resources, 2011, 11(6):1082-1092 (doi: 10.1111/j.1755-0998.2011.03052.x).
- Billerman S.M., Walsh J. Historical DNA as a tool to address key questions in avian biology and evolution: A review of methods, challenges, applications, and future directions. Molecular Ecology Resources, 2019, 19(5): 1115-1130 (doi: 10.1111/1755-0998.13066).
- Зиновьева Н.А., Сермягин А.А., Доцев А.В., Боронецкая О.И., Петрикеева Л.В., Абдель-манова А.С., Brem G. Генетические ресурсы животных: развитие исследований аллело-фонда российских пород крупного рогатого скота — мини-обзор. Сельскохозяйственная биология, 2019, 54(4): 631-641 (doi: 10.15389/agrobiology.2019.4.631rus).
- Lari M., Rizzi E., Mona S., Corti G., Catalano G., Chen K., Vernesi C., Larson G., Boscato P., De Bellis G., Cooper A., Caramelli D., Bertorelle G. The complete mitochondrial genome of an 11,450-year-old Aurochsen (Bos primigenius) from Central Italy. BMC Evol. Biol., 2011, 11: 32 (doi: 10.1186/1471-2148-11-32).
- Scheu A., Powell A., Bollongino R., Vigne J.D., Tresset A., Çakırlar C., Benecke N., Burger J. The genetic prehistory of domesticated cattle from their origin to the spread across Europe. BMC Genet., 2015, 16: 54 (doi: 10.1186/s12863-015-0203-2).
- Zhang X., Yang L., Zhao X., Xiang H. The complete mitochondrial genome of an ancient cattle (Bos taurus) from Taosi site, China, and its phylogenetic assessment. Mitochondrial DNA B Re-sources, 2022, 7(5): 804-806 (doi: 10.1080/23802359.2022.2073834).
- Gargani M., Pariset L., Lenstra J.A., De Minicis E., European Cattle Genetic Diversity Consortium, Valentini A. Microsatellite genotyping of medieval cattle from central Italy suggests an old origin of Chianina and Romagnola cattle. Front. Genet., 2015, 6: 68 (doi: 10.3389/fgene.2015.00068).
- Ballard J.W., Whitlock M.C. The incomplete natural history of mitochondria. Mol. Ecol., 2004, 13(4): 729-744 (doi: 10.1046/j.1365-294x.2003.02063.x).
- Глазко В.И., Косовский Г.Ю., Глазко Т.Т., Федорова Л.М. ДНК маркеры и «микро-сателлитный код» (обзор). Сельскохозяйственная биология, 2023, 58(2): 223-248 (doi: 10.15389/agrobiology.2023.2.223rus).
- Brenig B., Schütz E. Recent development of allele frequencies and exclusion probabilities of microsatellites used for parentage control in the German Holstein Friesian cattle population. BMC Genet., 2016, 17: 18 (doi: 10.1186/s12863-016-0327-z).
- Taberlet P., Waits L.P., Luikart G. Noninvasive genetic sampling: look before you leap. Trends in Ecology & Evolution, 1999, 14(8): 323-327 (doi: 10.1016/s0169-5347(99)01637-7).
- Taberlet P., Griffin S., Goossens B., Questiau S., Manceau V., Escaravage N., Waits L.P., Bou-vet J. Reliable genotyping of samples with very low DNA quantities using PCR. Nucleic Acids Research, 1996, 24(16): 3189-3194 (doi: 10.1093/nar/24.16.3189).
- Schlötterer C., Tautz D. Slippage synthesis of simple sequence DNA. Nucleic Acids Research, 1992, 20(2): 211-215 (doi: 10.1093/nar/20.2.211).
- Navidi W., Arnheim N., Waterman M.S. A multiple-tubes approach for accurate genotyping of very small DNA samples by using PCR: statistical considerations. American Journal of Human Genetics, 1992, 50(2): 347-359.
- Abdelmanova A.S., Kharzinova V.R., Volkova V.V., Dotsev A.V., Sermyagin A.A., Boro-netskaya O.I., Chinarov R.Y., Lutshikhina E.M., Sölkner J., Brem G., Zinovieva N.A. Compar-ative study of the genetic diversity of local steppe cattle breeds from Russia, Kazakhstan and Kyrgyzstan by microsatellite analysis of museum and modern samples. Diversity, 2021, 13(8): 351 (doi: 10.3390/d13080351).
- Arandjelovic M., Guschanski K., Schubert G., Harris T.R., Thalmann O., Siedel H., Vigilant L. Two-step multiplex polymerase chain reaction improves the speed and accuracy of genotyping using DNA from noninvasive and museum samples. Molecular Ecology Resources, 2009, 9(1): 28-36 (doi: 10.1111/j.1755-0998.2008.02387.x).
- FAO. Molecular genetic characterization of animal genetic resources: FAO animal production and health guidelines. Rome, Italy, 2011: 68-69. Режим доступа: http://www.fao.org/3/i2413e/i2413e00.pdf. Дата обращения 15.09.2023.
- Абдельманова А.С., Мишина А.И., Волкова В.В., Чинаров Р.Ю., Сермягин А.А., До-цев А.В., Боронецкая О.И., Петрикеева Л.В., Костюнина О.В., Brem G., Зиновьева Н.А. Методы экстракции ДНК из костных образцов крупного рогатого скота, сохраняемых в краниологической коллекции. Сельскохозяйственная биология, 2019, 54(6): 1110-1121 (doi: 10.15389/agrobiology.2019.6.1110rus).
- Mondol S., Karant K.U., Kumar N.S., Gopalaswamy A.M., Andheria A., Ramakrishnan U. Evaluation of non-invasive genetic sampling methods for estimating tiger population size. Biolog-ical Conservation, 2009, 142: 2350-2360 (doi: 10.1016/j.biocon.2009.05.014).
- Modi S., Habib B., Ghaskadbi P., Nigam P., Mondol S. Standardization and validation of a panel of cross-species microsatellites to individually identify the Asiatic wild dog (Cuon alpinus). PeerJ., 2019, 7: e7453 (doi: 10.7717/peerj.7453).
- Miquel C., Bellemain E., Poillot C., Bessière J., Durand A., Taberlet P. Quality indexes to assess the reliability of genotypes in studies using noninvasive sampling and multiple-tube approach. Molecular Ecology Notes, 2006, 6: 985-988 (doi: 10.1111/j.1471-8286.2006.01413.x).
- Broquet T., Petit E. Quantifying genotyping errors in noninvasive population genetics. Molecular Ecology, 2004, 13(11): 3601-3608 (doi: 10.1111/j.1365-294X.2004.02352.x).
- Valière N., Bonenfant C., Toïgo C., Luikart G., Gaillard J.M., Klein F. Importance of a pilot study for non-invasive genetic sampling: genotyping errors and population size estimation in red deer. Conservation Genetics, 2007, 8(1): 69-78 (doi: 10.1007/s10592-006-9149-2).
- Rehnus M., Bollmann K. Non-invasive genetic population density estimation of mountain hares (Lepus timidus) in the Alps: systematic or opportunistic sampling? European Journal of Wildlife Research, 2016, 62: 737-747 (doi: 10.1007/s10344-016-1053-6).
- He G., Huang K., Guo S., Ji W., Qi X., Ren Y., Jin X., Li B. Evaluating the reliability of microsatellite genotyping from low-quality DNA templates with a polynomial distribution model. Chin. Sci. Bull., 2011, 56: 2523-2530 (doi: 10.1007/s11434-011-4634-5).
- Hansen H., Ben-David M., McDonald D.B. Technical advances: Effects of genotyping protocols on success and errors in identifying individual river otters (Lontra canadensis) from their faeces. Molecular Ecology Resources, 2008, 8(2): 282-289 (doi: 10.1111/j.1471-8286.2007.01992.x).
- Zhan X., Zheng X., Bruford M.W., Wei F., Tao Y. A new method for quantifying genotyping errors for noninvasive genetic studies. Conservation Genetics, 2010, 11: 1567-1571 (doi: 10.1007/s10592-009-9950-9).
- Ebert C., Sandrini J., Welter B., Thiele B., Hohmann U. Estimating red deer (Cervus elaphus) population size based on non-invasive genetic sampling. European Journal of Wildlife Research, 2021, 67: 27 (doi: 10.1007/s10344-021-01456-8).
- Sefc K.M., Payne R.B., Sorenson M.D. Microsatellite amplification from museum feather sam-ples: Effects of fragment size and template concentration on genotyping errors. Auk, 2003, 120: 982-989 (doi: 10.1093/auk/120.4.982).
- Polanc P., Sindičić M., Jelenčič M., Gomerčič T., Kos I., Huber Đ. Genotyping success of his-torical Eurasian lynx (Lynx lynx L.) samples. Molecular Ecology Resources, 2012, 2: 293-298 (doi: 10.1111/j.1755-0998.2011.03084.x).
- Jamieson A., Carmagnini A., Howard-McCombe J., Doherty S., Hirons A., Dimopoulos E., Lin A.T., Allen R., Anderson-Whymark H., Barnett R., Batey C., Beglane F., Bowden W., Brat-ten J., De Cupere B., Drew E., Foley N.M., Fowler T., Fox A., Geigl E.M., Gotfredsen A.B., Grange T., Griffiths D., Groß D., Haruda A., Hjermind J., Knapp Z., Lebrasseur O., Librado P., Lyons L.A., Mainland I., McDonnell C., Muñoz-Fuentes V., Nowak C., O'Connor T., Peters J., Russo I.M., Ryan H., Sheridan A., Sinding M.S., Skoglund P., Swali P., Symmons R., Thomas G., Trolle Jensen T.Z., Kitchener A.C., Senn H., Lawson D., Driscoll C., Murphy W.J., Beaumont M., Ottoni C., Sykes N., Larson G., Frantz L. Limited historical admixture between European wildcats and domestic cats. Current Biology, 2023, 33(21): 4751-4760.e14 (doi: 10.1016/j.cub.2023.08.031).
- Uricoechea Patiño D., Collins A., Romero García O.J., Santos Vecino G., Aristizábal Espi-nosa P., Bernal Villegas J.E., Benavides Benitez E., Vergara Muñoz S., Briceño Balcázar I. Unraveling the genetic threads of history: mtDNA HVS-I analysis reveals the ancient past of the Aburra Valley. Genes (Basel), 2023, 14(11): 2036 (doi: 10.3390/genes14112036).
- Chen N., Zhang Z., Hou J., Chen J., Gao X., Tang L., Wangdue S., Zhang X., Sinding M.S., Liu X., Han J., Lü H., Lei C., Marshall F., Liu X. Evidence for early domestic yak, taurine cattle, and their hybrids on the Tibetan Plateau. Science Advances, 2023, 9(50): eadi6857 (doi: 10.1126/sciadv.adi6857).
- Zupanič Pajnič I., Geršak Ž.M., Leskovar T., Črešnar M. Kinship analysis of 5th- to 6th-century skeletons of Romanized indigenous people from the Bled-Pristava archaeological site. Forensic Science International Genetics, 2023, 65: 102886 (doi: 10.1016/j.fsigen.2023.102886).


