Оптимизация технологии генотипирования сои на основе анализа полиморфизма SSR-локусов ДНК
Автор: Рамазанова С.А., Коломыцева А.С.
Рубрика: Селекция и семеноводство сельскохозяйственных растений
Статья в выпуске: 1 (181), 2020 года.
Бесплатный доступ
Соя - основная белково-масличная культура в мире. С каждым годом растет производство соевых бобов. Селекции и семеноводству этой культуры уделяется особое внимание вследствие ее высокого экономического значения. Для характеристики новых сортов, заявленных на выдачу патента, в настоящее время используются морфологические признаки. Современные методы оценки позволяют применять для этих целей уникальные профили ДНК, полученные на основе микросателлитных (SSR) локусов. Целью данной работы было оптимизировать уже существующую технологию идентификации и паспортизации сортов сои. Были выбраны 13 пар SSR-праймеров, локализованных в разных хромосомах сои. Результаты амплификации ДНК 23 сортов сои разных селекцентров показали, что все 13 изученных SSR-локусов полиаллельны. В целом было выявлено 38 аллелей, что в среднем составило 2,9 на локус. Значение индекса информативности маркера (PIC) варьировало от 0,36 до 0,63 и в среднем составило 0,52. Среднее значение эффективного числа аллелей было 2,15...
Соя, микросателлиты, днк, идентификация, паспортизация, генетическое разнообразие
Короткий адрес: https://sciup.org/142223408
IDR: 142223408 | УДК: 633.853.52:575:577.29 | DOI: 10.25230/2412-608X-2020-1-181-42-48
Текст научной статьи Оптимизация технологии генотипирования сои на основе анализа полиморфизма SSR-локусов ДНК
Введение. Соя – важнейшая сельскохозяйственная культура мирового значения. Универсальность ее использования в пищевой, технической, кормовой отраслях, а также способность связывать атмосферный азот определяют широкий интерес к ее производству. Семена сои содержат в среднем 35–45 % белка, 17– 25 % масла и до 30 % углеводов [1]. По мнению многих экспертов, белок сои – это решение проблемы белкового дефицита в мире. Продукты, изготавливаемые из сои, отличаются высокой питательностью, так как наряду с полноценным белком и маслом, они содержат легко усвояемые углеводы, фосфатиды, токоферолы, минеральные элементы, ферменты и витамины, то есть все необходимые для человеческого организма вещества [1].
В России с каждым годом растет производство сои. По данным Росстата, посевные площади, занятые под соей в 2019 г., составили 3 039,4 тыс. га. За год они увеличились на 3,1 % (на 90,2 тыс. га), а за 5 лет – на 51,0 % (на 1 027,0 тыс. га).
Согласно Регламенту, утвержденному Государственной комиссией Российской Федерации по испытанию и охране селекционных достижений, сорта растений, заявленные на выдачу патента, должны отвечать требованиям отличимости, однородности и стабильности (критерий ООС) [2]. В настоящее время для оценки таких критериев используются морфологические признаки, но, как известно, их проявление зависит от условий окружающей среды, а также времени и места проведения испытаний. Для идентификации сортов сои удобно использовать уникальные профили ДНК, полученные с помощью микросателлитных маркеров. Такой метод анализа уже используется для сертификации и паспортизации сортов сои в странах крупнейших экспортеров этой культуры. Это обусловлено тем, что, во-первых, микросателлитные последовательности есть в геномах всех организмов, включая растения, во-вторых, они распределены по всему геному, в-третьих расположены в основном в некодирующих областях и, следовательно, мало подвержены действию естественного отбора [3]. SSR-маркеры подходят также для изучения генетического разнообразия и являются сравнительно недорогим классом ДНК-маркеров. В микросателлитных последовательностях накапливаются мутации, что и обусловливает высокий уровень их полиморфизма. Как правило, они почти всегда имеют кодоминантный тип наследования, это позволяет использовать их для оценки гибридности. Еще одним их преимуществом над другими классами ДНК-маркеров является воспроизводимость [4; 5].
Все эти достоинства микросателлитов способствовали широкому их применению для идентификации и паспортизации генотипов сои во всем мире. Считается, что 4–6 правильно подобранных микро-сателлитных маркеров достаточно для различения сортов сои. Еще в конце прошлого столетия M. Моргант с соавторами, используя семь микросателлитных локусов, получили уникальные ДНК-профили для 61 генотипа сои Glycine max и Glycine soja . Они также показали преимущество SSR-маркеров перед RFLP анализом [6]. С тех пор высокий уровень полиморфизма в SSR-локусах, достаточный для идентификации сортов сои, отмечали многие исследователи из разных стран [7; 8; 9; 10; 11].
С 2008 г. во ВНИИМК (г. Краснодар) проводится идентификация сортов, гибридов и линий сои с использованием восьми микросателлитных локусов [12]. С помощью этой маркерной системы, нами было паспортизировано 108 сортов, гибридов и линий сои (G. max) отечественной и зарубежной селекции, а также три образца G. gracilis, три образца G. soya. В последние годы для всех новых сортов селекции ВНИИМК созданы молекулярно-генетические паспорта [12]. Однако нам не удалось различить два дикорастущих генотипа Соя-93 и Соя-133, а также сорт сои селекции ВНИИМК Ника и реп-лоидную форму, полученную из сорта Вилана. Поэтому целью настоящего исследования было апробирование ряда новых микросателлитных маркеров для паспортизации сортов и гибридов сои селекции ВНИИМК и других селекционных учреждений и установление их пригодности для оптимизации существующей системы молекулярных маркеров на основе SSR-локусов ДНК.
Материалы и методы. В работе изучали 23 генотипа сои ( G. max ) разного происхождения (табл. 1).
Таблица 1
Генотипы сои, использованные для паспортизации
|
Генотип |
Оригинатор |
|
Ирбис |
ФГБНУ ФНЦ ВНИИМК |
|
Барс |
ФГБНУ ФНЦ ВНИИМК |
|
Вита |
ФГБНУ ФНЦ ВНИИМК |
|
77 |
ФГБНУ ФНЦ ВНИИМК |
|
Баргузин |
ФГБНУ ФНЦ ВНИИМК |
|
Иней |
ФГБНУ ФНЦ ВНИИМК |
|
Вилана |
ФГБНУ ФНЦ ВНИИМК |
|
Гном |
ФГБНУ ФНЦ ВНИИМК |
|
Пума |
ФГБНУ ФНЦ ВНИИМК |
|
Чара |
ФГБНУ ФНЦ ВНИИМК |
|
Славия |
ФГБНУ ФНЦ ВНИИМК |
|
Олимпия |
ФГБНУ ФНЦ ВНИИМК |
|
Армавирская 15 |
АОС ВНИИМК |
|
WassBon |
ВИР |
|
Санрайз |
не известен |
|
Пруденс |
University of Guelph, Канада |
|
Венус |
Semences Prograin Inc, Канада |
|
Шуна |
F 1 , RAGT Semens, Франция |
|
Солена |
RAGT Semens, Франция |
|
Палладор |
EURALIS, Франция |
|
ЕСГ 152 |
EURALIS, Франция |
|
Турмалин |
Delley Seeds and Plants, Швейцария |
|
Видра |
Delta Agrar, Сербия |
ДНК выделяли из фрагментов зеленых листьев 5–10 растений или из зародышей семян, применяли модифицированную методику с использованием СТАВ-буфера [13].
Для оптимизации технологии генотипирования сои к уже изученным SSR-локусам Satt 2, Satt5, Satt9, Soypr1, Sat36, Sat43, Soyhsp176 были добавлены еще шесть, выбранные из литературных источников: Soysc514, Satt141, Satt681, Satt181, Satt161 и Sat_263 [6; 7; 8]. Их характеристики и нуклеотидные последовательности представлены в таблице 2.
Таблица 2
Характеристика микросателлитных новых локусов, использованных для паспортизации генотипов сои
|
Локус |
Ло-кализа-ция |
Идентификатор после-дова-тель-ности* |
Иденти-фика-цион-ный номер** |
Повтор |
Нуклеотидные последовательности фланкирующих праймеров |
Размер фраг-мента |
|
Soysc514 |
LG18 |
X56139 |
18745 |
(AT)14 |
AGTCGTAGTCTAGCTACATGAC CAGTGGAATATGTGAAGCAATG |
179 |
|
Satt681 |
C2 |
CC454034 |
31044864 |
(ATT)20 |
GCGGTGCACTTGTCAATCTGTT GCGGTGAGGCATATGTCAGTC |
241 |
|
Sat_263 |
C2 |
CC453798 |
31044628 |
(AT)17 (TC)6 |
GCGGTCGATCGTTTCAATTAGTATG GCGCTGGCAGCCCTTTATTATC |
134 |
|
Satt141 |
D1b |
BH126353 |
14969856 |
(ATT)26 |
CGGTGGTGGTGTGCATAATAA CCGTCATAAAAAGTCCCTCAGAAT |
205 |
|
Satt181 |
H |
BH126389 |
14969892 |
(ATT)18 |
TGGCTAGCAGATTGACA GGAGCATAGCTGTTAGGA |
214 |
|
Satt161 |
C1 |
CC45395 |
31044783 |
(ATT)11 |
GGGTATATCAACATATCTTCAC CTTTTT GGGCTGCTTGTTAATGTTTTGTAGA |
225 |
* – уникальный идентификатор записи последовательности в базе данных GenBank;
** – идентификационный номер нуклеотидной последовательности в базе данных GenBank
Полимеразную цепную реакцию осуществляли в объеме реакционной смеси 25 мкл, содержащей: 67 мМ трис-HCl, рН 8,8; 16,6 мM сульфата аммония; 1,5–3,0 мM MgCl 2 ; 0.01 % Tween 20; по 0,2 мM дезоксирибонуклеозидфосфатов; по 10 пМ праймеров; 10 нг матричной ДНК и одну единицу рекомбинантной термостабильной ДНК полимеразы (НПО «СибЭнзим», Россия). Амплификацию проводили в термоциклере S1000тм (BioRad, США) при следующих температурных режимах: начальная денатурация при 96 оС в течение 2 мин, затем 32 цикла при температурно-временном режиме: денатурация при 94 оС – 30, отжиг праймера при 45–
60 оС, в зависимости от праймера – 40 сек, элонгация при 70 оС – 1 мин, финальная элонгация при 70 оС – 2 мин. Концентрацию ДНК определяли по интенсивности ее окрашивания бромистым этидием в 1%-ном агарозном геле.
Электрофорез продуктов амплификации проводили в агарозном геле (2 % агароза, 1х SB-буфер) с использованием камеры для горизонтального электрофореза (SE.1, ДНК-технология, Россия) в течение 1,5–2 ч при силе тока 50 mA и напряжении 90–100 V. Для локуса Satt161 электрофорез проводили в 8%-ном ПААГ (полиакриламидный гель) в течение 3 ч при силе тока 38–45 mA, 230 V. Документирование результатов электрофореза обеспечивалось при помощи системы цифровой документации видеоизображения BIO-PRINT (Vilber Lourmat, Франция).
Для каждого локуса был вычислен индекс полиморфного информационного содержания (PIC) [4; 14] и эффективное число аллелей [15]. Вычисления проводили по формулам:
PICi = 1 - 2 Рјі², (1)
J=1
где P – частота j паттерна для локуса i и суммирование распространяется на n паттернов .
=
∑ У=1РЛ2
где n e – эффективное число аллелей.
Идентификацию и определение размеров аллелей микросателлитных локусов проводили с использованием программы Bio-Capture (Vilber Lourmat, Франция).
Результаты и обсуждение. Результаты амплификации ДНК 23 сортов сои показали, что все 13 SSR-локусов были поли-аллельны. Всего в этой группе генотипов было выявлено 38 аллелей. Число аллелей на локус варьировало от 1 до 4, что в среднем составило 2,9. Максимальное ко- личество аллелей (4), было выявлено у локусов Satt9, Satt181 и Satt681 (табл. 3).
Таблица 3
Характеристика изученных микросател-литных локусов ДНК сортов сои
|
Локус |
Количество аллелей |
Эффективное число аллелей, n e |
PIC* |
|
Satt2 |
3 |
2,03 |
0,51 |
|
Satt5 |
3 |
2,53 |
0,61 |
|
Satt9 |
4 |
2,71 |
0,63 |
|
Soypr1 |
2 |
1,91 |
0,48 |
|
Soysc514 |
3 |
2,26 |
0,56 |
|
Sat36 |
3 |
2,70 |
0,63 |
|
Sat43 |
2 |
1,57 |
0,36 |
|
Soyhsp176 |
2 |
1,97 |
0,49 |
|
Satt181 |
4 |
2,73 |
0,63 |
|
Satt681 |
4 |
2,17 |
0,54 |
|
Satt141 |
3 |
2,06 |
0,51 |
|
Satt161 |
3 |
1,80 |
0,46 |
|
Satt263 |
2 |
1,56 |
0,36 |
|
Среднее |
2,9 |
2,15 |
0,52 |
*PIC – индекс полиморфного информационного содержания
Одним из показателей, характеризующих информативность локуса, является эффективное число аллелей. Вычисления проводили по формуле 2. Этот показатель характеризует выявленные аллели по частоте их встречаемости в изучаемой выборке генотипов. Количество выявленных аллелей будет равно количеству эффективных, если частоты их встречаемости одинаковы. Для изученных генотипов сои этот показатель варьировал от 1,56 до 2,73. Среднее эффективное число аллелей на локус составило 2,15 (табл. 3).
Важным показателем информативности микросателлитных локусов является индекс полиморфного информационного содержания (PIC). Этот показатель характеризует дискриминационную силу локуса не только по количеству выявленных аллелей, но так же и по относительным частотам их встречаемости [3]. Встречаемость редких аллелей меньше влияет на значение PIC, чем аллели, встречающиеся с высокими частотами. Значения PIC варьируют от 0 до 1. Этот индекс рассчитывался по формуле 1. Для изученных нами SSR-локусов PIC варьирует от 0,36 для Satt263 до 0,63 для локусов Satt9,
Sat36 и Satt181 (табл. 3). Среднее значение индекса полиморфного информационного содержания для изученной группы сортов составило 0,52.
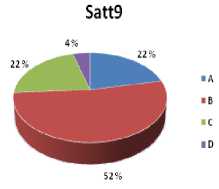
Для всех выявленных аллелей по каждому локусу были вычислены частоты их встречаемости в изученной выборке сортов. На рисунке 1 для примера показаны диаграммы для двух локусов Satt2 и Satt9. По ним у сортов сои селекции ВНИИМК Ирбис и Баргузин выявлены редкие аллели с частотой встречаемости 4 %. У остальных локусов частоты встречаемости аллелей распределены практически равномерно.
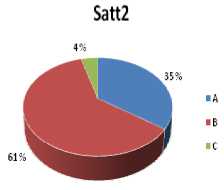
Рисунок 1 – Частоты встречаемости аллелей микросателлитных локусов
Satt2 и Satt9 у 23 генотипов сои:
A, B, C, D – аллели 1, 2, 3, 4 соответственно
Для всех изученных сортов сои селекции ВНИИМК получены уникальные наборы аллелей. Нумерацию аллелей по каждому локусу проводили следующим образом: фрагмент ДНК с максимальным значением молекулярного веса обозначали цифрой 1 и далее по мере уменьшения молекулярного веса цифрами 2, 3 и 4. Например, по локусу Satt181 выявлено 4 аллеля, их ориентировочные размеры от 200 пар нуклеотидов для аллеля 4 до 234 для аллеля 1. Отсутствие фрагмента принимали за нулевой аллель и обозначали цифрой 0. Минимальные различия в наборах аллелей в данной выборке сортов сои наблюдали по двум локусам у сортов Санрайз и Армавирская 15 (по локусам Sat43 и Satt181), Шуна и ЕСГ 152 (по локусам Satt5 и Sat43) (табл. 4).
Таблица 4
Полиморфизм микросателлитных локусов
ДНК сортов сoи
|
Сорт |
Локус |
||||||||||||
|
Satt 2 |
Satt 5 |
Satt 9 |
Soypr 1 |
Soysc 514 |
Sat 36 |
Sat 43 |
Soyhsp 176 |
Satt 181 |
Satt 681 |
Satt 141 |
Satt 161 |
Satt 263 |
|
|
Ирбис |
1 |
1 |
4 |
2 |
2 |
2 |
2 |
3 |
0 |
2 |
2 |
3 |
1 |
|
Барс |
1 |
1 |
2 |
2 |
2 |
3 |
1 |
3 |
2,3 |
3 |
2 |
3 |
1 |
|
Вита |
2 |
1 |
1 |
1 |
1 |
1 |
1 |
2 |
3 |
2 |
1 |
3 |
1 |
|
WassBon |
2 |
2 |
3 |
1 |
2 |
2 |
2 |
2 |
3 |
2 |
3 |
1 |
1 |
|
77 |
2 |
2 |
2 |
2 |
1 |
2 |
2 |
2 |
2 |
0 |
1 |
1 |
1 |
|
Баргузин |
3 |
1 |
2 |
2 |
2 |
1 |
2 |
2 |
2 |
3 |
2 |
2 |
1 |
|
Иней |
1 |
2 |
2 |
2 |
1 |
2 |
2 |
2 |
2 |
3 |
2 |
1,2 |
1 |
|
Вилана |
2 |
2 |
2 |
1 |
1 |
2 |
2 |
3 |
2 |
2 |
2 |
2 |
1 |
|
Гном |
1 |
1 |
3 |
1 |
2 |
1 |
2 |
2 |
2 |
3 |
2 |
3 |
2 |
|
Санрайз |
2 |
2 |
1 |
1 |
2 |
2 |
1 |
3 |
3 |
2 |
3 |
3 |
1 |
|
Пруденс |
2 |
1 |
2 |
1 |
2 |
3 |
2 |
3 |
2 |
2 |
2 |
3 |
1 |
|
Шуна |
2 |
3 |
2 |
1 |
2 |
3 |
2 |
3 |
2 |
3 |
2 |
3 |
1 |
|
Палла-дор |
1 |
3 |
3 |
2 |
2 |
1 |
1 |
3 |
3 |
3 |
2 |
2 |
1 |
|
ЕСГ152 |
2 |
2 |
2 |
1 |
2 |
3 |
1 |
3 |
2 |
3 |
3 |
3 |
1 |
|
Армавирская 15 |
2 |
2 |
1 |
1 |
2 |
2 |
2 |
3 |
2 |
2 |
2 |
3 |
1 |
|
Турмалин |
2 |
1 |
2 |
1 |
2 |
1 |
1 |
2 |
1 |
3 |
2 |
3 |
1 |
|
Солена |
2 |
2 |
2 |
1 |
2 |
2 |
2 |
2 |
1 |
3 |
1 |
3 |
1 |
|
Пума |
2 |
1 |
2 |
1 |
1 |
2 |
2 |
3 |
2 |
3 |
2 |
3 |
1 |
|
Чара |
1 |
2 |
2 |
2 |
1 |
2 |
2 |
2 |
1 |
3 |
2 |
1 |
2 |
|
Славия |
1 |
1 |
3 |
2 |
1 |
3 |
2 |
2 |
0 |
2 |
2 |
3 |
1 |
|
Олимпия |
2 |
2 |
1 |
1 |
2 |
3 |
2 |
3 |
2 |
3 |
3 |
3 |
2 |
|
Венус |
1 |
1 |
3 |
2 |
2 |
2 |
2 |
2 |
1 |
2 |
2 |
3 |
2 |
|
Видра |
2 |
3 |
1 |
1 |
2 |
2 |
1,2 |
3 |
1 |
2 |
1 |
3 |
1 |
На основании полученных данных о частоте встречаемости аллелей и их размере была проведена оценка степени генетического родства изученных генотипов сои. Чтобы оценить расстояния между кластерами использовали методы дисперсионного анализа. В качестве расстояния между кластерами брали прирост суммы квадратов расстояний объектов до центра кластера (метод Варда). Полученная дендрограмма представлена на рисунке 2.
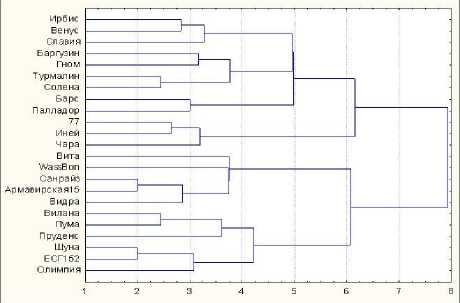
Рисунок 2 – Дендрограмма генетического сходства 23 генотипов сои на основе анализа полиморфизма 13 SSR-локусов ДНК
Все генотипы сои распределились в два крупных кластера, которые включают как сорта селекции ВНИИМК, так и сорта и гибриды других селекцентров. Максимальный уровень сходства между генотипами составил 7,9, минимальный – 2. Наиболее близкое генетическое родство наблюдается между гибридом Шуна (RAGT Semens, Франция) и сортом ЕСГ-152 (EURALIS, Франция), а так же сортами Армавирская 15 (АОС ВНИИМК) и Санрайз (оригинатор неизвестен).
Заключение. В результате проведенной работы было установлено, что дискриминационный потенциал изученной маркерной системы достаточно высок для того, чтобы использовать ее для идентификации и паспортизации сортов сои. В результате оптимизации существующей системы молекулярных маркеров были идентифицированы 23 генотипа сои, в том числе 12 сортов сои селекции ВНИ-ИМК. Для каждого из них были получены уникальные наборы аллелей. На основании полученных данных об аллельном разнообразии изученных микросателлит-ных локусов разработаны индивидуальные молекулярно-генетические паспорта.
Список литературы Оптимизация технологии генотипирования сои на основе анализа полиморфизма SSR-локусов ДНК
- Петибская В.С., Баранов В.Ф., Кочегура А.В., Зеленцов С.В. Соя: качество, использование, производство. - М.: Аграрная наука, 2001. - 64 с.
- Гражданский кодекс Российской Федерации: Части первая - четвертая: [Принят Гос. Думой 23 апреля 1994 года, с изменениями и дополнениями по состоянию на 10 апреля 2009 г.] // Собрание законодательства РФ. - 1994. - № 22. - Ст. 1438.
- Hamada H., Petrino M.C., Takugana T. A novel repeated element with Z-DNA-forming potential is widely found in evolutionarily diverse eukaryotic genomes // PNAS. - 1982. -Vol. 79. - No 23. - P. 6465-6469. DOI: 10.1073/pnas.79.21.6465
- Использование ПЦР-анализа в генетикоселекционных исследованиях: научно-методическое руководство / Под. ред. Ю.М. Сиволапа. - Киев: Аграрна наука, 1998. - 156 с.
- Varshney R.K., Graner A., Sorrells M.E. Genic microsatellite markers in plants: Features and applications // TRENDS in Biotechnology. -2005. - Vol. 23. - No 1. - P. 48-55.
- Morgante M., Rafalski A., Biddle P., Tingey S., Olivieri A.M. Genetic mapping and variability of seven soybean simple sequence repeat loci // Genome. - 1994. - V. 37. - No 5. -P. 763-769.
- Rongven J., Akkaya M.S., Bhagwat A.A., Lavi U., Cregan P.B. The use of microsatellite DNA markers for soybean genotype identification // Theor. Appl. Genet. - 1995. - V. 90. - P. 43-48.
- Абугалиева C.H., Волкова Л.А., Нурпанова А.А., Жанпеисова А.С., Туруспеков Е.К. ДНК-фингерпринтинг сортов сои Казахстана с использованием SSR-маркеров // Биотехнология. Теория и практика. - 2013. - № 3. -С. 26-34.
- Hudcovicovä M., Kraic J. Utilisation of SSRs for Characterisation of the Soybean (Glycine max (L.) Merr.) Genetic Resources // Czech Journal of Genetics and Plant Breeding. - 2003. -V. 39 (4). - P. 120-126.
- DOI: 10.17221/3729-CJGPB
- Bisen A., Khare D., Nair P., Tripathi N. SSR analysis of 38 genotypes of soybean (Glycine max (L.) Merr.) genetic diversity in India // Physiol. Mol. Biol. Plants. - 2015. - V. 21 (1). -P. 109-115.
- DOI: 10.1007/s12298-014-0269-8
- Kujane K., Sedibe M.M., Mofokeng A. Genetic diversity analysis of soybean (Glycine max (L.) Merr.) genotypes making use of SSR markers // Australian Journal of Crope Science. -2019. - V. 13 (07) - P. 1113-1119.
- DOI: 10.21475/ajcs.19.13.07.p1638
- Рамазанова С.А. Идентификация генотипов сои разного происхождения с использованием полиморфизма девяти микросателлитных локусов ДНК // Современные проблемы селекции и технологии возделывания сои: сб. статей 2-й международной конференции по сое. - Краснодар, 910 сентября 2008 г. - С. 129-136.
- Saghai-Maroof M.A., Soliman K.M., Jorgensen R.A., Allard R.W. Ribosomal DNA spacer-length polymorphisms in barley: Mendelian inheritance, chromosomal location, and population dynamics // PNAS USA. - 1984. - 81. - P. 8014-8018.
- Botstein D., White R.L., Skolnick M., Davis R. W. Construction of a genetic linkage map in man using restriction fragment length polymorphisms // Am. J. Hum. Genet. - 1980. - V. 32. - P. 314-331.
- Pejic I., Ajmorne-Marsan P., Morgante M. [et al.]. Comparative analysis of genetic similarity among maize inbred lines detected by RFLPs, RAPDs, SSRs and AFLPs // Theor. Appl. Genet. - 1998. - V. 83. - P. 194-200.


