Оптимизация выделения ДНК заразихи (Orobanche cumana Wallr.) и выявление полиморфных RAPD и SSR-локусов для генотипирования паразита
Автор: Гучетль С.З., Челюстникова Т.А., Антонова Т.С.
Рубрика: Селекция и семеноводство
Статья в выпуске: 2 (155-156), 2013 года.
Бесплатный доступ
Оптимизирована методика выделения ДНК из свежих и высушенных тканей растений заразихи Orobanche cumana Wallr., находящихся на разных стадиях онтогенеза. Наиболее оптимальным было признано выделение ДНК из свежих замороженных соцветий методом Doyle J.J. and J.L. Doyle (1987) с добавлением 1 % поливинилпирролидона в экстракционный буфер. Отобрано три RAPD- и 10 SSR-праймеров, инициирующих амплификацию полиморфных фрагментов ДНК. Выявлены стабильно воспроизводящиеся фрагменты амплифицированной ДНК, которые могут служить надежными маркерами для изучения меж- и внутрипопуляционной изменчивости популяций паразита юга России.
Заразиха, подсолнечник, выделение днк, rapd- и ssr-локусы
Короткий адрес: https://sciup.org/142151143
IDR: 142151143 | УДК: 633.854.78:631.523
Текст научной статьи Оптимизация выделения ДНК заразихи (Orobanche cumana Wallr.) и выявление полиморфных RAPD и SSR-локусов для генотипирования паразита
Заразиха ( Orobanche cumana Wallr.) – облигатный паразит из высших растений, паразитирующий на корнях подсолнечника. Потери урожая на зараженных семенами заразихи полях составляют от 50 до 90 % [1]. Интенсивное возделывание подсолнечника с нарушением севооборота во многих странах привело к формированию высоковирулентных биотипов заразихи, преодолевших устойчивость возделываемого сортимента как в России, так и за рубежом. Поэтому мировое сообщество проводит исследования, цель которых – создание новых источников устойчивости к самым вирулентным расам, а также изучение генетической структуры популяций заразихи и их эволюции для развития долгосрочной стратегии контроля над этим паразитом.
Вариабельность ДНК заразихи, выявляемая методами полимеразной цепной реакции, может являться достаточно точным инструментом для изучения внутривидового полиморфизма O. cumana и её происхождения. Исследователями из разных стран уже изучаются молекулярно-генетические различия как между разными видами рода Orobanche [2; 3], так и межпопуляционные и внутрипопуляци-онные различия вида O. cumana [1; 4; 5; 6]. Для создания системы ДНК маркеров, выявляющих внутривидовые различия, необходимо оптимизировать методы выделения ДНК, поскольку не существует единого оптимального метода экстракции ДНК для всех видов растений вообще и для заразихи, в частности. Так, Gagne с соавторами [6] выделяли ДНК из замороженных верхушек цветоносов заразихи методом Rogers and Bendich [7]. Atanasova с соавторами (2005) использовали методику выделения ДНК из навески семян заразихи методом Fulton et al. (1995) с модификациями Joel et al. [4]. Dimitrijević с соавторами (2013) были апробированы 10 прописей экстракции ДНК из сухих стеблей заразихи и два из них рекомендованы к применению: DNeasy® Plant Mini Kit, Qiagen и метод Rogers and Bendich [8]. С помощью Nucleon Phytopure DNA extraction kit (Amer-sham, Buchinghamshire, UK) была экстрагирована ДНК из побегов и соцветий заразихи в работе Benharrat et al. [5].
Для создания системы молекулярных маркеров на основе локусов ДНК необходимо также проведение скрининга разного типа праймеров, выявляющих полиморфизм фракций ДНК. Gagne с соавторами (1998) выполнили скрининг 177 RAPD праймеров для определения генетической изменчивости популяций O. cumana , происходящих из Болгарии, Испании, Румынии и Турции. 23 праймера показали воспроизводимость результатов [6]. RAPD технология также была использована для оценки генетической вариабельности 10 популяций заразихи O. cumana из Испании, Югославии и Румынии [1]. Pineda-Martos с соавторами (2013) выполнили скрининг 217 SSR праймеров. Из них отобраны 15 полиморфных и высокопродуктивных для изучения меж- и внутрипопуляционной изменчивости популяций испанской заразихи подсолнечника [9]. В нашей предыдущей работе, с целью изучения возможности молекулярно-генетической идентификации разных рас заразихи и характеристики её популяций, были испытаны 48 праймеров для ПЦР. Лишь два
RAPD праймера из испытанных выявили чёткий, воспроизводимый полиморфизм у разных образцов заразихи [10]. Анализ опубликованных работ показывает, что на выявление полиморфизма фрагментов ДНК заразихи разного происхождения работают разные праймеры и необходимо проведение сложных и объемных предварительных исследований для обеспечения надежности получаемых результатов.
Целью данной работы было оптимизировать методику выделения ДНК из разных органов растений заразихи, находящейся на разных этапах онтогенеза и выявить стабильный, воспроизводимый полиморфизм амплифицированных фрагментов для создания системы молекулярных маркеров, пригодных для изучения меж- и внутрипопуляционной изменчивости паразита из южных регионов России.
Материалы и методы. Материалом для исследования служили образцы заразихи из 4 популяций. Семена и сухие стебли заразихи были собраны в 2012 г. на полях подсолнечника Краснодарского, Ставропольского краёв и Ростовской области (табл. 1). Свежие клубеньки, побеги и соцветия заразихи выращены в теплице, в условиях, описанных ранее [11].
Таблица 1
Происхождение заразихи, использованной для анализа
|
Место сбора семян заразихи |
Гибрид подсолнечника |
|
Краснодарский край, Крыловской район |
Мегасан |
|
Ставропольский край, Труновский район |
Не известен |
|
Ростовская область, Азовский район (ДОС ВНИИМК) |
-//- |
|
Ростовская область, Милютинский район |
Тунка |
Для анализа брали различные органы растений заразихи, находящихся на разных этапах онтогенеза. ДНК экстрагировали из навесок ткани от 50 до 200 мг растительного материала с использованием 9 вариантов, комбинирующих метод экстракции и тканевый материал (табл. 2).
Для ПЦР анализа применяли 26 RAPD праймеров (табл. 3), 15 SSR (табл. 4) и один ISSR [10].
Методы, использованные для выделения ДНК из сухого и свежего материала заразихи
|
Номер варианта |
Метод выделения |
Используемый материал заразихи |
|
1 |
Diamond DNA Plant Kit D (Россия) |
Сухие стебли |
|
2 |
Методика Doyle J.J. and J.L. Doyle, 1987 с добавлением 1 % поливинил-пирролидона в экстракционный буфер [12] |
Сухие стебли |
|
3 |
Методика Doyle J.J. and J.L., Doyle, 1987, с добавлением 1 % поливинилпирролидона в экстракционный буфери дополнительной обработкой фенолом |
Сухие стебли |
|
4 |
Методика Saghai -Maroof et al., 1984, модифицированная Гучетль и др., 2007 [13;14] |
Сухие верхние цветочки с коробочками и семенами |
|
5 |
Методика Saghai -Maroof et al., 1984, модифицированная Гучетль и др., 2007 |
Сухие семена |
|
6 |
Методика Saghai- Maroof et al., 1984, модифицированная Гучетль и др., 2007 |
Свежие замороженные соцветия |
|
7 |
Методика Doyle J.J. and J.L., Doyle, 1987, с добавлением 1% поливинил-пирролидона в экстракционный буфер |
Свежие замороженные соцветия |
|
8 |
Методика Doyle J.J. and J.L., Doyle, 1987 с добавлением 1 % поливинил-пирролидона в экстракционный буфер |
Свежие замороженные клубеньки |
|
9 |
Методика Дж. Дрейпер, Р. Скотт [15] |
Свежие замороженные соцветия |
Для проведения полимеразной цепной реакции использовали 25 мкл реакционной смеси следующего состава: 67 мМ трис-HCl, рН 8,8; 16,6 мM сульфата аммония; 1,5–3 мM MgCl2; 0,01 % Tween 20; по 0,2 мM дезоксирибонуклеозидфосфатов; по 10 пМ праймеров; 10 нг матричной ДНК и 1 ед. рекомбинантной термостабильной ДНК полимеразы (Москва, Сибэнзим). Для амплификации применяли термоциклер S1000тм (BioRad, США). Условия амплификации: начальная денатурация 2 мин 96 °С, затем 30 циклов при соблюдении температурновременного режима: денатурация при 94 °С – 30 сек, отжиг при N °С в течение 30– 60 сек, элонгация – 1–2 мин при 70 °С, финальная элонгация – 2 мин, где N – температура отжига, подбираемая экспериментальным путем, исходя из теорети- ческой температуры отжига праймеров.
Электрофорез продуктов амплификации проводили в агарозном геле (1–2 % агароза, 1хТАЕ-буфер) с использованием камеры SE-2 для горизонтального электрофореза (Хеликон, РФ) и полиакриламидном геле (8 %, 1хТБЕ) с использованием камеры VE-20 для вертикального электрофореза (Хеликон, РФ). Последующее окрашивание осуществляли бромистым этидием. Документирование результатов электрофореза обеспечивалось при помощи системы цифровой документации видеоизображения BIO-PRINT (Vilber Lourmat, Франция).
Результаты и обсуждение. ДНК удавалось выделить при использовании восьми вариантов, кроме девятого. Качество выделенной ДНК было более или менее удовлетворительным при визуальной оценке (рис. 1). Активность ДНК, выделенной с помощью варианта 1 (рис. 1а), была или слабой, или её не было вообще. Вариант 3 отличался от 2-го дополнительной обработкой фенолом, которая улучшила качество ДНК (рис. 1в, 1б).
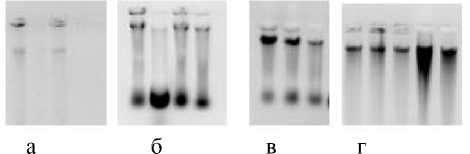
д е ж з
Рисунок 1 – Выделение ДНК различными методами: варианты:
1 – а; 2 – б; 3 – в; 4 – г; 5 – д;
6 – е; 7 – ж; 8 – з
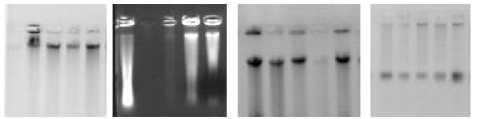
Чтобы определить, может ли выделенная ДНК быть пригодной для проведения ПЦР, были выполнены реакции амплификации с несколькими праймерами (рис. 2).

а б в г
д е ж з
Рисунок 2 – Электрофоретические спектры продуктов амплификации ДНК заразихи с праймерами: а – 1*– GACA4; б – 2*–
GACA4; в – 3 – GACA4 (отжиг у образца № 3); г – 4 – UBC215; д – 5 – Ocum 59; е – 6 – G06; ж – 7 – GACA4; з – 8 – GACA4* – варианты 1, 2, 3, 4, 5, 6, 7, 8
ДНК заразихи, выделенная при помощи вариантов 1 и 2 амплифицировалась с праймером GACA4, но не производила продуктов реакции при амплификации с RAPD праймерами Primer1, UBC 250 и UBC 215. ДНК заразихи, выделенная при помощи варианта 5, не производила продуктов реакции при амплификации с RAPD праймером UBC 215 и ISSR праймером GACA4. Эти методики основаны на выделении ДНК из высушенного растительного материала, и, возможно, что данные праймеры не гибридизируются с выделенной ДНК из-за её загрязненности полисахаридами, полифенолами и белками. Дополнительная обработка ДНК фенолом (вариант 3) немного улучшила качество ДНК и привела к отжигу RAPD праймеров. SSR и ISSR праймеры, по-видимому, не так чувствительны к качеству ДНК, поэтому гибридизировались с матричной ДНК практически во всех вариантах. Варианты 6–9 – выделение ДНК из свежих замороженных тканей заразихи. Как уже отмечалось выше, вариант 9
не привел к выделению ДНК, и в дальнейшем ПЦР с ней не проводилась. В варианте 6 амплифицированные фракции ДНК были плохого качества, и отжиг со стоялся не у всех образцов (рис. 2е). Наилучшее качество выделенной ДНК и амплифицированных на ней фрагментов получено при использовании варианта 7. Таким образом, для дальнейших исследований был выбран вариант 7, а именно выделение ДНК из свежих замороженных соцветий по методике Doyle J.J. and J.L. Doyle (1987) с добавлением 1 % поливи-нилпирролидона в экстракционный буфер. При необходимости работы с сухим (гербарным) материалом заразихи наиболее целесообразно использование вариантов 3 и 4, позволяющих работать со всеми типами молекулярных маркеров.
Для определения маркеров молекулярно-генетических различий у заразихи из популяций различной географической локализации использовали ДНК, выделенную по прописям вариантов 7 и 8. Проведены реакции с 26 RAPD (табл. 3) и 15 SSR праймерами (табл. 4). Отбор образцов заразихи из популяций осуществлялся по принципу наибольшей географической отдаленности последних друг от друга. Для экстракции ДНК заразихи из каждой популяции была взята смесь из пяти индивидуальных растений равного веса. Эти меры должны были обеспечить выявление полиморфизма молекулярных локусов в случае его наличия.
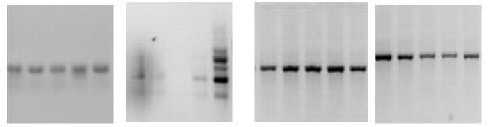
Как отражено в таблице 3, из 26 анализированных при ПЦР RAPD праймеров шесть не гибридизировались с матричной ДНК, 17 не выявили полиморфизм исследуемых популяций, и только три (UBC 72, UBC 252, UBC 292) выявили полиморфизм (рис. 3).
Характеристика RAPD-локусов, использованных для оценки генетических различий у O. cumana из разных популяций
|
RAPD праймер |
Последовательность фланкирующих праймеров 5’-3’ |
Наличие или отсутствие полиморфизма |
|
UBC 17 |
CCT GGG CCT C |
- |
|
UBC 72 |
GAG CAC GGG A |
+ |
|
UBC 85 |
GTG CTC GTG C |
Нет отжига |
|
UBC 146 |
ATG TGT TGC G |
- |
|
UBC 159 |
GAG CCC GTA G |
Нет отжига |
|
UBC 168 |
CTA GAT GTG C |
Нет отжига |
|
UBC 213 |
CAG CGA ACT A |
- |
|
UBC 252 |
CTG GTG ATG T |
+ |
|
UBC 268 |
AGG CCG CTT A |
Нет отжига |
|
UBC 272 |
AGC GGG CCA A |
- |
|
UBC 292 |
AAA CAG CCC G |
+ |
|
UBC 299 |
TGT CAG CGG T |
Нет отжига |
|
UBC 305 |
GCT GGT ACC C |
- |
|
UBC 313 |
ACG GCA GTG G |
- |
|
UBC 328 |
ATG GCC TTA C |
Нет отжига |
|
UBC 353 |
TGG GCT CGC T |
- |
|
UBC 358 |
GGT CAG GCC C |
- |
|
UBC 363 |
ATG ACG TTG A |
- |
|
UBC 377 |
GAC GGA AGA G |
- |
|
UBC 383 |
GAG GCG CTG C |
- |
|
UBC 215 |
TCACACGTGC |
- |
|
UBC 250 |
CGACAGTCCC |
- |
|
Primer 1 |
GGTGCGGGAA |
- |
|
Primer 4 |
AAGAGCCCGT |
- |
|
Primer 6 |
CCCGTCAGCA |
- |
|
OPG 06 |
GTGCCTAACC |
- |
Примечание: (–) – нет полиморфизма; (+) – есть полиморфизм
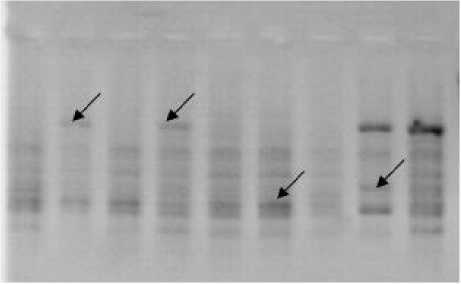
Рисунок 3 – Электрофоретические спектры продуктов амплификации ДНК образцов заразихи с праймером UBC 72.
Стрелками обозначены полиморфные фрагменты ДНК
Эти три спектра амплифицированной ДНК включали 3–6 фрагментов на праймер (в среднем, 4,6 фракций, генерируемых при использовании одного прайме- ра). Среднее число полиморфных фрагментов на праймер составляет два. Процент полиморфных локусов составил 11,5. Как сказано выше, Gagne с соавторами (1998) [6] был выполнен скрининг 177 RAPD праймеров при определении генетической изменчивости популяций O. cumana, происходящих из Болгарии, Испании, Румынии и Турции, 19,6 % из которых выявили полиморфизм. То есть полученные в обоих случаях цифры примерно соответствуют друг другу.
В таблице 4 представлены последовательности 15 пар праймеров, синтезированных для выявления полиморфизма микросателлитных локусов заразихи. Они были отобраны Pineda-Martos et al. (2013) из изученных ими 217 SSR праймеров и характеризовались как высокопродуктивные, для изучения меж- и внутрипопуля-ционной изменчивости заразихи, паразитирующей на подсолнечнике в Испании [9] .
Таблица 4
Характеристика микросателлитных локусов ДНК при оценке генетических различий у O. cumana из разных популяций
|
Локус |
Последовательность фланкирующих праймеров 5’-3’ |
Повтор |
Количество аллелей |
|
Ocum-52 |
CATGTCTAAGCTTTTGGCTCG |
(AG) 10 |
2 |
|
CAAGACTTGGAACAAGCAAATC |
|||
|
Ocum-59 |
TCTTGATTTGTATATGTCTGATGCAAT |
(TC) 11 |
2 |
|
ATGCTACAATAGAAATACACAACGAAC |
|||
|
Ocum-70 |
AAGCTGTAAACAATGCCTGAA |
(TG) 11 |
Нет отжига |
|
CCTCCTCCAGTACCACTAGGC |
|||
|
Ocum-74 |
CCTAAAATTGAAACCTTAAGGAAA |
(GA) 12 |
3 |
|
ACTTTCCGTGAGACGGAGTC |
|||
|
Ocum-75 |
TGTGGATAGAGTATAAGCTACCAGTTC |
(CA) 12 |
Нет отжига |
|
TTCCCGTAGCTTGGAGAATG |
|||
|
Ocum-81 |
TTACAAGGTGAAACCACCCA |
(CA) 13 |
3 |
|
CAGCTACTGTCCGCAAGAAA |
|||
|
Ocum-87 |
TTCTCGACAGCTTTGGGTAAA |
(TTC) 13 |
4 |
|
ATGCCAACTTCGAGTGATCC |
|||
|
Ocum-108 |
TCGTTAATAAGTGGTTCACGAAAA |
(GTAT) 6 |
2 |
|
TGACTAAAAATAAAATGTACGGGTG |
|||
|
Ocum-122 |
GGAATACATCATTAAAGTAGTTGTCCG |
(AGTGTG) 6 |
2 |
|
GAAGGAGTCATTAAACTCCGTGA |
|||
|
Ocum-141 |
CAGCAACTGTTTCTTCCATAGAG |
(CTT) 6 |
2 |
|
TCCAAGAAGAGGAAAAGAAGTGA |
|||
|
Ocum-160 |
TGAGGGTTTGTAAAGTGGGC |
(AG) 7 |
1 |
|
CGTACCTTATCCCTCCGTCA |
|||
|
Ocum-174 |
CAACCAACAAACAAGTAGTGACG |
(AAG) 7 |
1 |
|
TCTTGCGGCAAAACCATT |
|||
|
Ocum-196 |
GTATGTGCGCCCGTCTTG |
(GT) 7 |
2 |
|
GGGGATGACTGTGTTTCGAT |
|||
|
Ocum-197 |
AGAGACGGCATCATCAATCA |
(GA) 7 |
3 |
|
GTGATCGTGCAGGCACCTA |
|||
|
Ocum-206 |
CCGATTGCTGTTTATGTTGTATT |
(TG) 8 |
Нет отжига |
|
TGTAGGAGATGCCCAGTTCA |
На ДНК заразихи из российских популяций 3 праймера не выявили спектров амплифицированной ДНК, 2 не обнаружили полиморфизм и 10 показали полиморфизм (рис. 4).
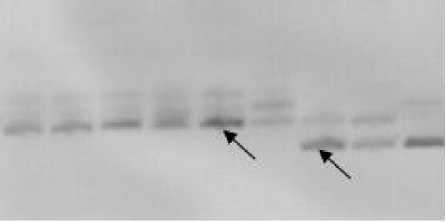
Рисунок 4 – Электрофоретические спектры продуктов амплификации ДНК у образцов заразихи с праймером Ocum 197.
Стрелками обозначены полиморфные фрагменты ДНК
Количество полученных аллелей для полиморфных локусов варьировало от двух до четырех (табл. 4), среднее число аллелей на локус составило 2,5. Процент полиморфных локусов – 67.
Полученные результаты показали, что из двух типов маркеров, продуцируемых RAPD- и SSR-праймерами, процент полиморфных фрагментов ДНК и среднее число аллелей на локус у микросателлитов выше, чем у RAPD. Но вместе с тем обе системы молекулярных маркеров пригодны для изучения меж- и внутрипо-пуляционной изменчивости заразихи из регионов юга России.
Заключение. Таким образом, наиболее оптимальным было выделение ДНК из свежих замороженных соцветий методом Doyle J.J. and J.L. Doyle (1987) [12] с добавлением 1 % поливинилпирролидона в экстракционный буфер. При необходимости работы с сухим материалом заразихи более подходящими были методика выделения ДНК, разработанная Doyle J.J. and J.L. Doyle (1987) [12], но с добавлением 1 % поливинилпирролидона в экстракционный буфер и дополнительной обработкой фенолом, и методика Saghai-
Maroof et al. (1984), модифицированная Гучетль и др. (2007) [13; 14].
Выделены три RAPD- и 10 SSR-праймеров, инициирующих амплификацию полиморфных фрагментов ДНК у заразихи, паразитирующей на подсолнечнике в южных регионах России, в частности в Краснодарском крае.
Исследования выполнены при финансовой поддержке РФФИ и Администрации Краснодарского края, грант № 13-04-96521.


