Опыт создания публичной базы данных мутаций oncoBRCA: биоинформационные проблемы и решения
Автор: Никитин Алексей Георгиевич, Бровкина Ольга Игоревна, Ходырев Дмитрий Сергеевич, Гусев Олег Александрович, Гордиев Марат Гордиевич
Журнал: Клиническая практика @clinpractice
Рубрика: Фундаментальная медицина
Статья в выпуске: 1 т.11, 2020 года.
Бесплатный доступ
Обоснование. Развитие наследственных онкологических синдромов обусловлено генетическими нарушениями в системе репарации ДНК, состоящей более чем из 100 генов. Однако в настоящее время в большинстве медицинских центров России диагностика наследственных заболеваний раком яичника и раком молочной железы сводится к определению наиболее частых мутаций (8 точек) в генах BRCA1 и BRCA2 с помощью методов полимеразной цепной реакции. При этом данные мутации являются частыми для славянской популяции, в то время как в остальных популяциях России они встречаются реже или не встречаются вообще. Цель исследования - получить представление о ландшафте наследственных патогенных вариантов в генах системы репарации; разработать методы быстрого анализа данных полногеномного секвенирования. Методы. Методом секвенирования нового поколения (Next Generation Sequencing, NGS) была проанализирована панель из 34 генов системы репарации в 1644 образцах биоматериала пациентов с наследственными онкологическими синдромами...
Онкогенетика, наследственные синдромы, секвенирование, рак
Короткий адрес: https://sciup.org/143170829
IDR: 143170829 | DOI: 10.17816/clinpract25860
Текст научной статьи Опыт создания публичной базы данных мутаций oncoBRCA: биоинформационные проблемы и решения
ОБОСНОВАНИЕ
Рак яичников и рак молочной железы являются основными причинами онкологической смертности среди женщин во всем мире [1]. Оба вида рака высокогетерогенные с сильным наследственным компонентом: примерно 10–15% случаев рака яичников и 5–7% случаев рака молочной железы являются наследственными [2]. Ранняя диагностика снижает смертность, связанную с этими заболеваниями, поэтому генетическое тестирование на предрасположенность к наследственным онкосиндромам будет полезным дополнением к рутинной клинической практике.
Развитие наследственных рака молочной железы и рака яичников обусловлено генетическими нарушениями в системе репарации ДНК [3]. В настоящее время в большинстве медицинских центров Российской Федерации диагностика наследственного рака молочной железы представляет собой определение наиболее частых мутаций (8 точек) в генах BRCA1 , BRCA2 с помощью методов полимеразной цепной реакции (ПЦР), хотя в мировой литературе описано более 1000 мутаций гена BRCA1 , и во многих популяциях присутствует свой собственный набор частых мутаций. Поэтому существующий на данный момент отечественный подход к генетической диагностике рака яичников и рака молочной железы выявляет только мутации, характерные для славянских популяций, населяющих территорию России [4, 5], и не учитывает остальные мутации в генах BRCA1 / BRCA2 , а также
других генах системы репарации, обнаружение которых позволило бы выбрать оптимальную тактику лечения [6]. Население Российской Федерации состоит более чем из ста этнических групп, что дает уникальную возможность изучения наследственных патогенных мутаций.
После внедрения в клиническую практику ингибиторов поли(АДФ-рибоза)-полимеразы (poly ADP ribose polymerase, PARP) открылись новые возможности в лекарственной терапии ряда онкологических заболеваний, обусловленных нарушениями в генах системы репарации двухцепочечных разрывов ДНК, ряд препаратов получил одобрение Управления по санитарному надзору за качеством пищевых продуктов и медикаментов США (Food and Drug Administration, FDA) — ни-рапариб (Niraparib, Tesaro), рукапариб (Rucaparib, Clovis Oncology) и олапариб (Olaparib, AstraZeneca), но возможность назначения таргетной терапии по результатам генетического анализа предъявляет особые требования к срокам проведения такого исследования: как правило, результат должен быть получен не позднее 14–20 дней с момента назначения, что требует ускорения и автоматизации как лабораторной (подготовка библиотек), так и био-информационной части (обработка сырых данных, аннотация и интерпретация), так как стандартные сроки выполнения аналогичных анализов на данный момент составляют более 30–40 дней, причем значительное количество времени тратится именно на биоинформационную обработку.
<линическая’2п20 п эакти ка Том 11 №1
Цель исследования — анализ мутаций в генах системы репарации у пациентов с наследственными онкологическими синдромами; создание публичной базы данных с информацией о встречаемости этих мутаций в различных этнических выборках, что позволит расширить знания о генетических основах рака молочной железы, яичников и других с помощью объединения информации о вариантах генов и соответствующих клинических данных со всей территории России, а также поможет в интерпретации мутаций, встречающихся при анализе российских популяций.
МЕТОДЫ
Критерии соответствия
Пациенты включались в исследование при наличии возможности собрать образцы крови для проведения поиска герминальных мутаций, наличии добровольного информированного согласия об участии в исследовании.
Пациенты исключались из исследования при наличии предшествующей или настоящей системной противоопухолевой или лучевой терапии по поводу выявленного рака молочной железы (РМЖ), любом доказательстве наличия тяжелой или неконтролируемой системной патологии, активной инфекции, активного геморрагического диатеза или почечного трансплантата, в том числе пациенты с известным гепатитом В, гепатитом С или вирусом иммунодефицита человека (HIV).
В исследуемую группу включались больные РМЖ или раком яичников (РЯ) с наличием хотя бы одного из клинических критериев (отягощенный семейный анамнез по злокачественным новообразованиям: случаи РМЖ, РЯ, рака поджелудочной железы у кровных родственников), отягощенный собственный анамнез (наличие в прошлом или настоящем РМЖ контрлатеральной молочной железы, РЯ, рака поджелудочной железы), возраст младше 50 лет, тройной негативный рак молочной железы).
У пациентов при первичном визите был собран семейный и собственный анамнез, на основании которого пациент включался в данное исследование. Для молекулярно-генетического тестирования производился забор венозной крови в количестве 4–6 мл.
Этическая экспертиза
Работа выполнена в соответствии с требованиями международного стандарта этических норм и качества научных исследований GCP (Good
Clinical Practice — надлежащая клиническая практика ) и Хельсинкской декларации по защите прав человека.
Условия проведения
Исследование включало 1644 образца биоматериала от пациентов с наследственным раком, проходивших обследование и лечение в Республиканском клиническом онкологическом диспансере Министерства здравоохранения Республики Татарстан (Казань) в 2014–2016 гг.
Методы регистрации исходов
Гены, включенные в панель: ATM , BARD1 , BRCA1 , BRCA2 , CDH1 , CDK4 , CDK12 , CDKN2A , CFTR , CHEK1 , CHEK2 , CTNNA1 , EPCAM , FANCI , FANCJ/BRIP1 , FANCL , MLH1 , MSH2 , MSH6 , MUTYH , PALB2 , PARP1 , PDGFRA , PMS2 , PPP2R2A , PRSS1 , RAD51B , RAD51C , RAD51D , RAD54L , SPINK1 , STK11 , TP53 , XRCC3 .
ДНК из цельной периферической крови выделялась с помощью набора QIAamp DNA Blood Mini Kit (Qiagen, Германия) на автоматической станции QIAcube (Qiagen). Концентрация ДНК измерялась на спектрофотометре NanoVue Plus (GE Healthcare, США) и составляла 30–50 нг/мкл. Подготовка библиотек для секвенирования осуществлялась с помощью NimblGen SepCapEZ Choice (Roche, Швейцария) по протоколу, рекомендованному производителем. Секвенирование проводилось на приборе MiSeq (Illumina, США). Картирование прочтений на референсную последовательность генома человека (hg19) проводилось при помощи алгоритма BWA-MEM, качество исходных данных, выравнивания, обогащения и покрытия целевых регионов проверялось с помощью FastQC, BAMQC и NGSrich. Среднее покрытие составило 274х, доля корректно картированных прочтений — 99,1%, доля целевых регионов с покрытием выше 100x — 92,8%.
Дедупликация, рекалибровка и поиск нуклеотидных вариаций выполнялись с помощью GAT-K4+Strelka2, полученный VCF-файл обрабатывался с помощью программы SnpSift (глубина прочтения более 10) и аннотировался с помощью SnpEff (анализ всех транскриптов), ANNOVAR (анализ частот аллелей в ExAC, 1000G и ESP6500, алгоритмы проверки функциональной значимости SIFT, PolyPhen2, MutationTaster, FATMM, CADD, DANN, M-CAP, REVEL), баз данных dbSNP, ClinVar, HGMD Professional 2019.4, BRCA Exchange (блок-схема представлена на рис. 1).

Рис. 1. Схема универсального пайплайна для версий генома hg19/hg38

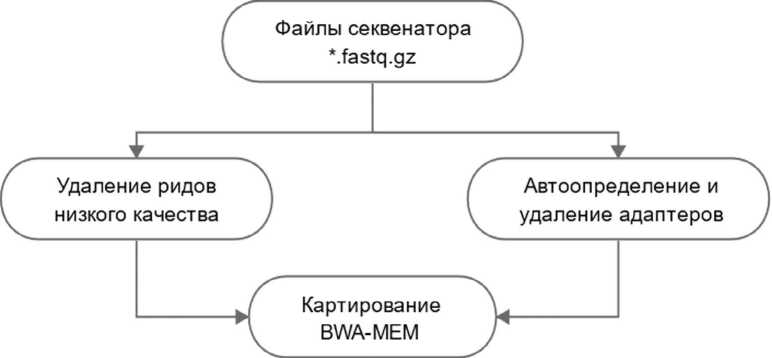
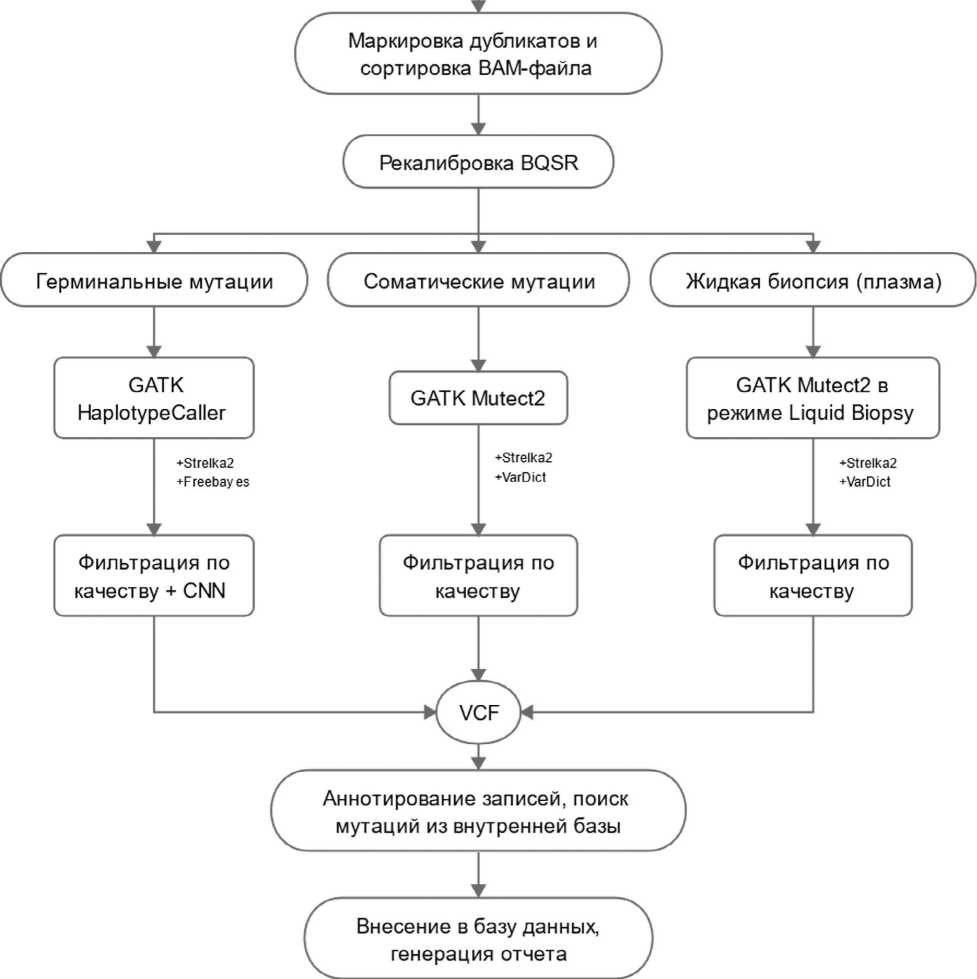
<линическая’2п20 п эакти ка Том 11 №1
Таблица 1
Представленность в изучаемой выборке 8 самых частых мутаций генов BRCA1/2 , используемых в ПЦР-диагностике в РФ
|
Наименование мутации |
Число мутаций в выборке |
|
BRCA1: 5382insC |
87 |
|
BRCA1: 300T>G |
9 |
|
BRCA1: 4153delA |
9 |
|
BRCA1: 2080delA |
6 |
|
BRCA1: 185delAG |
4 |
|
BRCA1: 3819delGTAAA |
4 |
|
BRCA1: 3875delGTCT |
1 |
|
BRCA2: 6174delT |
0 |
Пайплайн1 построен по модульному принципу с использованием общих вычислительных блоков для облегчения внесения изменений в алгоритмы и контроля версий. Вычислительный конвейер автоматически подстраивается под размер входных файлов и распределяет нагрузку по необходимому числу контейнеров, что дает возможность быстро и эффективно обрабатывать данные любого размера — от таргетных панелей до экзомов и геномов. Каждый модуль выполняется в изолированном окружении с динамическим выделением ресурсов, требуемых для обработки загруженного набора файлов, а облачная среда снимает ограничения на количество одновременно анализируемых образцов. Время получения готовых отчетов для 80% пациентов из запуска MiSeq (96 образцов) не более 3 ч, для всех 96 — не более 24 ч.
РЕЗУЛЬТАТЫ
Основные результаты исследования
В результате секвенирования для генов BRCA1/ BRCA2 были обнаружены 29 912 нуклеотидных вариантов (однонуклеотидные замены, короткие вставки/делеции размером до 50 пар нуклеотидов), из них 119 мутаций у 374 пациентов являлись патогенными.
Широко распространенная ПЦР-панель из 8 частых мутаций (табл. 1) не отражает истинного распределения патогенных мутаций BRCA1/ BRCA2 в российских популяциях (например, мутация BRCA2: 6174delT из этой панели в нашей выборке не встретилась ни разу), что подтверждается результатами исследования OVATAR компании «АстраЗенека», и необходима разработка новых диагностических тест-систем для скрининга носительства частых мутаций в генах BRCA1/BRCA2 .
Предлагаемая нами панель из 15 мутаций с учетом частоты их встречаемости в различных этносах приведена в табл. 2.
С учетом полученных результатов мы считаем, что в Российской Федерации необходимо проведение исследования по изучению распространенности
Таблица 2
Распределение частот 15 самых частых мутаций по данным настоящего исследования в различных этносах
|
Мутация |
Славяне (%) |
Татары (%) |
Башкиры (%) |
Чуваши (%) |
Все (%) |
|
c.5266dupC BRCA1:NM_007294.3 p.Q1756fs |
8,5 |
6,6 |
10 |
3,2 |
6,7 |
|
c.181T>G BRCA1:NM_007294.3 p.C61G |
0,6 |
1,4 |
0 |
0 |
0,7 |
|
c.4035delA BRCA1:NM_007294.3 p.E1345fs |
0,8 |
0,4 |
0 |
0,3 |
0,7 |
|
c.5251C>T BRCA1:NM_007294.3 p.R1751X |
1 |
0 |
0 |
0 |
0,5 |
|
c.5161C>T BRCA1:NM_007294.3 p.Q1721X |
0 |
0,8 |
6,9 |
0 |
0,5 |
Таблица 2. Окончание
|
c.1961delA BRCA1:NM_007294.3 p.K654fs |
0,8 |
0 |
0 |
0 |
0,46 |
|
c.3749dupA BRCA2:NM_000059.3 p.E1250fs |
0 |
1 |
0 |
0 |
0,38 |
|
c.4675G>A BRCA1:NM_007294.3 p.E1559K |
0,6 |
0,2 |
0 |
0 |
0,38 |
|
c.8754+1G>A BRCA2:NM_000059.3 |
0,3 |
0 |
0 |
3,2 |
0,3 |
|
c.961_962insAA BRCA2:NM_000059.3 p.Q321fs |
0,1 |
0,8 |
3,4 |
0 |
0,3 |
|
c.3700_3704del BRCA1:NM_007294.3 p.V1234fs |
0,1 |
0,6 |
0 |
0 |
0,3 |
|
c.4689C>G BRCA1:NM_007294.3 p.Y1563X |
0,4 |
0 |
0 |
0 |
0,2 |
|
c.2897_2898del BRCA2:NM_000059.3 p.T966fs |
0 |
0,4 |
0 |
0 |
0,2 |
|
c.3629_3630del BRCA1:NM_007294.3 p.E1210fs |
0 |
0 |
0 |
0 |
0,2 |
|
c.4327C>T BRCA1:NM_007294.3 p.R1443X |
0,3 |
0 |
0 |
0 |
0,2 |
|
c.1301_1304del BRCA2:NM_000059.3 p.K434fs |
0,3 |
0 |
0 |
0 |
0,2 |
|
c.3143delG BRCA1:NM_007294.3 p.G1048fs |
0,2 |
0,2 |
0 |
0 |
0,2 |
|
c.5075-1G>A BRCA1:NM_007294.3 |
0,3 |
0 |
0 |
0 |
0,2 |
|
c.7007+1G>A BRCA2:NM_000059.3 |
0 |
0,4 |
0 |
0 |
0,2 |
|
c.9117G>A BRCA2:NM_000059.3 p.P3039P |
0,2 |
0,2 |
0 |
0 |
0,2 |
|
c.2806_2809del BRCA2:NM_000059.3 p.K936fs |
0,3 |
0 |
0 |
3,2 |
0,2 |
|
c.3756_3759del BRCA1:NM_007294.3 p.L1252fs |
0,2 |
0 |
0 |
0 |
0,1 |
<линическая’2п20 п эакти ка Том 11 №1
герминальных мутаций при наследственных раках в панели генов (в том числе системы репарации), а также для идентификации патогенных вариантов, приводящих к развитию онкозаболеваний, причем это исследование должно включать несколько тысяч пациентов для увеличения статистической мощности и крупную выборку популяционного контроля для оценки популяционной частоты.
Проведенный анализ результатов секвенирования нового поколения (Next Generation Sequencing, NGS) 1644 образцов наследственной онкопатологии выявил 119 патогенных мутаций генов BRCA1 / BRCA2 у 374 пациентов, при этом была обнаружена выраженная разница в частоте встречаемости мутаций между различными этносами — славянами, татарами, башкирами и чувашами.
Частота мутаций в генах BRCA1 , BRCA2 существенно отличается между пациентками с раком яичников и раком молочной железы славянского и татарского происхождения, что подтверждает необходимость NGS-анализа в случае отсутствия положительных результатов анализа ПЦР в реальном времени.
ОБСУЖДЕНИЕ
При анализе данных полногеномного секвенирования в клинической практике возникает две основные проблемы — скорость обработки/получения итогового результата и показатели чувствительно-сти/специфичности проводимого исследования. Вариантов проведения анализа у лаборатории существует несколько:
-
• встроенное ПО прибора (например, MiSeq Reporter);
-
• облачное ПО производителя прибора (например, Illumina BaseSpace);
-
• готовое платное решение (например, SOPHiA GENETICS);
-
• использование собственного пайплайна на локальном сервере;
-
• использование собственного пайплайна в облаке.
Все варианты, кроме последнего, не соответствовали заданным параметрам:
Главным преимуществом облачного подхода к биоинформационному анализу является независимость обработки образцов друг от друга, что позволяет одновременно анализировать десятки и сотни образцов, получая на примере панели генов системы репарации общее время биоинфор-мационного анализа всего запуска прибора в пределах 3 ч с обеспечением максимальной точности результата. Используемый пайплайн обладает следующими характеристиками при анализе референсного образца GiaB/NIST NA12878/HG001 на экзомной панели Illumina Nextera Expanded Exome с учетом высокодостоверных регионов:
-
• Precision 0,99004;
-
• Recall 0,99993;
-
• F-measure 0,99496.
Автоматизация поиска ранее известных патогенных мутаций дает возможность формировать готовый отчет об исследовании для 70–80% образцов в запуске сразу по завершении обработки, при этом в нашем проекте после роста размера базы данных свыше 1000 пациентов около 50–60% новых образцов имели ранее обнаруженные и интерпретированные патогенные мутации, 10–15% не имели мутаций вообще и лишь 20% требовали дальнейшей интерпретации возможно патогенных вариантов. Мы ожидаем, что при росте количества образцов в базе свыше 10 000 более 90% образцов в запуске не потребует затрат времени на интерпретацию.
ЗАКЛЮЧЕНИЕ
С помощью метода NGS появилась возможность в короткие сроки выявлять редкие мутации, характерные для различных этносов, что дает возможность оптимизировать диагностическую и лечебную тактику пациентов из данной популяции.
В результате проведенной работы была создана публичная база данных частоты мутаций и полиморфизмов генов системы репарации в различных этносах .
ИСТОЧНИК ФИНАНСИРОВАНИЯ
Работа поддержана компанией «АстраЗенека» (грант ESR-17-12934).
Список литературы Опыт создания публичной базы данных мутаций oncoBRCA: биоинформационные проблемы и решения
- Torre LA, Bray F, Siegel RL, et al. Global cancer statistics, 2012. CA Cancer J Clin. 2015;65(2):87-108. DOI: 10.3322/caac.21262
- Sokolenko AP, Iyevleva AG, Mitiushkina NV, et al. Hereditary Breast-Ovarian Cancer Syndrome in Russia. Acta Naturae. 2010;2(4):31-35.
- Foretova L, Machackova E, Navratilova M, et al. BRCA1 and BRCA2 mutations in women with familial or early-onset breast/ovarian cancer in the Czech Republic. Hum Mutat. 2004;23(4):397-398. DOI: 10.1002/humu.9226
- Хасанова А.И., Гордиев М.Г., Ратнер Е.Ю., и др. BRCA-ассоциированный рак молочной железы у представительниц татарской национальности на примере клинического случая // Приволжский онкологический вестник. - 2016. - №2. - С. 104-108.
- Fackenthal JD, Olopade OI. Breast cancer risk associated with BRCA1 and BRCA2 in diverse populations. Nat Rev Cancer. 2007;7(12):937-948. DOI: 10.1038/nrc2054
- Matsuda S. Defective DNA repair systems and the development of breast and prostate cancer (Review). Int J Oncol. 2013;42(1):29-34. DOI: 10.3892/ijo.2012.1696
- Zook JM, McDaniel J, Olson ND, et al. An open resource for accurately benchmarking small variant and reference calls. Nat Biotechnol. 2019;37(5):561-566. DOI: 10.1038/s41587-019-0074-6
- Miller NA, Farrow EG, Gibson M, et al. A 26-hour system of highly sensitive whole genome sequencing for emergency management of genetic diseases. Genome Med. 2015;7:100. DOI: 10.1186/s13073-015-0221-8
- Chen J, Li X, Zhong H, et al. Systematic comparison of germline variant calling pipelines cross multiple next-generation sequencers. Sci Rep. 2019;9(1):1-13. DOI: 10.1038/s41598-019-45835-3
- Van der Auwera GA, Carneiro MO, Hartl C, et al. From FastQ data to high confidence variant calls: the Genome Analysis Toolkit best practices pipeline. Curr Protoc Bioinformatics. 2013;43:11.10.1-11.10.33. DOI: 10.1002/0471250953.bi1110s43


