Полногеномные ассоциативные исследования качества мяса по показателям цвета грудки у кур (Gallus gallus L.)
Автор: Ветох А.Н., Джагаев А.Ю., Белоус А.А., Волкова Н.А., Зиновьева Н.А.
Журнал: Сельскохозяйственная биология @agrobiology
Рубрика: Генетика и геномика
Статья в выпуске: 6 т.58, 2023 года.
Бесплатный доступ
Одним из наиболее важных признаков качества мяса служит его цветовая характеристика, которая во многом определяет потребительский спрос. Качество мяса оценивают по его цветовому спектру по специальным шкалам. В частности, широко применяется шкала L*a*b*, эффективность которой показана во многих отраслях мясного животноводства. В ряде исследований установлена генетическая обусловленность цветовых характеристик мяса сельскохозяйственных животных и птицы. Выявлены однонуклеотидные полиморфизмы (SNPs) и гены-кандидаты, детерминирующие степень проявления этого признака (J. Sun с соавт., 2022; X. Guo с соавт., 2023). Мы выполнили полногеномные ассоциативные исследования цветовых показателей мяса грудки у кур из F2 ресурсной популяции на основании данных полногеномного генотипирования. Цель работы - поиск SNPs и идентификация генов, ассоциированных с показателем цвета мяса у кур. Объектом изучения были куры F2 модельной ресурсной популяции ( n = 260, виварий ФИЦ животноводства - ВИЖ им. академика Л.К. Эрнста, 2021-2023 годы). Для получения популяции использовали две контрастные по мясным качествам породы кур - русскую белую (медленный рост) и корниш (быстрый рост). Генотипирование полученной птицы проводили с использованием чипов высокой плотности Illumina Chicken iSelect BeadChip 60k («Illumina Inc.», США). В возрасте 9 нед после экспериментального убоя птицы измерили характеристики мяса грудки по цветовой шкале L*a*b* с использованием портативного спектрофотометра CM-700d («Konica Minolta», Япония). На основании полученных данных гено- и фенотипирования выполнили полногеномные ассоциативные исследования, использовав программное обеспечение PLINK 1.9 с принятыми ограничениями (geno 0,1; mind 0,1; maf 0,03). В качестве порогового критерия достоверности установили p 2 ресурсная популяция кур характеризовалась высоким коэффициентом изменчивости по показателям зеленого (a*) и голубого (b*) участков спектров цвета мяса (от 19,99 % до 97,23 %). По показателю L вариабельность в исследованной популяции была относительно низкой (коэффициент изменчивости не превышал 9,75 %). На основании проведенного GWAS-анализа выявлено 60 значимых SNPs, в том числе ассоциированных с цветовым спектром: по L* - 28 SNPs, по a* - 48 SNPs, по b* - 4 SNPs. Указанные SNPs локализованы на хромосомах GGA1 (10 SNPs), GGA2 (3 SNPs), GGA3 (18 SNPs), GGA7 (2 SNPs), GGA8 (4 SNPs), GGA10 (2 SNPs), GGA12 (7 SNPs), GGA13 (9 SNPs), GGA17 (4 SNPs) и GGA18 (1 SNP). Идентифицированы 270 генов-кандидатов, связанных с изученными признаками, в 30 из этих генов были локализованы выявленные SNPs. Результаты исследования могут быть использованы в геномной селекции на улучшение качественных характеристик мяса кур.
Gallus gallus, куры, snp, gwas, гены-кандидаты, качество мяса, цвет мяса, шкала l*a*b*
Короткий адрес: https://sciup.org/142240678
IDR: 142240678 | УДК: 636.5:637.04:577.2 | DOI: 10.15389/agrobiology.2023.6.1068rus
Текст научной статьи Полногеномные ассоциативные исследования качества мяса по показателям цвета грудки у кур (Gallus gallus L.)
Развитие мясного птицеводства связано с высоким спросом на продукцию отрасли. С каждым годом возрастают требования к качеству мяса птицы и его товарному выходу (1, 2). По прогнозам ФАО ожидается, что до 2050 года общее производство и потребление этого продукта будет увеличиваться ежегодно на 1,8 %, что значительно превышает ожидаемый рост производства и потребления свинины (0,8 % в год) (3). Мясо кур служит источником белка с высокой биологической ценностью, особенно в сравнении с растительными белками (в частности, по содержанию железа, фосфора, витамина А, тиамина, никотиновой кислоты) (4). Кроме того, низкая энергетическая ценность делает куриное мясо продуктом здорового питания с пониженным содержанием жира и повышенной долей полиненасы-щенных жирных кислот (ПНЖК) по сравнению с мясом других видов (5).
При современном производстве мяса птицы делается упор на повышение качественных показателей с учетом различных характеристик конечного продукта, включая внешний вид, текстуру и упругость, влагоудержива-
∗ Работа выполнена при финансовой поддержке Министерства науки и высшего образования РФ, тема ¹ FGGN-2023-0002.
ющую способность, цвет, pH, продолжительность хранения, содержание коллагена, растворимость белка, способность связывать жиры (6). На многие из этих факторов существенное влияние оказывают условия кормления и содержания птицы (7), а также пол, возраст и порода (7, 8).
Цвет — важный показатель качества мяса, в значительной мере определяющий потребительский спрос (9). Производству продукции с желательным цветом и отсутствием дефектов внешнего вида, определяющими эффективность ее реализации и конечную цену, уделяется большое внимание (9, 10). К дефектам цвета мяса относится так называемое бледное, мягкое и экссудативное состояние (PSE). PSE становится растущей проблемой в мясной промышленности. При PSE состоянии снижается водоудерживающая способность (ВУС) мяса, а его текстура становится более мягкой (11). У бройлеров на формирование мяса PSE влияют различные предубойные факторы, методы оглушения и режимы охлаждения (3, 12).
Цель работы — поиск SNPs и идентификация генов, ассоциированных с показателем цвета мяса у кур.
Методика. Исследования проводили на курах F 2 модельной ресурсной популяции ( n = 260; виварий ФИЦ животноводства — ВИЖ им. академика Л.К. Эрнста, 2021-2023 годы), для создания которой использованы две породы — корниш (мясная порода) и русская белая (яичная порода). Цыплят до 3-недельного возраста выращивали в брудерах с постепенным понижением температуры с 34 ° С (в первые часы после выхода из яйца) до 23 ° С, затем переводили на напольное содержание. Условия содержания птицы в соответствии с ее возрастом предусматривали постоянный доступ к полнорационному промышленному комбикорму и свежей воде, хорошую приточную вентиляцию, обеспечивающую отсутствие сырости, сквозняков и загазованности, нормальную освещенность. В возрасте 9 нед проводили экспериментальный убой птицы в соответствии с национальным стандартом Российской Федерации ГОСТ Р 52837-2007 «Птица сельскохозяйственная для убоя. Технические условия». Голодная выдержка составляла 8-10 ч. После убоя тушки ошпаривали, снимали оперение и проводили их обвалку.
ДНК выделяли из пульпы пера с использованием коммерческого набора «ДНК Экстран-2» (ООО «НПФ Синтол», Россия) в соответствии с рекомендациями производителя. Качество и целостность выделенной ДНК проверяли в 1 % агарозном геле методом горизонтального гель-электрофо-реза. Чистоту препарата оценивали на спектрофотометре NanoPhotometer® N60 («Thermo Fisher Scientific», США), отбирали пробы с соотношением OD 260/280 больше 1,8. Концентрацию ДНК определяли на флуориметре Qubit® 2.0 («Invitrogen/Life technologies», США) c использованием набора Qubit™ dsDNA BR Assay («Invitrogen/Life technologies», США) для количественного определения ДНК в диапазоне 2-1000 нг.
Полногеномное генотипирование кур выполняли с использованием ДНК-чипа Illumina Chicken iSelect BeadChip («Illumina, Inc.», США), содержащего 60 тыс. SNP. Контроль качества и фильтрацию данных генотипирования для каждого образца и каждого SNP выполняли в программной среде R c программным пакетом PLINK 1.9 , применяя следующие фильтры в программе: --mind 0.10, --geno 0.10, --maf 0.05, --hwe 1e-3. Для полногеномного ассоциативного исследования (GWAS) и выявления SNP, связанных с показателями цвета мышечных тканей, использовали команды для регрессионного анализа –assoc, --adjust, --qt-means. Для подтверждения достоверного влияния SNP и определения значимых регионов в геноме кур использовали тест для проверки нулевых гипотез по Бон-феррони. Данные визуализировали в пакете qqman . Поиск генов-кандидатов в области идентифицированных SNP проводили с помощью средства просмотра геномных данных (Genome Data Viewer) в базе данных NCBI Gallus gallus (chicken) . Функциональные аннотации генов выполняли с привлечением базы данных GeneCards и программы DAVID .
Статистические показатели рассчитывали с использованием пакета анализа данных в Microsoft Office 365 по следующим параметрам: значение средних ( М ), стандартная ошибка (±SEM), минимум (min), максимум (max), коэффициент вариации ( Cv , %).
|
Показатель 1 |
M |
] ±SEM |
min 1 |
max |
] Cv , % |
|
L |
38,88 |
1,98 |
42,08 |
62,28 |
7,62 |
|
a |
2,12 |
0,19 |
-0,31 |
14,07 |
97,23 |
|
b |
10,07 |
0,18 |
5,73 |
16,81 |
19,99 |
Примечание. Координата L характеризует светлоту по спектру от темного (0) к светлому (100); a — спектр от зеленого ( - 128) до пурпурного (127); b — спектр от голубого ( - 128) до желтого (127).
SNP dencity plot 0 22 44 66 88 IIO' 132 154 176 198
Chr 1 ■■■■■■■ Chr21| 1ИШ II ■ сьгз I Hill IMIMII Ml Chr4 И 11IIИ МНИМ
Chr5 ИIIIMIIMIIIMIIII
Chr6 ПП 11 !■ chrvMIIIMMIII
ChrSниа inn
CM !■ I I
Chit) II I I
СЫ1И1 I II
СМ21 II
СМ3 Hill
CM4l I .11
С11Г15 11 I
ChM I
CM 7 I
CM S 11 II
I)
Рис. 1. График плотности SNPs по изучаемым цветовым характеристикам мяса кур ( Gallus gallus L.) из F 2 ресурсной популяции по хромосомам ( n = 260, виварий ФИЦ животноводства — ВИЖ им. академика Л.К. Эрнста, 2021-2023 годы).
После фильтрации данных для полногеномных исследований ассоциаций всего использовали 16630 SNPs. Плотность распределения выявленных SNPs по хромосомам представлена на рисунке 1.
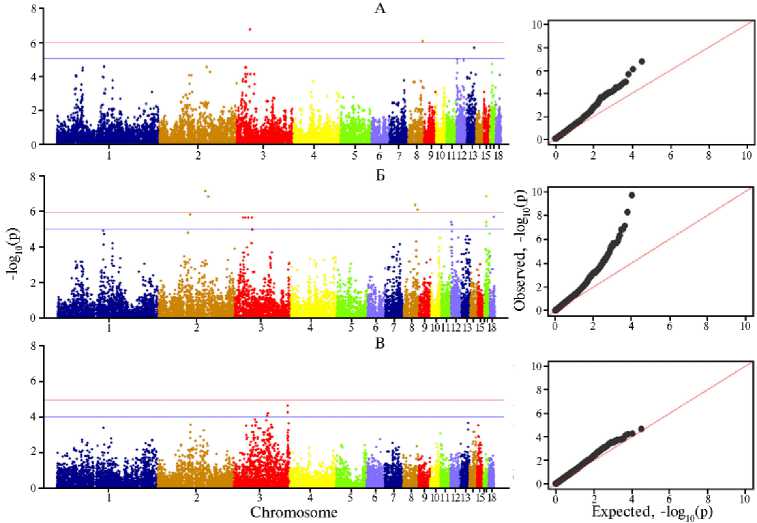
По всем изученным цветовым показателям мяса у кур из F 2 ресурсной популяции мы в результате идентифицировали 60 значимых SNPs (p < 0,00001) на хромосомах GGA1, GGA2, GGA3, GGA7, GGA8, GGA10,
GGA12, GGA13, GGA17, GGA18 (рис. 2).
гена.
Продолжение таблицы 4
|
EDN1 |
62406640..62411539 |
Клеточный гомеостаз ионов кальция, сокращение гладких мышц вен |
|
GPX3 |
12661488..12663434 |
Реакция на окислительный стресс |
|
INSIG2 |
28449946..28462775 |
Биосинтез холестерина |
|
LCTL |
18547541..18551236 |
Метаболизм углеводов |
|
LEFTY1 |
16864275..16879586 |
Развитие селезенки |
|
LACAAT2L |
12628444..12633004 |
Транспорт аминокислот |
|
MAP2K1 |
18496305..18527713 |
Развитие сердца, развитие щитовидной железы |
|
NRSN1 |
57666592..57673732 |
Развитие нервной системы |
|
PGM1 |
27548636..27567569 |
Метаболизм углеводов, процесс обмена глюкозы |
|
PPP1CB |
26901055..26932174 |
Метаболизм гликогена |
|
SCCPDH |
33530313..33540359 |
Биосинтез гликолипидов |
|
SFXN1 |
9634295..9669465 |
Транспорт ионов, транспорт аминокислот |
|
STX19 |
89905548..89921452 |
Внутриклеточный транспорт белков |
|
TNNC1 |
643058..648527 |
Сокращение скелетных мышц, регуляция мышечного сокращения |
Цвет служит важным показателем качества мяса курицы и тесно связан с гликолизом и внутримышечным жировым обменом. Отмечены функции ряда выявленных генов-кандидатов (транспорт липидов, аминокислот, белков, холестерина, биосинтез жирных кислот, развитие селезенки, печени, сердца, связывание белков), которые напрямую или косвенно влияют на цветообразование мяса. Выявленные гены отвечают за биологический функционал, включая развитие многоклеточного организма и его органов, гомеостаз жирных кислот, биохимические процессы. Как известно, стресс сильно влияет на органолептические показатели мяса, поэтому развитие нервной системы играет немаловажную роль в формировании его цветовых спектров. Для 7 генов-кандидатов, идентифицированных в наших исследованиях, в ряде научных работ показана связь с селекционно значимыми признаками у кур. В частности, установлены высокодостоверные ассоциации гена AKT3 с пигментацией перьев у кур (24) и развитием мышечных волокон (25). Показано влияние генов FSTL4 и VDAC1 на содержание влаги в желтке яйца (26), гена TTC7A — на накопление гликогена в мышцах кур (27). Также сообщается о связи гена FSTL4 с быстрым ростом мышц у бройлеров (28). Установлено влияние генов MAGI1 и VDAC1 на показатели иммунитета (29-31), генов FZD3 и EPHA6 — на состояние и развитие нервной системы (32). Для других выявленных нами генов-кандидатов в ряде работ обнаружена связь с показателями мясной продуктивности и качеством мяса у других видов сельскохозяйственных животных. Так, установлены ассоциации генов TTC7A и AFG1L с толщиной шпика у свиней (33, 34), гена BRINP1 — с показателями роста у свиней (35) и развития (линейные промеры) у коз (36).
Таким образом, в проведенном исследовании мы выявили 60 значимых SNPs, ассоциированных с цветом мяса у кур, в том числе 28 SNPs, связанных с цветовым спектром L*, 48 SNPs — со спектром a*, 4 SNPs — со спектром b*. Обнаруженные нами SNPs локализованы на хромосомах GGA1 (10 SNPs), GGA2 (3 SNPs), GGA3 (18 SNPs), GGA7 (2 SNPs), GGA8 (4 SNPs), GGA10 (2 SNPs), GGA12 (7 SNPs), GGA13 (9 SNPs), GGA17 (4 SNPs), GGA18 (1 SNP). Также идентифицированы 270 генов-кандидатов, связанных с изученными признаками, из которых в 30 генах локализуются выявленные SNPs. Эти результаты могут быть использованы в геномной селекции кур на качественные характеристики мяса.
Список литературы Полногеномные ассоциативные исследования качества мяса по показателям цвета грудки у кур (Gallus gallus L.)
- Sandercock D.A., Barker Z.E., Mitchell M.A., Hocking P.M. Changes in muscle cell cation reg-ulation and meat quality traits are associated with genetic selection for high body weight and meat yield in broiler chickens. Genetics Selection Evolution, 2009, 41: 8 (doi: 10.1186/1297-9686-41-8).
- Фисинин В.И., Буяров В.С., Буяров А.В., Шуметов В.Г. Мясное птицеводство в регионах России: современное состояние и перспективы инновационного развития. Аграрная наука, 2018, 2: 30-38.
- Alnahhas N., Berr C., Chabault M., Chartrin P., Le Bihan-Duval E. Genetic parameters of white striping in relation to body weight, carcass composition, and meat quality traits in two broiler lines divergently selected for the ultimate pH of the pectoralis major muscle. BMC Genomics, 2016, 17: 61 (doi: 10.1186/s12863-016-0369-2).
- Karunanayaka D.S., Jayasena D.D., Jo C. Prevalence of pale, soft, and exudative (PSE) condition in chicken meat used for commercial meat processing and its effect on roasted chicken breast. Journal of Animal Science and Technology, 2016, 58: 27 (doi: 10.1186/s40781-016-0110-8).
- Riovanto R., Marchi M., Cassandro M., Penasa M. Use of near infrared transmittance spectros-copy to predict fatty acid composition of chicken meat. Food Chemistry, 2012, 134(4): 2459-2464 (doi: 10.1016/j.foodchem.2012.04.038).
- Семикопенко Н.И. Повышение эффективности производства мяса птицы с помощью процесса массирования. Все о мясе, 2017, 5: 38-41.
- Дорохин Н.А. Качественные характеристики мяса цыплят-бройлеров и факторы, влияющие на них: обзор. Сельскохозяйственный журнал, 2020, 5(13): 59-64.
- Боголюбова Н.В., Некрасов Р.В., Зеленченкова А.А. Антиоксидантный статус и качество мяса у сельскохозяйственной птицы и животных при стрессе и его коррекция с помощью адаптогенов различной природы (обзор). Сельскохозяйственная биология, 2022, 57(4): 628-663 (doi: 10.15389/agrobiology.2022.4.628rus).
- Qiao M., Fletcher D.L., Smith D.P., Northcutt J.K. The effect of broiler meat color on pH, moisture, water-holding capacity, and emulsification capacity. Poultry Science, 2001, 80: 676-680 (doi: 10.1093/ps/80.5.676).
- Petracci M., Betti M., Bianchi M., Cavani C. Color variation and characterization of broiler breast meat during processing in Italy. Poultry Science, 2004, 83: 2086-2092 (doi: 10.1093/ps/83.12.2086).
- Woelfel R.L., Owens C.M., Hirschler E.M., Martinez R.D., Sams A.R. The characterization and incidence of pale, soft, and exudative broiler meat in a commercial processing plant. Poultry Sci-ence, 2002, 81: 579-584 (doi: 10.1093/ps/81.4.579).
- Oba A., Almeida M.D., Pinheiro J.W., Ida E.I., Marchi D.F., Soares A.L., Shimokomaki M. The effect of management of transport and lairage conditions on broiler chicken breast meat quality and DOA (Death on Arrival). Brazilian Archives of Biology and Technology, 2009, 52: 205-211 (doi: 10.1590/S1516-89132009000700026).
- Mir N.A., Rafiq A., Kumar F., Singh V., Shukla V. Determinants of broiler chicken meat quality and factors affecting them: a review. Journal of Food Science and Technology, 2017, 54: 2997-3009 (doi: 10.1007/s13197-017-2789-z).
- Anadon H.L.S. Biological, nutritional, and processing factors affecting breast meat quality of broilers. Ph.D. Thesis. Virginia Polytechnic Institute and State University, Blacksburg, VA, 24061, USA, 2002.
- Ежкова Г.О., Пономарев В.Я., Решетник О.А. Технология повышения качества мясного сырья PSE и DFD на организменном и тканевом уровне: монография. Казань, 2007.
- Bowker B. Chapter 4. Developments in our understanding of water-holding capacity. In: Woodhead publishing series in food science, technology and nutrition, poultry quality evaluation. Woodhead Pub-lishing, 2017: 77-113 (doi: 10.1016/B978-0-08-100763-1.00004-0).
- Allais S., Hennequet-Antier C., Berri C., Salles L., Demeure O., Le Bihan-Duval E. Mapping of QTL for chicken body weight, carcass composition, and meat quality traits in a slow-growing line. Poultry Science, 2019, 98(5): 1960-1967 (doi: 10.3382/ps/pey549).
- Seo D.W., Park H.B., Jung S., Cahyadi M., Choi N.R., Jin S., Heo K.N., Jo C., Lee J.H. QTL analyses of general compound, color and pH traits in breast and thigh muscles in Korean native chicken. Livestock Science, 2015, 182: 145-150 (doi: 10.1016/j.livsci.2015.09.020).
- Sun Y., Zhao G., Liu R., Zheng M., Hu Y., Wu D., Zhang L., Li P., Wen J. The identification of 14 new genes for meat quality traits in chicken using a genome-wide association study. BMC Genomics, 2013, 14: 458 (doi: 10.1186/1471-2164-14-458).
- Sun J., Tan X., Yang X., Bai L., Kong F., Zhao G., Wen J., Liu R. Identification of candidate genes for meat color of chicken by combing selection signature analyses and differentially expressed genes. Genes, 2022, 13: 307 (doi: 10.3390/genes13020307).
- Коршунова Л.Г., Карапетян Р.В., Комарчев А.С., Куликов Е.И. Ассоциации однонуклео-тидных замен в генах-кандидатах с хозяйственно полезными признаками у кур (Gallus gallus domesticus L.) (обзор). Сельскохозяйственная биология, 2023, 58(2): 205-222 (doi: 10.15389/agrobiology.2023.2.205rus).
- Guo X., Zhang H., Wang H., He X.X., Wang J.X., Wei W., Liu M., Xu J.M., Liu Y.N., Jiang R.S. Identification of key modules and hub genes involved in regulating the color of chicken breast meat using WGCNA. Animals, 2023, 13(14): 2356 (doi: 10.3390/ani13142356).
- Горбунова Е.В., Коротаев В.В., Ластовская Е.А. Компьютерное моделирование цвета мяса. Известия высших учебных заведений. Приборостроение, 2014, 57(12): 64-65.
- Park M.N., Choi J.A., Lee K.T., Lee H.J., Choi B.H., Kim H., Kim T.H., Cho S., Lee T. Genome-wide association study of chicken plumage pigmentation. Asian-Australas. J. Anim. Sci., 2013, 26(11): 1523-1528 (doi: 10.5713/ajas.2013.13413).
- Chen S., An J., Lian L., Qu L., Zheng J., Xu G., Yang N. Polymorphisms in AKT3, FIGF, PRKAG3, and TGF-β genes are associated with myofiber characteristics in chickens. Poultry Sci-ence, 2013, 92(2): 325-330 (doi: 10.3382/ps.2012-02766).
- Zhang R., Yao F., Cheng X., Yang M., Ning Z. Identification of candidate genomic regions for egg yolk moisture content based on a genome-wide association study. BMC Genomics, 2023, 24: 110 (doi: 10.1186/s12864-023-09221-8).
- Liu X., Liu L., Wang J., Cui H., Chu H., Bi H., Zhao G., Wen J. Genome-wide association study of muscle glycogen in jingxing yellow chicken. Genes, 2020, 11(5): 497 (doi: 10.3390/genes11050497).
- Dou D., Shen L., Zhou J., Cao Z., Luan P., Li Y., Xiao F., Guo H., Li H., Zhang H. Genome-wide association studies for growth traits in broilers. BMC Genomic Data, 2022, 23: 1 (doi: 10.1186/s12863-021-01017-7).
- Sun Y., Li Q., Hu Y., Sun Y, Liu R., Zheng M., Wen J., Li P., Liu L., Zhao G. Genome-wide association study of immune traits in chicken F2 resource population. J. Anim. Breed. Genet., 2016, 133(3): 197-206 (doi: 10.1111/jbg.12186).
- Ridgway L.D., Kim E.Y., Dryer S.E. MAGI-1 interacts with Slo1 channel proteins and suppresses Slo1 expression on the cell surface. American Journal of Physiology-Cell Physiology, 2009, 297(1): 55-65 (doi: 10.1152/ajpcell.00073.2009).
- Han C., Zeng X., Yao S., Gao L., Zhang L., Qi X., Duan Y., Yang B., Gao Y., Liu C., Zhang Y., Wang Y., Wang X. Voltage-dependent anion channel 1 interacts with ribonucleoprotein complexes to enhance infectious bursal disease virus polymerase activity. Journal of Virology, 2017, 91(16): e00584-17 (doi: 10.1128/JVI.00584-17).
- Nishida K., Flanagan J.G., Nakamoto M. Domain-specific olivocerebellar projection regulated by the EphA-ephrin-A interaction. Development, 2002, 129(24): 5647-5658 (doi: 10.1242/dev.00162).
- Xue Y., Liu S., Li W., Mao R., Zhuo Y., Xing W., Liu J., Wang C., Zhou L., Lei M. Genome-wide association study reveals additive and non-additive effects on growth traits in Duroc pigs. Genes, 2022, 13: 1454 (doi: 10.3390/genes13081454).
- Lee Y.-S., Shin D. Genome-wide association studies associated with backfat thickness in landrace and Yorkshire pigs. Genomics Informatics, 2018, 16(3): 59-64 (doi: 10.5808/GI.2018.16.3.59).
- Ruan D., Zhuang Z., Ding R., Qiu Y., Zhou S., Wu J., Xu C., Hong L., Huang S., Zheng E., Cai G., Wu Z., Yang J. Weighted single-step GWAS identified candidate genes associated with growth traits in a Duroc pig population. Genes, 2021, 12: 117 (doi: 10.3390/genes12010117). 36. Gu B., Sun R., Fang X., Zhang J., Zhao Z., Huang D., Zhao Y., Zhao Y. Genome-wide associ-ation study of body conformation traits by whole genome sequencing in Dazu black goats. Animals, 2022, 12: 548 (doi: 10.3390/ani12050548).


