Популяционно-генетическая характеристика домашнего северного оленя в Республике Якутия на основании полногеномного SNP анализа
Автор: Харзинова В.Р., Доцев А.В., Соловьева А.Д., Федоров В.И., Охлопков И.М., Виммерс К., Рейер Х., Брем Г., Зиновьева Н.А.
Журнал: Сельскохозяйственная биология @agrobiology
Рубрика: Структура генома и генетическое разнообразие
Статья в выпуске: 4 т.52, 2017 года.
Бесплатный доступ
Республика Саха (Якутия) - один из основных оленеводческих регионов Российской Федерации. Из четырех утвержденных пород северного оленя ( Rangifer tarandus ) в республике разводят три - эвенскую, эвенкийскую и чукотскую (харгин). Многолетнее снижение поголовья домашних северных оленей в настоящее время приостановлено, но для сохранения популяции и нивелирования неблагоприятных последствий сокращения численности необходимы современные подходы для оценки генетического разнообразия. Наиболее востребован для этих целей анализ однонуклеотидных полиморфизмов (single nucleotide polymorphism, SNP) с помощью ДНК-микроматриц (ДНК-чипов). В настоящей работе с использованием Bovine SNP50 BeadChip проведено генотипирование и дана популяционно-генетическая характеристика трех пород домашнего северного оленя, разводимых на территории Республики Саха (Якутия). Биологическим материалом для исследований служили образцы ткани уха животных эвенской (EVN, n = 8), эвенкийской (EVK, n = 11) и чукотской (харгин) пород (CHU, n = 7). Программное обеспечение PLINK 1.07 применяли для проведения контроля качества генотипирования. Для статистической обработки данных использовали программное обеспечение PLINK 1.07, Admixture 1.3, R пакеты diveRsity и VennDiagram с последующей визуализацией в R пакетах pophelper и ggplot2. По результатам контроля качества для дальнейшего анализа было отобрано 512 полиморфных SNP. Анализ диаграммы Венна показал, что олени эвенской и эвенкийской пород имели максимальное число уникальных полиморфизмов (14 SNP). У оленей чукотской породы детектировали 11 таких SNP. При расчете основных внутрипопуляционных параметров оказалось, что представители чукотской породы характеризуются более высоким генетическим разнообразием (Ho = 0,180±0,011, He = 0,156±0,008, Ar = 1,488±0,022), а также более высоким избытком гетерозигот (FIS = -0,124) по сравнению с эвенкийской (Ho = 0,161±0,009, He = 0,153±0,008, Ar = 1,487±0,020, FIS = -0,047) и эвенской (Ho = 0,164±0,010, He = 0,149±0,008, Ar = 1,471±0,021, FIS = -0,089) породами. Результаты многомерного шкалирования (MDS) и расчета попарных генетических дистанций (FST) показали, что наибольшей генетической близостью характеризуются олени эвенской и эвенкийской пород. Адмикс-анализ выявил высокую степень генетической обособленности каждой из исследуемых пород. Вместе с тем среди CHU и EVK были обнаружены особи, имеющие смешанное генетическое происхождение, близкое к EVN. Таким образом, мы успешно использовали ДНК-чип, разработанный для крупного рогатого скота, при генетической дифференциации пород северного оленя. Полученные данные найдут применение в разработке программ сохранения и рационального использования этого важнейшего для человека вида животных.
Одиночный нуклеотидный полиморфизм, генетическое разнообразие, породы северного оленя
Короткий адрес: https://sciup.org/142214061
IDR: 142214061 | УДК: 636.294:575.174.015.3 | DOI: 10.15389/agrobiology.2017.4.669rus
Текст научной статьи Популяционно-генетическая характеристика домашнего северного оленя в Республике Якутия на основании полногеномного SNP анализа
Оленеводство представляет собой важнейшую составляющую экономики Севера, Сибири и Дальнего Востока России (1). В Дальневосточном федеральном округе Республика Саха (Якутия) — один из самых крупных оленеводческих регионов. Домашнее оленеводство — исконное занятие для жителей севера Якутии, которое обеспечивает сохранение жизнедеятельности представителей этноса, народных традиций и культуры. Главное конкурентное преимущество республики заключается в том, что вся ее территория относится к районам Крайнего Севера с высоким природно-ресурсным экономическим потенциалом. Якутия входит в таежно-тундровую зоогеографическую зону, пригодную для оленеводства и подразделяемую на приморскую, субарктическую и горную тундры с необыкновенно богатой фауной (2). Домашним оленеводством занимаются в 20 из 36 улусов и в од-
Исследования выполнены при поддержке Российского научного фонда, проект ¹ 14-36-00039. При проведении исследований использовано оборудование ЦКП «Биоресурсы и биоинженерия сельскохозяйственных животных» ВИЖ им. академика Л.К. Эрнста.
ном городском округе Республики Саха. Из 3103,2 тыс. км2 территории Якутии оленеводческие хозяйства владеют 2456,5 тыс. км2, или 79,2 % (3). До 1990-х годов по численности поголовья домашних северных оленей в Российской Федерации Якутия стабильно занимала третье место (361 тыс. гол.) после Чукотского (491 тыс. гол.) и Ямало-Ненецкого (490,5 тыс. гол.) автономных округов (3). С начала 1990-х годов распад крупных оленеводческих хозяйств на мелкие, сокращение товарного оленеводства и спад производственно-экономических показателей отрасли привели к снижению численности оленей (1). Однако благодаря реализации ряда программ Правительства Российской Федерации (4), Президентской программы (5) и государственных целевых программ (6, 7) многолетнее снижение поголовья домашних северных оленей было приостановлено, и по итогам 2016 года их количество в республике составило более 156 тыс. гол. (3).
Из четырех официально признанных пород северных оленей на территории Якутии разводят три — эвенскую (в 12 улусах горно-таежной, тундровой и лесотундровой зон), эвенкийскую (в 9 улусах таежной и тундровой зон) и чукотскую (харгин) (на территории Нижнеколымского улуса). Популяция оленей этих пород по состоянию на 1 января 2015 года насчитывала соответственно 89913, 41774 и 20750 особей, что составляло 59,0; 27,4 и 13,6 % от общего поголовья оленей в республике (8, 9). Эвенская порода представлена оленями c мясо-транспортным типом телосложения, длинным и относительно узким туловищем. Живая масса взрослых самцов составляет 135-145, самок — 91-110 кг (9). Эвенкийская порода — транспортно-мясного типа телосложения. Олени характеризуются наиболее крупными размерами, большей живой массой и хорошей грузоподъ е мностью, имеют длинные конечности и вытянутое туловище, что обеспечивает лучшие рабочие качества. Средняя масса самцов перед гоном — 140170 кг, важенок в возрасте 5,5 лет на осеннем корале — 108-120 кг (9). Чукотскую породу оленей образуют животные мясного типа телосложения, не особо приспособленные для транспортных целей (10). Харгинам свойственна скороспелость, высокая эффективность нагула и нажировки. Живая масса взрослых самцов составляет 130-140 кг, самок — 93-96 кг (9).
Генетическое разнообразие северного оленя достаточно широко изучено с использованием разных типов генетических маркеров: белковых полиморфизмов (11, 12), митохондриальной ДНК (13, 14) и ядерных микросателлитов (15-18). До недавнего времени именно микросателлиты наиболее часто использовались для изучения дрейфа генов, эволюционных процессов, степени дифференциации популяций и оценки генетического разнообразия (19). Однако за последнее десятилетие разработка и совершенствование высокопроизводительных методов генотипирования однонуклеотидных полиморфизмов (single nucleotide polymorphism, SNP) привели к революции в их использовании в качестве молекулярных маркеров, позволяющих давать точную генетическую характеристику популяций (20). Огромное количество SNP в геноме (по разным оценкам, от 3 до 10 млн) позволяет отобрать порядка 100 тыс. маркеров при среднем расстоянии между ними 30 т.п.н. (21). При этом на каждый известный или предполагаемый ген приходится в среднем по 2 SNP. Никакой другой тип геномных различий не способен обеспечить такую плотность картирования. Помимо высокой плотности, SNP имеют очень низкое число мутаций на поколение (∼ 10 - 8), что делает их удобными маркерами для изучения молекулярной эволюции (22, 23).
Практический интерес к SNP сильно возрос в процессе реализации проектов по определению полных нуклеотидных последовательностей у ви-670
дов сельскохозяйственных животных. Так, секвенирование ряда полных геномов крупного рогатого скота выявило наличие у них значительного числа SNP (24). В базе данных dbSNP NCBI (National Center for Biotechnological Information, США) в настоящее время их число составляет 73 439 641 (25). Позже были расшифрованы геномы лошадей (26), свиней (27), овец (28).
Востребованность SNP привела к созданию и использованию так называемых микроматриц (ДНК-чипов), представляющих собой твердый носитель небольшого размера (не больше предметного стекла для микроскопирования) с прикрепленными к нему в определенном порядке короткими олигонуклеотидами размером 8-25 п.н. или фрагментами ДНК размером более 100 п.н. (29). Создание микроматриц разной плотности для основных видов сельскохозяйственных животных помогло определить параметры изменчивости внутри и между породами, идентифицировать географическую локализацию отдельных популяций и перемешивание популяций с различным генетическим происхождением, получить информацию об эволюционных взаимоотношениях (филогенетические деревья) и определить центры происхождения и маршруты миграции, осуществить картирование генов, создать ДНК-хранилища (30). Наиболее широкое практическое применение находят коммерческие ДНК-чипы, сконструированные фирмой «Illumina, Inc.» (США), которые позволяют генотипировать почти все основные модельные виды сельскохозяйственных животных и получать информацию о генетическом разнообразии по десяткам и сотням тысяч маркеров. Для немодельных видов (включая северного оленя) из-за отсутствия информации о полном геноме создание такой SNP-матрицы пока остается неисполнимой задачей (31). Однако многими авторами показана возможность применения на таких видах коммерческих ДНК-чипов, разработанных для домашних видов животных, с целью получения информации о генетическом разнообразии и структуре популяций (32-37). В 2015 году были опубликованы первые данные об успешном применении на северных оленях ДНК-чипов для крупного рогатого скота (Illumina Bo-vineSNP50 BeadChip) и домашних овец (Illumina OvineSNP50 BeadChip) (38). Полученные результаты послужили основой для использования Bo-vineSNP50 BeadChip с целью генетической характеристики ранее не исследованных пород и популяций этого представителя рода Rangifer .
В настоящем сообщении нами впервые дана более точная генетическая характеристика трех пород домашнего северного оленя в Республике Саха (Якутия). Полученные данные могут быть использованы для совершенствования селекционно-племенной работы в оленеводстве региона.
Целью исследования была оценка биоразнообразия домашних пород северного оленя ( Rangifer tarandus ) эвенской, эвенкийской и чукотской, разводимых на территории Якутии, с использованием ДНК-чипа средней плотности BovineSNP50K BeadChip.
Методика. Анализировали биоматериал (ушные выщипы) оленей эвенской (EVN, ФГУП «Ючюгейское», n = 8), эвенкийской (EVK, НАОК «Таба», n = 11) и чукотской (харгин) пород (CHU, ОАО КМНС «Хаты-стыр», n = 7), отобранный в 2016 году в Республике Саха (Якутия). Группой сравнения служили образцы от животных из дикой якутской популяции (WLD, n = 16), собранные в 2016 году в таежной (TGA, n = 4) и тундровой (TUN, n = 12) природно-климатических зонах при экспедиционных обследованиях.
Геномную ДНК выделяли с помощью колонок Nexttec («Nexttec Biotechnologie GmbH», Германия) согласно рекомендациям фирмы-изготовителя. Качество выделенной ДНК проверяли посредством электрофоре-671
за в агарозном геле. Концентрацию ДНК в растворе оценивали по поглощению при X = 260 нм (OD2 60 ), чистоту ДНК — по соотношению поглощения при длинах волн 260 и 280 нм (OD2 6 0/OD280). Скрининг одиночных нуклеотидных полиморфизмов проводили с использованием микроматрицы средней плотности Bovine SNP50 BeadChip («Illumina, Inc.», США), включающей 54609 SNP.
Программное обеспечение PLINK v1.07 (39) было применено для проведения контроля качества генотипирования с учетом следующих фильтров: исключение SNP, генотипированных менее чем у 90 % индиви- дуумов (--geno - 0.1); исключение индивидуумов, у которых отсутствуют более чем 10 % SNP от их общего числа (--mind - 0.1); исключение локусов с частотой минорных аллелей менее 5 % (--maf - 0.05); исключение маркеров, не соответствующих критерию χ2 на равновесие в популяции по Харди-Вайнбергу (р < 1х10-6); удаление SNP со значениями неравновесия по сцеплению (linkage disequilibrium, LD) между однонуклеотидными полиморфизмами r2 > 0,05; окно размером 50 SNP с шагом 5 маркеров. Дополнительно для оценки качества генотипирования SNP использовались критерии GC Score (качество чтения SNP) и GT Score (уровень кластеризации SNP) не менее 0,5 (50 %) (40).
Для построения диаграммы Венна, определяющей уникальные и общие для каждой породы SNP, применяли R пакет VennDiagram. Рассчитывали значения показателей наблюдаемой (Ho) и ожидаемой (He) гетерозиготности, аллельного разнообразия с применением процедуры рарифи-кации (Аr), коэффициента инбридинга (FIS) (41). Визуализацию осуществляли в пакете R diveRsity (42). Многомерное шкалирование (MDS), осно- ванное на матрице дистанций идентичности по состоянию (identical-by- state, IBS), было выполнено в PLINK 1.07 (--cluster, --mds-plot 4), последующая визуализация — в R пакете ggplot2. Степень генетической дифферен- циации популяций оценивали на основании попарных значений FST (43), рассчитанных в пакете R diveRsity (42). Результаты оценки генетической структуры популяций и межпопуляционных связей были получены с по- мощью программы Admixture 1.23 (44) с последующей визуализацией в R пакете pophelper (45).
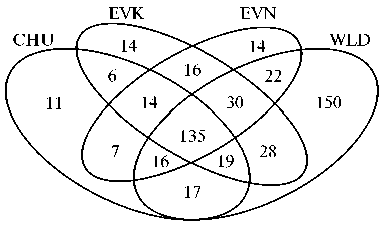
Рис. 1. Диаграмма Венна, отражающая распределение числа уникальных и общих SNP для исследуемых групп северного оленя ( Rangifer tarandus ): CHU — чукотская (харгин), EVK — эвенкийская, EVN — эвенская породы; WLD — популяция дикого северного оленя (Республика Саха—Якутия, 2016-2017 годы).
При статистической обработке данных рассчитывали среднюю арифметическую ( М ) и стандартные ошибки средней (± m ).
Результаты. После контроля качества осталось 22807 SNP, из которых 512 были полиморфными и использовались для дальнейшего анализа. Небольшое число полиморфных маркеров объясняется уменьшением числа полиморфных SNP с увеличением филогенетических дистанций между семействами Bovidae и Cervidae, что также проявляется в снижении общего количества генотипи- рованных SNP (call rate) с 99,54 до 61,19 % (37).
Установлено 135 SNP, которые были общими для всех групп на диаграмме Венна (рис. 1). Среди домашних пород количество уникальных SNP оказалось максимальным у EVN и EVK (14 SNP). У оленей чукот- ской породы было детектировано 11 уникальных SNP. Если рассматривать число специфичных аллелей для двух популяций, то оно было выше для пары EVN—EVK (16 SNP), наименьшим — для EVK—CHU (6 SNP). Олени домашней популяции уступали диким по числу уникальных SNP (14 против 150), при этом общее количество SNP, специфичных только для домашней популяции, составило 349.
1. Генетическое разнообразие домашних пород северного оленя ( Rangifer taran-dus ), оцененное на основании профилей одиночных нуклеотидных полиморфизмов (Республика Саха—Якутия, 2016-2017 годы)
|
Порода |
] n 1 |
H o ( М ± m ) |
H e ( М ± m ) |
] FIS 1 |
A r ( М ± m ) |
|
CHU |
7 |
0,180±0,011 |
0,156±0,008 |
- 0,124 |
1,488±0,022 |
|
EVK |
11 |
0,161±0,009 |
0,153±0,008 |
- 0,047 |
1,487±0,020 |
|
EVN |
8 |
0,164±0,010 |
0,149±0,008 |
- 0,089 |
1,471±0,021 |
П р и м е ч а н и е. CHU — чукотская (харгин), EVK — эвенкийская, EVN — эвенская породы; Ho — наблюдаемая гетерозиготность, He — ожидаемая гетерозиготность, Аr — аллельное разнообразие, рассчитанное с применением процедуры рарификации, F IS — коэффициент инбридинга.
Наибольшую степень наблюдаемой гетерозиготности отмечали у чукотской породы — Ho = 0,180±0,011 (табл. 1). Ранее было установлено, что она превосходила эвенскую и эвенкийскую породы по генетическому разнообразию, оцененному с использованием микросателлитов (18). Выборка эвенкийской породы характеризовалась минимальными значения-
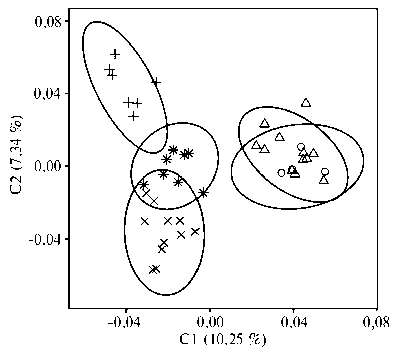
Рис. 2. Графическое изображение результатов многомерного шкалирования (MDS) образцов, полученных от особей северного оленя ( Rangifer tarandus ) разных пород и генотипированных с использованием Bovine SNP50 BeadChip: + — чукотская (харгин), х — эвенкийская, * — эвенская породы; оид— соответственно таежная и тундровая популяции дикого северного оленя; ось Х — главная компонента 1 (C1), ось Y — главная компонента 2 (C2) (Республика Саха— Якутия, 2016-2017 годы).
ми наблюдаемой гетерозиготности (Ho = 0,161±0,009). Показатель аллельного разнообразия (Ar), скорректированный методом рарифи-кации, не выявил существенных различий между породами: значения Ar варьировали от 1,471 (EVN) до 1,488 (CHU). Все три породы имели незначительный избыток гетерозигот, о чем свидетельствовали отрицательные значения коэффициента инбридинга (FIS) — от -0,124 (CHU) до -0,047 (EVN).
Диаграмма расположения исследованных образцов в про- странстве, полученная на основании анализа SNP профилей с помощью метода многомерного шкалирования (MDS), представлена на рисунке 2. Первая координата объясняла большую часть (10,25 %) общей дисперсии всего массива данных и четко разделяла домашнюю и дикую популяции, в то вре- мя как вторая координата, отражающая около 8 % общей изменчивости, позволила дифференцировались домашние породы. Наибольшей генетической близостью с незначительным перекрыванием массивов характеризовались особи эвенской и эвенкийской пород. Смешение можно объяснить более близким географическим расположением племенных хозяйств, где содержатся эти пород. Дикие северные олени таежной и тундровой популяций формировали еди- ный кластер, что указывает на их генетическое сходство.
Расчет значений FST при парном сравнении (табл. 2) показал сходство полученных данных с результатами MDS-анализа: минимальное значение (FST = 0,006) было установлено для эвенской и эвенкийской пород, что подтверждает их большую генетическую близость. Олени чукотской породы оказались равноудалены от эвенской (FST = 0,098) и эвенкийской (FST = 0,095) пород и наиболее дифференцированы от дикой популяции (F ST = 0,101).
2. Дифференциация исследуемых пород северного оленя ( Rangifer tarandus ) по показателю FST при парном сравнении, рассчитанном на основании SNP профилей (Республика Саха—Якутия, 2016-2017 годы)
Группа CHU EVK EVN WLD
CHU 0,0000
EVK 0,0953 0,0000
EVN 0,0981 0,0632 0,0000
WLD 0,1007 0,0659 0,0597 0,0000
Прим еч ани е. CHU — чукотская (харгин), EVK — эвенкийская, EVN — эвенская породы; WLD — популяция дикого северного оленя.
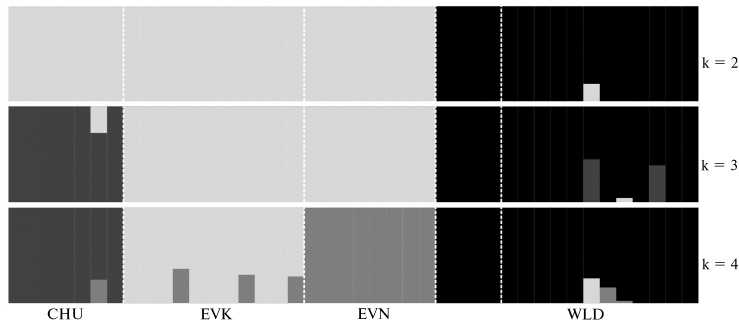
Рис. 2. Генетическая структура изучаемых пород северного оленя ( Rangifer tarandus ) по результатам адмикс-анализа образцов, генотипированных с использованием Bovine SNP50 BeadChip, для числа кластеров k от 2 до 4: CHU — чукотская (харгин), EVK — эвенкийская, EVN — эвенская породы; WLD — популяция дикого северного оленя (Республика Саха—Якутия, 2016-2017 годы).
На основании адмикс-анализа генетической структуры исследуемых пород северного оленя (рис. 2) при числе кластеров k = 2 была установлена четкая дифференциация домашних пород от дикой популяции. При k = 3 наблюдалось формирование особями чукотской породы собственного кластера, в то время как животные эвенской и эвенкийской пород кластеризовались вместе. При k = 4 порода образовала отдельный кластер с высокой степенью генетической обособленности. Следует, однако, указать на наличие среди оленей чукотской и эвенкийской пород животных, имеющих смешанное генетическое происхождение, близкое к EVN. Особи дикой популяции, используемые в качестве группы сравнения, сохраняли членство в собственном кластере при k от 2 до 4, хотя у некоторых из них были обнаружены проявления адмиксии домашних северных оленей.
Имеется достаточное число публикаций, демонстрирующих успешные исследования немодельных видов с использованием коммерческого ДНК-чипа BovineSNP50K BeadChip, разработанного для крупного рогатого скота. Так, G.D. Haynes и E.K Latch. (35) охарактеризовали двух представителей семейства Cervidae : чернохвостого ( Odocoileus hemionus ) и белохвостого оленя ( O. virginianus ). Авторы смогли успешно генотипировать 38,7 % локусов, из которых 5 % ( n = 1068) оказались полиморфными. 674
R. Kasarda с соавт. (37) исследовали два вида семейства Cervidae — благородного оленя ( Cervus elaphus ) и лань ( Dama dama ). Всего было успешно генотипировано 94,76 % SNP, из которых 5,24 % (1542) оказались полиморфными. M. Tokarska с соавт. (46) выявили 960 полиморфных локусов у европейского бизона ( Bison bonasus ). C. Pertoldi с соавт. (32) идентифицировали 929, 1403 и 1524 полиморфных SNP соответственно у европейского бизона и двух подвидов американского бизона — равнинного ( B. bison bison ) и лесного ( B. bison athabascae ). Авторы показали, что системы генотипирования SNP, разработанные для домашних видов, могут быть эффективны на немодельных видах, прошедших через «бутылочное горлышко», у которых гетерозиготность других маркеров, таких как микросателлиты, очень низка (32, 46). SNP-маркеры применяют не только при изучении биоразнообразия и эволюционных взаимоотношений, но и для определения достоверности происхождения (47).
Таким образом, мы успешно использовали ДНК-чип, разработанный для крупного рогатого скота, при генетической дифференциации пород северного оленя. В результате исследований одиночных нуклеотидных полиморфизмов дана популяционно-генетическая характеристика трех пород северного оленя — чукотской (харгин), эвенской и эвенкийской, разводимых на территории Республики Саха (Якутия). Оленеводство в этом регионе не только связано с сельскохозяйственным сектором, но и составляет неотъемлемую часть культуры коренных народов. Полученные данные могут быть использованы для разработки программ сохранения и рационального использования этого важнейшего для человека вида животных.
Список литературы Популяционно-генетическая характеристика домашнего северного оленя в Республике Якутия на основании полногеномного SNP анализа
- Азарова Л.В. Современное состояние оленеводства в Якутии. Молодой ученый, 2013, 5: 831-833.
- Роббек Н.С., Абрамов А.Ф. Эвенская порода оленей Якутии: мясная продуктивность, биологическая и пищевая ценность: монография/Под ред. А.Д. Решетникова. Новосибирск, 2017 (ISBN 978-5-4379-0519-7).
- Окороков А.И. О состоянии и развитии домашнего северного оленеводства в Республике Саха (Якутия). Вестник Северо-Восточного федерального университета имени М.К. Аммосова, 2013, 10(3): 36-41.
- Сыроватский Д.И. Меры пресечения спада оленеводства. Достижения науки и техники АПК, 2006, 5: 10-11.
- Президентская программа социально-экономического развития села Республики Саха (Якутия) на 2002-2006 годы. Утв. распоряжением Президента Республики Саха (Якутия) от 23 декабря 2002 года ¹ 328-РП.
- Закон Республики Саха (Якутия) от 11 июля 2007 года, З ¹ 991-III. О государственной целевой программе «Социально-экономическое развитие села Республики Саха (Якутия) на 2007-2011 годы».
- Государственная целевая программа «Социально-экономическое развитие села Республики Саха (Якутия) на 2012-2016 годы». Утв. Распоряжением Правительства РС (Я) № 1394-р от 24.12.2011 г.
- Алексеев Е.Д. Технология круглогодичного изгородного содержания домашних оленей в условиях таежной зоны Якутии. Автореф. канд. дис. Якутск, 2009.
- Племенная работа в оленеводстве. Информационный портал ФГБОУ ВО «Якутская государственная сельскохозяйственная академия», Институт дополнительного профессионального образования. Режим доступа: http://reindeer.sakha-plem.ru/plemennaya-rabota-v-olenevodstve/. Без даты.
- Мухачев А.Д. Оленеводство. М., 1990 (ISBN 5-10-000725-7).
- Roed K.H. Genetic differences at the transferrin locus in Norwegian semidomestic and wild reindeer (Rangifer tarandus L.). Hereditas, 1985, 102: 199-206.
- Shubin P.N. Genetics transferrin of European reindeer and moose. Genetics, 1988, 5(1): 37-41.
- Flagstad O., Røed K.H. Refugial origins of reindeer (Rangifer tarandus L.) inferred from mitochondrial DNA sequences. Evolution, 2003, 57(3): 658-670.
- Cronin M.A., MacNeil M.D., Patton J.C. Variation in mitochondrial DNA and microsatellite DNA in caribou (Rangifer tarandus) in North America. J. Mammalogy, 2005, 86: 495-505.
- Ball M.C., Finnegan L., Manseau M., Wilson P. Integrating multiple analytical approaches to spatially delineate and characterize genetic population structure: an application to boreal caribou (Rangifer tarandus caribou) in central Canada. Conserv. Genet., 2010, 11: 2131-2143 ( ) DOI: 10.1007/s10592-010-0099-3
- McDevitt A.D., Mariani S., Hebblewhite M., De Cesare N.J., Morgantini L., Seip D., Weckworth B.V., Musiani M. Survival in the Rockies of an endangered hybrid swarm from diverged caribou (Rangifer tarandus) lineages. Mol. Ecol., 2009, 18: 665-679 ( ) DOI: 10.1111/j.1365-294X.2008.04050.x
- Wilson G.A., Strobeck C., Wu L., Coffin J.W. Characterization of microsatellite loci in caribou Rangifer tarandus, and their use in other artiodactyls. Mol. Ecol., 1997, 65: 697-699 ( ) DOI: 10.1046/j.1365-294X.1997.00237.x
- Харзинова В.Р., Гладырь Е.А., Федоров В.И., Романенко Т.М., Шимит Л.Д., Лайшев К.А., Калашникова Л.А., Зиновьева Н.А. Разработка мультиплексной панели микросателлитов для оценки достоверности происхождения и степени дифференциации популяций северного оленя (Rangifer tarandus). Сельскохозяйственная биология, 2015, 50(6): 756-765 ( ) DOI: 10.15389/agrobiology.2015.6.756rus
- Kharzinova V.R., Dotsev A.V., Kramarenko A.S, Layshev K.A., Romanenko T.M., Solov'eva A.D., Deniskova T.E., Kostyunina O.V., Brem G., Zinovieva N.A. Study of the allele pool and the degree of genetic Introgression of semi-domesticated and wild populations of reindeer (Rangifer tarandus L., 1758) using microsatellites. Agricultural Biology, 2016, 51(6): 811-823 ( ) DOI: 10.15389/agrobiology.2016.6.811eng
- Денискова Т.Е., Сермягин А.А., Багиров В.А., Охлопков И.М., Гладырь Е.А., Иванов Р.В., Брем Г., Зиновьева Н.А. Сравнительное исследование информативности STR и SNP маркеров для внутривидовой и межвидовой дифференциации рода Ovis. Генетика, 2016, 52(1): 90-96 ( ) DOI: 10.7868/S0016675816010021
- Li J., Butler J.M., Tan J., Lin H., Royer S., Ohler L., Shaler T.A., Hunter J.M., Pollart D.J., Monforte J.A., Becker C.H. Single nucleotide polymorphism determination using primer extension and time off mass spectrometry. Electrophoresis, 1999, 20: 1258-1265.
- Crow J.F. Spontaneous mutation as a risk factor. Exp. Clin. Immunogenet., 1995, 12: 121-128.
- Li W.H., Ellsworth D.L., Krushkal J., Chang B.H., Hewett-Emmet D. Rates of nucleotide substitution in primates and rodents and the generation-time effect hypothesis. Mol. Phylogenet. Evol., 1996, 5: 182-187.
- The Bovine HapMap Consortium Genome-Wide Survey of SNP Variation Uncovers the Genetic Structure of Cattle Breeds. Science, 2009, 324: 528-532.
- NCBI Resource Coordinators. Database resources of the National Center for Biotechnology Information. Nucleic Acids Research, 2015, 43: 6-17 ( ) DOI: 10.1093/nar/gku1130
- Wade C.M., Giulotto E., Sigurdsson S., Zoli M., Gnerre S., Imsland F., Lear T.L., Adelson D.L., Bailey E., Bellone R.R., Blöcker H., Distl O., Edgar R.C., Garber M., Leeb T., Mauceli E. Genome sequence, comparative analysis, and population genetics of the domestic horse. Science, 2009, 326: 865-867 ( ) DOI: 10.1126/science.1178158
- Archibald A.L., Bolund L., Churcher C., Fredholm M., Groenen M.A., Harlizius B., Lee K.T., Milan D., Rogers J., Rothschild M.F., Uenishi H., Wang J., Schook L.B., Swine Genome Sequencing Consortium. Pig genome sequence -analysis and publication strategy. BMC Genomics, 2010, 11: 438-442 ( ) DOI: 10.1186/1471-2164-11-438
- Jiang Y., Xie M., Chen W. et al. The sheep genome illuminates biology of the rumen and lipid metabolism. Science, 2014, 344: 1168-1173 ( ) DOI: 10.1126/science.1252806
- Hacia J.G., Collins F.S. Mutation analysis using oligonucleotide microarrays. J. Med. Genet., 1999, 36: 730-736.
- Глазко В.И. Молекулярная биология для животноводства. Farm animals, 2012, 1: 24-29.
- Seeb J.E., Carvalho G.R., Hauser L., Naish K., Roberts S., Seeb L.W. Single-nucleotide polymorphism (SNP) discovery and applications of SNP genotyping in nonmodel organisms. Molecular Ecology Resources, 2011, 11: 1-8 ( ) DOI: 10.1111/j.1755-0998.2010.02979.x
- Pertoldi C., Wójcik J.M, Tokarska M., Kawałko A., Kristensen T.N., Loeschcke V., Gregersen V.R., Coltman D., Wilson G.A., Randi E., Henryon M., Bendixen C. Genome variability in European and American bison detected using BovineSNP50 BeadChip. Conservation Genetics, 2010, 11: 627-634.
- Miller J.M., Poissant J., Kijas J.W., Coltman D.W., International Sheep Genomics Consortium. A genome-wide set of SNPs detects population substructure and long range disequilibrium in wild sheep. Molecular Ecology Resources, 2011, 11(2): 314-322 ( ) DOI: 10.1111/j.1755-0998.2010.02918.x
- Ogden R., Baird J., Senn H., McEwing R. The use of cross-species genome-wide arrays to discover SNP markers for conservation genetics: a case study from Arabian and scimitar-horned oryx. Conservation Genetics Resources, 2012, 4: 471-473.
- Haynes G.D., Latch E.K. Identification of novel single nucleotide polymorphisms (SNPs) in deer (Odocoileus spp.) using the BovineSNP50 BeadChip. PLoS ONE, 2012, 7: e36536 ( ) DOI: 10.1371/journal.pone.0036536
- Денискова Т.Е., Охлопков И.М., Сермягин А.А., Гладырь Е.А., Багиров В.А., Сёлкнер И., Мамаев Н.В., Брем Г., Зиновьева Н.А. Полногеномное SNP-сканирование снежного барана Ovis nivicola. Доклады Академии наук, 2016, 469(5): 625-630.
- Kasarda R., Moravcíková N., Židek R., Mészáros G., Kadlečík O., Trakovická A., Pokorádi J. Investigation of the genetic distances of bovids and cervids using BovineSNP50k BeadChip. Arch. Anim. Breed., 2015, 58: 57-63 ( ) DOI: 10.5194/aab-58-57-2015
- Kharzinova V.R., Sermyagin A. A., Gladyr E.A., Okhlopkov I. M., Brem G., Zinovieva N.A. A study of applicability of SNP chips developed for bovine and ovine species to whole-genome analysis of reindeer Rangifer tarandus. Journal of Heredity, 2015, 106(6): 758-761 ( ) DOI: 10.1093/jhered/esv081
- Purcell S., Neale B., Todd-Brown K., Thomas L., Ferreira M.A.R., Bender D., Maller J., Sklar P., de Bakker P.I., Daly M.J., Sham P.C. PLINK: a tool set for whole-genome association and population-based linkage analyses. American Journal of Human Genetics, 2007, 81: 559-575 ( ) DOI: 10.1086/519795
- Fan J.B., Oliphant A., Shen R., Kermani B.G., Garcia F., Gunderson K.L., Hansen M., Steemers F., Butler S.L., Deloukas P., Galver L., Hunt S., McBride C., Bibikova M., Rubano T., Chen J., Wickham E., Doucet D., Chang W., Campbell D., Zhang B., Kruglyak S., Bentley D., Haas J., Rigault P., Zhou L., Stuelpnagel J., Chee M.S. Highly parallel SNP genotyping. Cold Spring Harb. Symp. Quant. Biol., 2003, 68: 69-78.
- Hartl D.L., Clark A.G. Principles of population genetics. Sunderland, Massachusetts, 1997.
- Keenan K., McGinnity P., Cross T.F., Crozier W.W., Prodohl P.A. diveRsity: An R package for the estimation of population genetics parameters and their associated errors. Methods in Ecology and Evolution, 2013, 4: 782-788 ( ) DOI: 10.1111/2041-210X.12067
- Weir B.S., Cockerham C.C. Estimating F-statistics for the analysis of population structure. Evolution, 1984, 38: 1358-1370.
- Alexander D.H., Novembre J., Lange K. Fast model based estimation of ancestry in unrelated individuals. Genome Res., 2009, 19: 1655-1664 ( ) DOI: 10.1101/gr.094052.109
- Francis R.M. pophelper: An R package and web app to analyse and visualise population structure. Molecular Ecology Resources, 2016 ( ) DOI: 10.1111/1755-0998.12509
- Tokarska M., Marshall T., Kowalczyk R., Wójcik J.M., Pertoldi C., Kristensen T.N., Loeschcke V., Gregersen V.R., Bendixen C. Effective ness of microsatellite and SNP markers for parentage and identity analysis in species with low genetic diversity: the case of European bison. Journal of Heredity, 2009, 103: 326-332 ( ) DOI: 10.1038/hdy.2009.73
- Oleński K., Kamiński S., Tokarska M., Hering D.M. Subset of SNPs for parental identification in European bison Lowland-Białowieża line (Bison bonasus bonasus). Conservation Genetics Resources, 2017, First Online 13 May 2017: 1-6 ( ) DOI: 10.1007/s12686-017-0768-3


