Прогнозирование появления высокотрансмиссивных штаммов вируса Sars-Cov-2 на территории Санкт-Петербурга с использованием рекуррентной нейронной сети
Автор: Кустова Д.В., Кириенко А.Н., Мартынкевич И.С.
Журнал: Вестник гематологии @bulletin-of-hematology
Рубрика: Оригинальные статьи
Статья в выпуске: 1 т.18, 2022 года.
Бесплатный доступ
В 2020 году Всемирная организация здравоохранения (ВОЗ) объявила тяжелый острый респираторный синдром, вызванный вирусом SARS-CoV-2, пандемией. Трансмиссивность SARS-CoV-2 связывают с аффинностью связывания спайкового S-белка вируса с рецептором ангиотензинпревращающего фермента 2 (АПФ2), что является ключевым событием при проникновении вируса в клетку. Не случайно большинство вакцин нацелено на блокирование S-белка для невозможности связывания с АПФ2. Предсказание возникновения штаммов с мутациями, приводящих к повышению аффинности комплекса S-белок/ АПФ2, является важной задачей для предотвращения новых вспышек эпидемии и быстрого реагирования на появление высококонтагиозных штаммов. Предсказание появления высоко-трансмиссивных штаммов является важной медико-биологической задачей, в том числе и онкогематологии, поскольку именно пациенты с иммунодефицитом и находящиеся на иммуносупрессивной терапии подвержены наибольшему риску заражения и тяжелого течения COVID-19. В нашей статье был проведен анализ вариабельности аминокислотных последовательностей S-белка SARS-CoV-2, полученных из клинических образцов на территории Санкт-Петербурга с 15 марта 2020 года по 16 июня 2021 года. На основе проанализированных последовательностей с помощью рекуррентной нейронной сети было предсказано появление новых мутаций в рецептор-связывающем мотиве и проведена оценка их влияния на связывание с АПФ2 рецептором. Было предсказано появление мутаций в позициях L455, R457, N481, T500 и G504 и показано, что данные мутации усиливают аффинность связывания с АПФ2 увеличивая трансмиссивность.
Sars-cov-2, s-белок, ангиотензинпревращающий фермент 2, мутации, молекулярный докинг, нейронные сети
Короткий адрес: https://sciup.org/170194034
IDR: 170194034
Текст научной статьи Прогнозирование появления высокотрансмиссивных штаммов вируса Sars-Cov-2 на территории Санкт-Петербурга с использованием рекуррентной нейронной сети
Введение. Первые случаи нового заболевания, приводящего к развитию тяжёлого острого респираторного синдрома, были зарегистрированы в конце 2019 года в китайском городе Ухань. Вскоре стало известно, что болезнь вызвана новым одноцепочечным РНК(+)-вирусом SARS-CoV-2, относящимся к семейству β-коронавирусов. В настоящее время болезнь, получившая название COVID-19 (аббревиатура от англ. COronaVIrus Disease 2019 — коронавирусная инфекция 2019 года), распространилась по всему миру и приняла характер пандемии [1,2].
Проникновение вирусных частиц внутрь клетки-хозяина происходит за счет связывания гликозилированных S-белков, которые находятся на поверхности вируса, с рецептором ангио-тензинпревращающего фермента 2 (АПФ2). Внутри клетки происходит репликация вирусной РНК, синтез структурных белков и сборка вирусных частиц, после чего упакованные вирусные частицы высвобождаются через лизосомы [3,4].
Вирус SARS-CoV-2 вызывает развитие респираторной инфекции, оказывающей также значительное влияние на систему кроветворения и гемостаз. Среди наиболее распространенных гематологических изменений отмечают лимфопению, нейтрофилию, эозинопению, тромбоцитопению и тромбоцитоз [5]. Кроме того, у пациентов с тяжелым течением заболевания отмечалось аномальное присутствие предшественников эритроцитов в периферической крови [6,7].
Значительным открытием стало обнаружение рецептора АПФ2 на поверхности гемопоэтических стволовых клеток и клеток-предшественников [8,9]. Важной особенностью является тот факт, что клетки-предшественники эритроцитов - ERP-S2 (CD71+ CD235A-) и ERP-S3 (CD71+ CD235a+) способны инфицироваться SARS-CoV-2, который успешно амплифици-ровал свой геном внутри этих клеток [9]. Именно связывание S-белка с рецептором АПФ2 является ключевым этапом инфицирования. При изучении этого процесса было показано, что S-белок, находящийся на поверхности вириона в тримерном состоянии, связывается с АПФ2 и расщепляется на субъединицы S1 и S2 [10–12]. Субъединица S1 содержит домен связыва- ния рецептора (RBD, 319-541 а.к.), который непосредственно связывается с доменом пептидазы (PD) рецептора АПФ2, в то время как S2 отвечает за слияние мембран. В структуре RBD особо выделяют рецептор-связывающий мотив RBM (437-508 а.к.) – участок, содержащий большинство контактирующих аминокислотных остатков S-белка, которые связываются с АПФ2 рецептором. В составе S1 субъединицы важным является также N-терминальный домен (NTD), который хоть и является менее иммуногенным, чем RBD, однако, недавние исследования показали способность анти-NTD антител нейтрализовать SARS-CoV-2 [12,13].
Важно отметить, что RBD является наиболее изменчивым у родов Coronavirinae и рассматривается как ответственный за вирусную трансмиссивность и адаптацию [1,14]. Шесть аминокислот SARS-CoV-2 являются критическими для связывания с АПФ2 рецептором и определения видоспецифичности – L455, F486, Q493, S494, N501 и Y505. Интересным является тот факт, что 5 из шести данных аминокислот не соответствуют вирусу SARS-CoV, который также проникает в клетки за счет связывания с АПФ2 рецептором [15]. Кроме того, для SARS-CoV-2 были получены доказательства существования положительного отбора, направленного на закрепление определенных мутаций в S-гене [16]. Поэтому, совершенно неудивительно, что внимание многих ученых направлено на изучение механизмов формирования комплекса S-белок - АПФ2, отслеживание мутаций, появляющихся в S гене, создание лекарств и вакцин, блокирующих именно S-белок SARS-CoV-2 [17–19].
В решении таких задач большую роль играют методы математической биологии. Так, компьютерное моделирование белков нашло широкое применение при анализе формирования комплекса S-белок – АПФ2-рецептор [10,20–23]. Моделирование позволяет оценить влияние мутаций в S-белке на связывание с АПФ2 рецептором, что определяет патогенность и вирулентность новых штаммов [24]. Неслучайно, наиболее быстрое распространение в популяции имеют штаммы, мутации в которых повышают связывающую способность S-белка с АПФ2 рецептором [25,26].
Эпидемиологические и биохимические исследования показывают, что трансмиссивность различных штаммов SARS-CoV-2 пропорциональна аффинности связывания [11,27]. Следовательно, трансмиссовность SARS-CoV-2 может быть теоретически оценена по аффинности связывания RBM-АПФ2. Соответственно, следует отслеживать появление мутаций, повышающих аффинность данного комплекса, поскольку именно таким мутациям благоприятствует естественный отбор [28].
Алгоритмы машинного обучения активно используются в вычислительной биологии и биоинформатике для прогнозирования мутаций. Например, нейронные сети успешно применяются для прогнозирования мутаций в вирусах гриппа [29], точечных мутаций, возникающих при выравнивании последовательностей РНК [30] и мутаций в определенных сайтах [31]. Несмотря на разнообразие алгоритмов, возникает проблема с динамическим моделированием последовательных данных. Так, появление рекуррентных нейронных сетей (RNN) c долговременной и кратковременной памятью (LSTM) нашло применение для решения проблемы управления нелинейной динамикой в исследованиях временных рядов [32]. Было показано, что модели, основанные на RNN превосходят модели на основе логистической регрессии (LR) и методе опорных векторов (SVM) [33]. Это связано с тем, что модели на основе RNN в отличие от LR и SVM могут использовать свою внутреннюю память для обработки последовательностей произвольной длины. Модель на основе RNN-LSTM использовалась для предсказания возникновения мутаций в геномной последовательности SARS-CoV-2 и показала увеличение количества замен C>T в будущем времени [34], а также легла в основу конвейера «Tempel», предсказывающего вероятность появления мутаций вируса гриппа в следующем сезоне, опираясь на временные данные о последовательностях гемагглютинина [33].
Помимо выбора модели и обучающего алгоритма необхо- димо правильно представить входные данные. Применение нейронных сетей к численно преобразованным последовательностям аминокислот или нуклеиновых кислот демонстрирует неоднозначные результаты [30]. Поэтому одним из решений проблемы подготовки входных данных является применение методов обработки естественного языка (NLP) [33,35,36]. NLP позволяет представить аминокислотную последовательность в виде многомерных векторов. В частности, ProtVec преобразует аминокислотную последовательность в виде перекрывающихся «биологических» слов, состоящих из 3 аминокислот, представленных в векторе 1*100 [32]. Предсказание возникновения новых штаммов с новыми свойствами, в частности, повышенной трансмиссивностью за счет более эффективного связывания вирусных частиц с рецепторами на поверхности клеток, позволяет заранее решить ряд эпидемиологических задач в период пандемии – оценить эффективность уже существующих вакцин против возможных штаммов, дает время на создание новых вакцин и на подготовку системы здравоохранения к новым «волнам» эпидемии. Разработка механизмов оценки появления новых трансмиссивных штаммов найдет свое значение и в фундаментальной науке, позволив, например, объяснить влияние SARS-CoV-2 на гематологический статус пациента. Именно поэтому целью нашей работы стало предсказание возникновения мутантных штаммов с повышенной трансмиссивностью SARS-CoV-2 на территории Санкт-Петербурга. Для этого нами была изучена вариабельность S-белка у штаммов SARS-CoV-2, выделенных на территории Санкт-Петербурга с 15 марта 2020 года по 16 июня 2021 года. На основе проанализированных последовательностей методами машинного обучения нами было предсказано появление новых мутаций и проведена оценка их влияния на связывание S-белка с АПФ2 рецептором, т.е. транс-миссивность штаммов.
Материалы и методы. Для проведения анализа использовались данные, представленные на сайте GISAID (https://www. [37]. В анализ брали аминокислотные последовательности S-белка SARS-CoV-2, полученные из образцов клинического материала в период с середины марта 2020 года по середину июня 2021 года. По состоянию на 16 июня 2021 года в этой базе содержалось 852 полных последовательности S-белка SARS-CoV-2, полученных на территории Санкт-Петербурга. В качестве референсной последовательности использовали Wuhan-Hu-1 (EPI_ISL_402124) [38].
Филогенетический анализ проводили с использованием пакета BioPython [39] для Python и свободного программного обеспечения UGENE v.39.0 ru/) [40]. Множественное выравнивание последовательностей проводили c помощью алгоритма MUSCLE [41]. Для построения филогенетического дерева использовали метод максимального правдоподобия (ML).
Для предсказания мутаций использовали конвейер, основанный RNN, представленный в работах Rui Yin, а также Shashata Sawmya с соавторами [31,33].
Для предварительной подготовки входных данных следовали протоколу, представленному в работе Rui Yin [33]. Выровненные последовательности были разделены на три волны заболеваемости в соответствии с данными Роспотребнадзора. Первая волна – с марта 2020 по сентябрь 2020, вторая волна – с октября 2020 по март 2021 и третья волна – с апреля 2021 по июнь 2021. Входные данные представлены в виде 1254*100-мерного векторного пространства на основе ProtVec [36]. Прогнозирование мутаций проводили на фрагменте RBM мотива (437-507 а.к.).
Для прогнозирования мутировавшей аминокислоты в предсказанной позиции использовали таблицу вероятности мутаций аминокислот, представленную в работе Shaomin Yan и Guang Wu [42].
Для моделирования по гомологии фрагмента S-белка SARS-CoV-2 (331-524 а.к.), содержащей предсказанные мутации в RBM мотиве, использовали сервер SWISS-MODEL (https://swissmodel. [43–47]. В качестве шаблона использовали структуру S-белка SARS-CoV-2 (7ddn) из базы данных PDB (http://www.
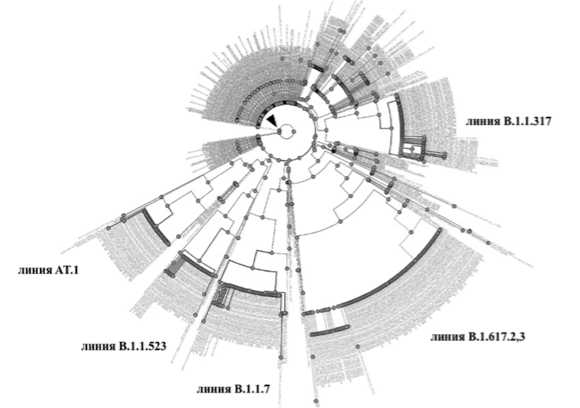
Рис. 1. Филогенетическое дерево вариантов S-белка, распространенных на территории Санкт-Петербурга. Стрелкой отмечена референсная последовательность S-белка Wuhan-Hu-1 (EPI_ISL_402124).
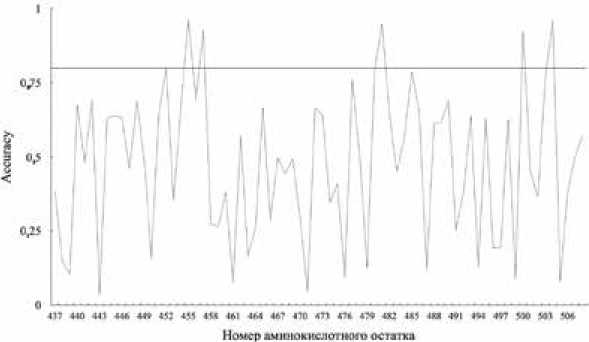
Рис. 2. Точность прогноза мутаций аминокислотных остатков. Черная линия обозначает базовую линию.
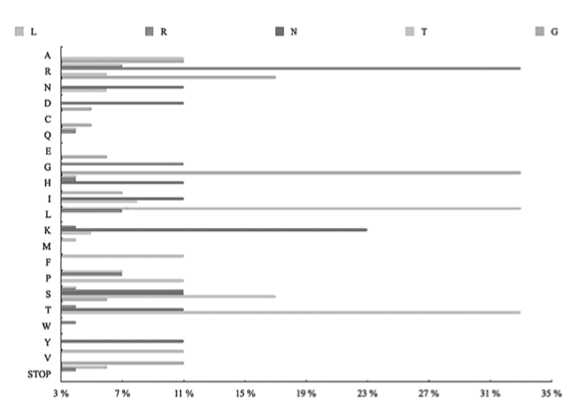
Рис. 3. Прогноз потенциальных мутаций аминокислот по позициям L455,
R457, N481, T500, G504 на основе вероятности трансляции между кодо-
нами РНК и мутированными аминокислотами. Аланин; R, Аргинин; N, Аспарагин; D, Аспарагиновая кислота; V, Валин; H, Гистидин; G, Глицин; Q, Глутамин; E, Глутаминовая кислота; I, Изолейцин; L, Лейцин; K, Лизин; M, Метионин; P, Пролин; S, Серин; Y, Тирозин; T, Треонин; W, Триптофан; F, Фенилаланин; C, Цистеин; STOP, стоп-кодон
Для моделирования взаимодействия между фрагментами, содержащими предсказанные мутации в RBM мотиве S-белка (331-524 а.к.) и АПФ2 рецептором (6M17) использовался сервер ClusPro [49–52]. Скоринг функция для отбора наиболее правдоподобных моделей:
E=0,4E_rep+(-0,4E_att )+600E_elec+1,00E_DARS, где E_rep и E_att- вклады в межмолекулярные силы Ван-дер-ваальса, E_elec-электростатическая энергия, E_DARS-потенциал, основанный на парной структуре [49–52].
Для прогнозирования аффинности в белок-белковых комплексах использовали сервер PRODIGY [53,54]. Полученные данные визуализировали с помощью программы PyMol [55].
Результаты. Анализ множественного выравнивания последовательностей, показал, что NTD область (139-168 а.к.) и RBD домен (319-541 а.к.) являются наиболее вариабельными. Филогенетический анализ вариантов S-белка показал, что на территории Санкт-Петербурга преобладают линии AT.1, B.1.1.523, B.1.1.7, B.1.617.2,3, B.1.1.317 (Рисунок 1). Далее работу вели только с фрагментом RBD домена (331–524 а.к.), поскольку именно он является ключевым в связывании S-белка с АПФ2 рецептором [12,13].
Конвейер, основанный на рекуррентной нейронной сети, предсказал 5 ключевых аминокислот, в которых может произойти мутация в ходе эволюции. Лейцин (L) в 455 положении, аргинин (R) в 457, аспарагин (N) в 481, треонин (T) в 500 и глицин (G) в 504. Результат точности (Accuracy) прогнозирования мутаций аминокислотных остатков представлен на рисунке 2. На рисунке 3 показана вероятность мутации аминокислот в предсказанных положениях в другую аминокислоту.
Для дальнейшего анализа были выбраны преобладающие замены: L455F, L455V, R457G, R457S, N481K, T500S, G504R.
С помощью сервера SWISS-MODEL были получены третичные структуры фрагментов (331–524 а.к.) S-белка SARS-CoV-2 линии B.1.617.2 и SARS-CoV-2 линии B.1.617.2 с внесенной мутацией в 455 положении, а также, референсного S-белка SARS-CoV-2 с мутациями в положении 455, 457, 481, 500, 504. С помощью сервера ClusPro было проведено молекулярное моделирование взаимодействия полученных фрагментов с АПФ2 рецептором. Визуализация моделей взаимодействия фрагментов S-белка линии B.1.617.2 и референсного S-белка SARS-CoV-2 с мутацией в ключевой аминокислоте L455 с АПФ2 рецептором представлена на рисунке 4.
Для всех построенных моделей была проведена оценка энергии и аффинности связывания. Средняя и низшая энергия связывания фрагментов (331–524 а.к.) S-белка SARS-CoV-2 с АПФ2 рецептором, полученная с помощью сервера ClusPro, представлена на рисунке 5А. Предсказание аффинности связывания моделей белок-белкового комплекса представлено на рисунке 5Б.
Обсуждение. Анализ геномных изменений вирусов, в частности SARS-CoV-2, является одним из ключевых этапов реагирования и борьбы с распространением заболеваний. Секвенирование решает широкий спектр задач, направленных на идентификацию и характеризацию вируса (в частности, разработку диагностических тест-систем), изучение передачи и географического распространения и оценку эволюции вируса (WHO). Совместно с методами компьютерного моделирования секвенирование позволяет оценить влияние мутаций на трансмиссивность. Подключение к таким исследованиям машинного обучения даёт возможность предсказать появление новых опасных мутаций, что позволяет заранее принимать меры для предотвращения распространения новых штаммов и дает время на разработку новых лекарств и вакцин, что особенно актуально для пациентов с злокачественными опухолями системы крови, характеризующихся иммунодефицитом и находящихся на иммуносупрессивной терапии.
В нашей работе мы провели филогенетический анализ аминокислотных последовательностей штаммов, выделенных в Петербурге с марта 2020 по июнь 2021 гг., и оценили их генетическое разнообразие, обращая особое внимание на линии, которые имеют мутации в S-гене. Штаммы с мутациями в S гене, которые приводят к замене аминокислот в RBD домене и усиливают связывание с АПФ2, привлекают особое внимание из-за высокой скорости распространения, такие штаммы ВОЗ обозначает как «вызывающие опасения».
Так, среди линий, выявленных в Санкт-Петербурге с марта 2020 по июнь 2021, долю в 14,41% занимает линия B.1.1.317, имеющая мутации в S-белке Q675R, D138Y, S477N, A845S (Рисунок 1). Данная линия была впервые выявлена в марте 2020 во Вьетнаме и характеризуется, кроме того, мутацией в нуклеокап-сиде A211V. Варианты с такой мутацией мало распространились в мире, однако в России в феврале – марте 2020 года занимали 26,9% [56]. Данный набор мутаций обеспечивает штамму эффективное связывание с АПФ2 рецептором и ускользание от иммунного ответа [56].
Линия B.1.1.523, имеющая долю в 4,28% в Санкт-Петербурге (Рисунок 1), несет в себе мутацию E484K, характерную и для других штаммов, например, B.1.351 (Южная Африка) и B.1.1.28 (Бразилия) и вызывает особый интерес, поскольку способна снижать нейтрализацию антителами [57]. Штаммы с мутацией E484K, которая влияет на формирование комплекса с АПФ2, быстро вытеснили другие варианты вируса и распространились на другие страны [58]. Варианты SARS-CoV-2. Новости о вспышках болезней 31 декабря 2020 г. – ВОЗ; Nelson et al., 2021) [59].
Важно отметить значительную долю (5,64%) линии АТ-1 (B.1.1.370.1) [60] в анализируемых нами последовательностях (Рисунок 1). Данная линия впервые была выявлена в Северо-Западном федеральном округе и характеризуется вставкой N679delinsKGIAL и делецией по позициям C136_Y144del, находящимися в S-белке. Данные мутации могут оказывать влияние на эффективность разрезания S-белка фурином и на эффективность проникновения вируса в клетку [61]. Данный штамм распространился не только на Северо-Западе России, но и в таких странах как Финляндия, Германия, Британия .
На июнь 2021 года 22,87% секвенированных штаммов относятся к линии B.1.617 (Рисунок 1). Филогенетический анализ показывает наличие в Санкт-Петербурге линий B.1.617.2 и B.1.617.3. Данные линии были выделены в октябре-феврале 2020-2021 гг в Индии и несут важные замены в RBD домене – L452R и T478K. Линия B.1.617.2 характеризуется большей трансмиссивностью, а антитела и сыворотки вакцинированных людей хоть и способны нейтрализовывать данный штамм, но с гораздо более низкой эффективностью [62–64]. Большая доля данных штаммов от всех секвенированных образцов связана, как с доминированием данного штамма в мире на июнь 2021, так и с увеличением количества секвенируемых образцов в Санкт-Петербурге.
При сравнении филогенетических данных штаммов из Санкт-Петербурга с данными, полученными в других регионах, выявляется ряд общих черт распространения SARS-Cov-2 в России. Так, ни в одном регионе не был выявлен исходный штамм Wuhan-Hu-1, подавляющее большинство штаммов, циркулировавших на ранних этапах пандемии, относились к линиям B.1, B.1.1 и B.1.* (кладам G, GR, и GH), также обнаруживаются штаммы с мутациями, с которым ряд исследователей связывают уклонение от иммунного ответа (Водопьянов и др., 2020, Осина и др., 2020) [65]. Особенность филогении SARS-CoV-2 в Санкт-Петербурге является появление штамма AT-1, получившего название «северо-западный» [61].
Аминокислотные последовательности S-белка штаммов, получивших распространение на территории Санкт-Петербурга, использовались как входные данные для нейронной сети. В нашей работе мы показали возможное появление мутаций в позициях – L455, R457, N481, T500 и G504. В дальнейший анализ брали варианты S-белка с аминокислотными заменами, имеющими наибольшую вероятность (Рисунок 3) – L455F, L455V, R457S, R457G, N481K, T500S, G504R.
На основе предсказанных мутаций были построены модели взаимодействия фрагментов S-белка (331-524 а.к.) с измененными аминокислотами с АПФ2 (Рисунок 4). Оценку влияния
Рис 4. Визуализация моделей взаимодействия фрагмента S-белка SARS-CoV-2 линии B.1.617.2 и референсного S-белка SARS-CoV-2 c АПФ2 рецептором. Черным прямоугольником выделена область, в которой происходит взаимодействие с АПФ2. Пунктирные линии – длина связи меньше 4 Å. А) Фрагмент референсного S-белка SARS-CoV-2. Б) Фрагмент референсного S-белка SARS-CoV-2 с мутацией в L455F. В) Фрагмент референсного S-белка SARS-CoV-2 с мутацией в L455V. Г) Фрагмент S-белка SARS-CoV-2 линии B.1.617.2. Д) Фрагмент S-белка SARS-CoV-2 линии B.1.617.2 с мутацией в L455F. Е) Фрагмент S-белка SARS-CoV-2 линии B.1.617.2 с мутацией в L455V.
Рис. 5. А) Средняя и низшая энергия связывания фрагментов (331–524 а.к.) S-белка SARS-CoV-2 с АПФ2 рецептором. Б) Предсказанная аффинность моделей белок-белкового комплекса. DELTA - Фрагмент S-белка SARS-CoV-2 линии B.1.617.2, REF - Фрагмент S-белка SARS-CoV2 Wuhan-Hu-1.
таких мутаций проводили в сравнении с соответствующим участком S-белка исходного штамма Wuhan-Hu-1. Мы показали, что данные мутации увеличивают аффинность связывания, что свидетельствует о формировании более стабильных комплексов (Рисунок 5Б).
Мутация в позиции L455 представляет особый интерес, поскольку данная аминокислота напрямую взаимодействует с АПФ2 рецептором в позициях D30, K31 и H34 [10,66]. Наши данные говорят о возможности возникновения замен L455V и L455F, причем мутация L455F предпочтительнее исходя из значений энергии связывания (-11,1 ккал/моль-1 и -12,1 ккал/ моль-1 соответственно) (Рисунок 5A). Штаммы с заменой L455F уже были идентифицированы в США [28]. Такая замена хоть и встречается с низкой частотой на территории США, однако, имеет наиболее высокие абсолютные изменения свободной энергии связывания из изученных авторами [28]. Примечательно предположение о том, что замена Tyr442→Leu455 повысила способность SARS-CoV-2-RBD связываться с рецептором АПФ2 по сравнению с SARS-CoV-RBD [22].
Исходя из ключевой роли данной аминокислоты в формировании комплекса S-белок – АПФ2 и полученных нами данных о более стабильных комплексах с мутациями L455V и L455F, был проведен анализ вариантов S-белка штамма B.1.617.2 (22,87% от всех проанализированных нами последовательностей) с АПФ2 рецептором (Рисунок 4Г-4Е). Аффинность комплекса S-белок (B.1.617.2) – АПФ2 составляет –11,4 ккал/моль-1, с мутацией L455F –11,8 ккал/моль-1, с мутацией - L455V –10,9 ккал/ моль-1 , что говорит о том, что данные мутации не оказывают существенного влияния на формирование комплекса (Рисунок 5). Однако данные, полученные нами на основе прогнозирования мутаций, а также тот факт, что мутация L455F уже выявлялась в популяции, говорят о том, что вероятность появления новых штаммов с такой мутацией достаточно высока. Так, на- пример, штаммы с мутацией D614G появились независимо в разных странах и закрепились в популяции, заменив исходный штамм Wuhan-Hu-1[67].Таким образом, проанализировав вариабельность S-белка у штаммов, выявленных на территории Санкт-Петербурга в период с 15 марта 2020 г. по 16 июня 2021 г., мы определили возможные сайты возникновения мутаций, предсказали преобладающие аминокислотные замены в данных сайтах и показали, что все предсказанные нами мутации оказывают влияние на связывание RBD домена S-белка с АПФ2 рецептором, увеличивая аффинность связывания. Это свидетельствует о формировании более стабильных комплексов S-белок/АПФ2-рецептор, а значит такие штаммы могут обладать большей трансмиссивностью. Полученные нами данные, а также опыт использования нейронных сетей для предсказания высококонтагиозных штаммов позволит в следующих работах оценить появление таких штаммов у когорты онкогематологи-ческих больных.
Конфликты интересов
Источник финансирования
Вклад авторов
Концепция и дизайн: Кустова Д.В., Кириенко А.Н.
Сбор и обработка данных: Кустова Д.В., Кириенко А.Н.
Представление материалов исследования: Кустова Д.В., Кириенко А.Н.
Анализ и интерпретация: Кустова Д.В., Кириенко А.Н.
Подготовка рукописи: Кустова Д.В., Кириенко А.Н., Мартын-кевич И.С.
Окончательное одобрение рукописи: Мартынкевич И.С.
Список литературы Прогнозирование появления высокотрансмиссивных штаммов вируса Sars-Cov-2 на территории Санкт-Петербурга с использованием рекуррентной нейронной сети
- Zhou P., Yang X. Lou, Wang X.G., et al. A pneumonia outbreak associated with a new Coronavirus of probable bat origin // Nature.-2020.- Vol. 579, P.270-273.
- Huang Y., Yang C., Xu X., et al. Structural and functional properties of SARS-CoV-2 spike protein: potential antivirus drug development for COVID-19 // Acta Pharmacologica Sinica.-2020.- Vol. 41, No. 9.- P.1141-1149.
- Fehr A.R., Perlman S. Coronaviruses: An Overview of Their Replication and Pathogenesis // Coronaviruses: Methods and Protocols-2015.- Vol. 1282, No. 1.- P. 1-23.
- Ghosh S., Dellibovi-Ragheb T.A., Pak E., et al. ß-Coronaviruses use lysosomal organelles for cellular egress // Cell.-2020.- Vol. 183, No. 6.- P. 1520-1535.
- Leticia de Oliveira Toledo S., Sousa Nogueira L., das Graças Carvalho M., et al. COVID-19: Review and hematologic impact // Clinica Chimica Acta.-2020.- Vol. 510.- P. 170-176.
- Bernardes J.P., Mishra N., Tran F., et al. Longitudinal Multi-omics Analyses Identify Responses of Megakaryocytes, Erythroid Cells, and Plasmablasts as Hallmarks of Severe COVID-19 // Immunity.-2020.- Vol. 53, No. 6.- P.1296-1314.
- Shahbaz S., Xu L., Osman M., et al. Erythroid precursors and progenitors suppress adaptive immunity and get invaded by SARS-CoV-2 // Stem Cell Reports.-2021.- Vol. 16, No. 5.- P.1165-1181.
- Ropa J., Cooper S., Capitano M.L., et al. Human Hematopoietic Stem, Progenitor, and Immune Cells Respond Ex Vivo to SARS-CoV-2 Spike Protein // Stem Cell Reviews and Reports.-2021.- Vol. 17, No. 1.- P.253-265.
- Huerga Encabo H., Grey W., Garcia-Albornoz M., et al. Human Erythroid Progenitors Are Directly Infected by SARS-CoV-2: Implications for Emerging Erythropoiesis in Severe COVID-19 Patients // Stem Cell Reports.-2021.- Vol. 16, No. 3.- P.428-436.
- Lan J., Ge J., Yu J., et al. Structure of the SARS-CoV-2 spike receptor-binding domain bound to the ACE2 receptor // Nature.-2020.- Vol. 581.- P.215-220.
- Hoffmann M., Kleine-Weber H., Schroeder S., et al. SARS-CoV-2 Cell Entry Depends on ACE2 and TMPRSS2 and Is Blocked by a Clinically Proven Protease Inhibitor // Cell.-2020.- Vol. 181, No. 2.- P.271-280.
- Chi X., Yan R., Zhang J., et al. A neutralizing human antibody binds to the N-terminal domain of the Spike protein of SARS-CoV-2 // Science.-2020.- Vol. 369.- P. 650-655.
- Liu L., Wang P., Nair M.S., et al. Potent neutralizing antibodies against multiple epitopes on SARS-CoV-2 spike // Nature.-2020.- Vol. 584.- P.450-456.
- Wu F., Zhao S., Yu B., et al. A new coronavirus associated with human respiratory disease in China // Nature.-2020.- Vol. 579.- P.265-269.
- Wan Y., Shang J., Graham R., et al. Receptor Recognition by the Novel Coronavirus from Wuhan: an Analysis Based on Decade-Long Structural Studies of SARS Coronavirus // Journal of Virology.-2020.- Vol. 94, No. 7.
- Zhan X.Y., Zhang Y., Zhou X., et al. Molecular evolution of SARS-CoV-2 structural genes: Evidence of positive selection in spike glycoprotein // bioRxiv.-2020.- No. 628.- P.0-3.
- Komissarov A.B., Safina K.R., Garushyants S.K., et al. Genomic epidemiology of the early stages of the SARS-CoV-2 outbreak in Russia // Nature Communications.-2021.- Vol. 12, No. 1.- P.1-13.
- Li Q., Wu J., Nie J., et al. The Impact of Mutations in SARS-CoV-2 Spike on Viral Infectivity and Antigenicity // Cell.-2020.- Vol. 182, No. 5.- P.1284-1294.
- Min L., Sun Q. Antibodies and Vaccines Target RBD of SARS-CoV-2 // Frontiers in Molecular Biosciences.-2021.- Vol. 8.- P.1-9.
- Xiantian X., Ping C., Jingfang W., et al. Evolution of the novel coronavirus from the ongoing Wuhan outbreak and modeling of its spike protein for risk of human transmission // SCIENCE CHINA Life Sciences.-2020.- Vol. 63, No. 3.- P. 457-460.
- Wrapp D., Wang N., Corbett K.S., et al. Cryo-EM structure of the 2019-nCoV spike in the prefusion conformation // Science.-2020.- Vol. 367.- P.1260-1263.
- Renhong Y., Yuanyuan Z., Yaning L., et al. Structural basis for the recognition of SARS-CoV-2 by full-length human ACE2 // Science.-2020.- Vol. 367.- P.1444-1448.
- Zhou T., Tsybovsky Y., Gorman J., et al. Cryo-EM Structures of SARS-CoV-2 Spike without and with ACE2 Reveal a pH-Dependent Switch to Mediate Endosomal Positioning of Receptor-Binding Domains // Cell Host and Microbe.-2020.- Vol. 28, No. 6.- P.867-879.
- Arul Murugan N., Javali y P.S., Jeyaraj Pandian C., et al. Computational Investigation of Increased Virulence and Pathogenesis of SARS-CoV-2 Lineage B.1.1.7 // bioRxiv.-2021.-Vol. 449.
- Khateeb J., Li Y., Zhang H. Emerging SARS-CoV-2 variants of concern and potential intervention approaches // Critical Care.-2021.- Vol. 25, No. 1.- P.1-8.
- Harvey W.T., Carabelli A.M., Jackson B., et al. SARS-CoV-2 variants, spike mutations and immune escape // Nature Reviews Microbiology.-2021.- Vol. 19, No. 7.- P.409-424.
- Walls A.C., Park Y.J., Tortorici M.A., et al. Structure, Function, and Antigenicity of the SARS-CoV-2 Spike Glycoprotein // Cell.-2020.- Vol. 181, No. 2.- P.281-292.
- Wang R., Chen J., Gao K., et al. Analysis of SARS-CoV-2 mutations in the United States suggests presence of four substrains and novel variants // Communications Biology.-2021.- Vol. 4, No. 1.- P.1-14.
- Yan S., Wu G. Application of neural network to predict mutations in proteins from influenza A viruses - A review of our approaches with implication for predicting mutations in coronaviruses // Journal of Physics: Conference Series.-2020.- Vol. 1682, No. 1.
- Salama M.A., Hassanien A.E., Mostafa A. The prediction of virus mutation using neural networks and rough set techniques // Eurasip Journal on Bioinformatics and Systems Biology.-2016.- Vol. 2016, No. 1.- P.1-11.
- Sawmya S., Haisam A., Rafid M. Analyzing hCov Genome Sequences : Predicting Virulence and Mutation // bioRxiv. -2021.- P.1-14.
- Hochreiter S., Schmidhuber J. Long Short-Term Memory // Neural Computation.-1997.- Vol. 9, No. 8.- P.1735-1780.
- Yin R., Luusua E., Dabrowski J., et al. Tempel: Time-series mutation prediction of influenza A viruses via attention-based recurrent neural networks // Bioinformatics.-2020.-Vol. 36, No. 9.- P.2697-2704.
- Hossain M.S., Pathan A.Q.M.S.U., Islam M.N., et al. Genome-wide identification and prediction of SARS-CoV-2 mutations show an abundance of variants: Integrated study of bioinformatics and deep neural learning. // bioRxiv.-2021.
- Hie B., Zhong E.D., Berger B., et al. Learning the language of viral evolution and escape // Science.-2021.- Vol. 371.- P.284-288.
- Asgari E., Mofrad M.R.K. Continuous distributed representation of biological sequences for deep proteomics and genomics // PLoS 0NE.-2015.- Vol. 10, No. 11.- P.1-15.
- Elbe S., Buckland-Merrett G. Data, disease and diplomacy: GISAID's innovative contribution to global health // Global Challenges.-2017.- Vol. 1, No. 1.- P.33-46.
- Okada P., Buathong R., Phuygun S., et al. Early transmission patterns of coronavirus disease 2019 (COVID-19) in travellers from Wuhan to Thailand, January 2020 // Eurosurveillance.-2020.- Vol. 25, No. 8.
- Cock P.J.A., Antao T., Chang J.T., et al. Biopython: Freely available Python tools for computational molecular biology and bioinformatics // Bioinformatics.-2009.- Vol. 25, No. 11.- P.1422-1423.
- Okonechnikov K., Golosova O., Fursov M., et al. Unipro UGENE: A unified bioinformatics toolkit // Bioinformatics.-2012.- Vol. 28, No. 8.- P.1166-1167.
- Edgar R.C. MUSCLE: Multiple sequence alignment with high accuracy and high throughput // Nucleic Acids Research.-2004.- Vol. 32, No. 5.- P.1792-1797.
- Yan S., Wu G. Prediction of Mutation Positions in H5N1 Neuraminidases From Influenza A Virus by Means of Neural Network // Annals of Biomedical Engineering.-2010.- Vol. 38, No. 3.- P.984-992.
- Waterhouse A., Bertoni M., Bienert S., et al. SWISS-MODEL: Homology modelling of protein structures and complexes // Nucleic Acids Research.-2018.- Vol. 46, No. 1.-P.296-303.
- Bienert S., Waterhouse A., De Beer T.A.P., et al. The SWISS-MODEL Repository-new features and functionality // Nucleic Acids Research.-2017.- Vol. 45, No. 1.- P.313-319.
- Guex N., Peitsch M.C., Schwede T. Automated comparative protein structure modeling with SWISS-MODEL and Swiss-PdbViewer: A historical perspective // Electrophoresis.-2009.- Vol. 30, No.1.- P.162-173.
- Studer G., Rempfer C., Waterhouse A.M., et al. QMEANDisCo—distance constraints applied on model quality estimation // Bioinformatics.-2020.- Vol. 36, No. 6.- P.1765-1771.
- Bertoni M., Kiefer F., Biasini M., et al. Modeling protein quaternary structure of homo- and hetero-oligomers beyond binary interactions by homology // Scientific Reports.-2017.- Vol. 7, No. 1.- P.1-15.
- Berman H.M. The Protein Data Bank // Nucleic Acids Research.-2000.- Vol. 28, No. 1.- P.235-242.
- Desta I.T., Porter K.A., Xia B., et al. Performance and Its Limits in Rigid Body Protein-Protein Docking // Structure.-2020.- Vol. 28, No. 9.- P.1071-1081.
- Vajda S., Yueh C., Beglov D., et al. New additions to the ClusPro server motivated by CAPRI // Proteins: Structure, Function and Bioinformatics.-2017.- Vol. 85, No. 3.-P.435-444.
- Kozakov D., Hall D.R., Xia B., et al. The ClusPro web server for protein-protein docking // Nature Protocols.-2017.- Vol. 12, No. 2.- P.255-278.
- Kozakov D., Beglov D., Bohnuud T., et al. How good is automated protein docking? // Proteins: Structure, Function, and Bioinformatics.-2013.- Vol. 81, No. 12.- P.2159-2166.
- Vangone A., Bonvin A.M.J.J. Contacts-based prediction of binding affinity in protein-protein complexes // eLife.-2015.- Vol. 4.- P.1-15.
- Xue L.C., Rodrigues J.P., Kastritis P.L., et al. PRODIGY: A web server for predicting the binding affinity of protein-protein complexes // Bioinformatics.-2016.- Vol. 32, No. 23.-P.3676-3678.
- Schrödinger, LLC The {PyMol} Molecular Graphics System, Version~1.8 // -2015.-.
- Klink G. V, Safina K.R., Garushyants S.K., et al. Spread of endemic SARS-CoV-2 lineages in Russia // medRxiv.-2021.
- Jangra S., Ye C., Rathnasinghe R., et al. SARS-CoV-2 spike E484K mutation reduces antibody neutralisation // The Lancet Microbe.-2021.- Vol. 2, No. 7.- P.283-284.
- Akkiz H. Implications of the Novel Mutations in the SARS-CoV-2 Genome for Transmission, Disease Severity, and the Vaccine Development. // Frontiers in medicine.-2021.-Vol. 8.
- Nelson T.L., Fosdick B.K., Biela L.M., et al. Association Between COVID-19 Exposure and Self-reported Compliance With Public Health Guidelines Among Essential Employees at an Institution of Higher Education in the US // JAMA Network Open.-2021.- Vol. 4, No. 7.
- O'Toole Â., Scher E., Underwood A., et al. Assignment of epidemiological lineages in an emerging pandemic using the pangolin tool // Virus Evolution.-2021.-Vol. 7, No. 2.
- Gladkikh A., Dolgova A., Dedkov V., et al. Characterization of a Novel SARS-CoV-2 Genetic Variant with Distinct Spike Protein Mutations // Viruses -2021.- Vol. 13, No. 6.
- Planas D., Veyer D., Baidaliuk A., et al. Reduced sensitivity of SARS-CoV-2 variant Delta to antibody neutralization // Nature.-2021.- Vol. 596, No.- P.276-280.
- Wall E.C., Wu M., Harvey R., et al. Neutralising antibody activity against SARS-CoV-2 VOCs B.1.617.2 and B.1.351 by BNT162b2 vaccination // The Lancet.-2021.- Vol. 397.-P.2331-2333.
- Winger A., Caspari T. The Spike of Concern—The Novel Variants of SARS-CoV-2 // Viruses.-2021.- Vol. 13, No. 6.
- Osina N.A., Krasnov Y.M., Guseva N.P., et al. Molecular-genetic monitoring of sARs-CoV-2 genovariants in the territory of the volga federal district of the Russian Federation. Communication // Problemy Osobo Opasnykh Infektsii.-2021.- Vol. 2, No. 1.- P.122-127.
- Yi Y., Lagniton P.N.P., Ye S., et al. COVID-19: what has been learned and to be learned about the novel coronavirus disease // International journal of biological sciences.-2020.-Vol. 16, No. 10.- P. 1753-1766.
- Korber B., Fischer W.M., Gnanakaran S., et al. Tracking Changes in SARS-CoV-2 Spike: Evidence that D614G Increases Infectivity of the COVID-19 Virus // Cell.-2020.- Vol. 182, No. 4.- P.812-827.


