Разработка схемы ПЦР-ПДРФ-генотипирования BLV в соответствии с филогенетической классификацией
Автор: Шаева А.Ю., Вафин Р.Р., Хазипов Н.З., Камалов Б.В., Алимов А.М., Тагиров М.Ш.
Статья в выпуске: 1 т.205, 2011 года.
Бесплатный доступ
Целью данной работы являлся сравнительный анализ представителей ВЛКРС на предмет их классификации в контексте предложенных стратегий типизации с дальнейшим усовершенствованием схемы генотипической идентификации BLV по локусу env-гена возбудителя. Подобранные нами условия проведения ПЦР-ПДРФ-анализа с использованием 5 ферментов BamHI, PvuII, DdeI, SspI и AsuHPI позволяют идентифицировать генотипы ВЛКРС в соответствии с филогенетической классификацией BLV.
Влкрс, классификация, генотипирование, пцр, пдрф
Короткий адрес: https://sciup.org/14287146
IDR: 14287146 | УДК: 619:57.065:578.828
Текст научной статьи Разработка схемы ПЦР-ПДРФ-генотипирования BLV в соответствии с филогенетической классификацией
Лейкоз крупного рогатого скота , имеющий повсеместное широкое распространение , остается одной из наиболее актуальных проблем животноводства в виду нанесения данной отрасли сельского хозяйства ощутимого экономического ущерба [1, 2, 3, 4, 5].
Лабораторно - диагностические исследования в системе мониторинга инфицированности стад Bovine Leukemia virus (BLV) являются неразрывным звеном противолейкозных мероприятий [4, 5].
Ранее разработанные способы типизации ВЛКРС на основе ПЦР - ПДРФ - анализа [1, 2, 3], несмотря на определенную идентификационную ценность , всё же не позволяют охарактеризовать весь спектр генетического многообразия BLV, нежели филогенетическая классификация , предложенная S.M. Rodriguez et al. (2009) [5], и регламентирующая наличие 7 генотипов у данного инфекционного агента .
Целью данной работы являлся сравнительный анализ представителей ВЛКРС на предмет их классификации в контексте предложенных стратегий типизации с дальнейшим усовершенствованием схемы генотипической идентификации BLV по локусу env-гена возбудителя.
Материалы и методы . В работе были использованы 2 изолята ВЛКРС : «N10» и «N28», выделенные от кр . рог . ск . из Мензелинского и Спасского районов Республики Татарстан , соответственно .
При постановке ПЦР с выделенными образцами провирусной ДНК BLV применялась модификация «nested» ПЦР , генерирующая амплификацию фрагмента env - гена ВЛКРС длиной 444 п . н . [1, 2, 3].
Эндонуклеазы рестрикции , использованные для ПЦР - ПДРФ - анализа : Ksp22I , HaeIII , BamHI , PvuII, DdeI, SspI и AsuHPI .
ПЦР - ПДРФ - фрагменты разгоняли в 2,5% агарозном геле (20 В / см , 40 мин ) и визуализировали в УФ - трансиллюминаторе ( λ =310 нм ).
ПЦР - ПДРФ - моделирование : NEBcutter v.2.0.
Секвенирование продуктов амплификации локуса env - гена BLV с внутренними праймерами «env5099» и «env5521» изолятов «N10» и «N28» ВЛКРС выполнено на приборе ABI-300 в лабораториях НПО « СибЭнзим ».
Изоляты «N10» (GenBank A/N: HM102355) и «N28» (GenBank A/N: HM102356) были выравнены по длине 444 нуклеотидов локуса env - гена c соответствующими опубликованными в GenBank последовательностями представителей известных генотипов BLV, используя программы BLAST и CLUSTAL W (v. 1.83) с последующим филогенетическим анализом .
Результаты исследования и их обсуждение . В результате исследования 2- х выделенных нами изолятов ВЛКРС на предмет их таксономической принадлежности , установлено , что в зависимости от выбранной стратегии типизации на основе ПЦР - ПДРФ - анализа , изолят «N10» BLV относится к Бельгийскому подтипу ( по D. Beier et al. 2001, [1]), к 6- му генотипу ( по M. Licursi et al. (2002) [3]), и к группе «A» ( по H Fechner, 1997 [2]); а изолят «N28» BLV характеризуется признаком Австралийского подтипа ( по D. Beier et al. 2001, [1]), 1- го генотипа ( по M. Licursi et al. (2002) [3]), и группы « С » ( по H Fechner, 1997 [2]). По филогенетической же классификации , предложенной S.M. Rodriguez (2009) [5], изоляты «N10» и «N28» принадлежат к кластерам 4- го и 7- го генотипов , соответственно .
При сравнительном анализе 184 заявленных в GenBank NCBI нуклеотидных последовательностей изолятов BLV на предмет их классификации в зависимости от выбранной стратегии идентификации установлено , что известные ранее способы типизации представителей BLV, разработанные на основе ПЦР - ПДРФ - анализа [1 2, 3], не согласуются с новым подходом оценки генотипического разнообразия ВЛКРС на основе филогенетического анализа env - гена [4, 5] ( табл . 1).
-
1. Классификация заявленных в GenBank NCBI изолятов BLV в зависимости от выбранной стратегии типизации ( количественное отношение )
ТИПИЗАЦИЯ
ФИЛОГЕНЕТИЧЕСКИЙ АНАЛИЗ
ГЕНОТИПЫ
1- ый
2- ой
3- ий
4- ый
5- ый
6- ой
7- ой
! ё в В
в -н ^
4
0 в
Бельгийский
17
Австралийский
49
4
8
4
Японский
8
2
?
42
37
1
10
2
Н m
0 “
В ы ^
1- ый
87
1
7
4
2- ой
3- ий
8
2
1
4- ый
5- ый
1
4
1
6- ой
36
17
8
?
3
1
2
1
Рн b
A
17
B
8
2
1
C
48
4
8
4
D
39
1
E
F
36
8
G
1
?
3
1
2
1
Дальнейшие исследования были направлены на разработку схемы ПЦР - ПДРФ - генотипирования ВЛКРС , согласующейся с филогенетической классификацией , предложенной S.M. Rodriguez (2009) [5], для чего , на первом этапе , провели скрининг известных эндонуклеаз рестрикций путем моделирования ПЦР - ПДРФ - профилей нуклеотидных последовательностей локуса env - гена представителей известных генотипов BLV с последующим формированием оптимального протокола идентификации .
-
2. Env - ПЦР - ПДРФ - профили BLV ( праймеры env5099-env5521)
Г
Типовой изолят
GenBank A/N
ПЦР - продукт ( п . н .)
ПДРФ - фрагменты ( п . н .)
К
N
BamHI
PvuII
DdeI
SspI
HphI ( AsuHPI )
1
AL-63
FJ808571
444
316/128
444
444
399/45
224/220
1
57
1
AL-2106
FJ808578
444
444
444
444
399/45
224/220
2
37
1
Uru38
FM209471
444
444
444
330/114
399/45
224/220
3
3
1
VdM
M35239
444
316/128
444
444
399/45
224/181/39
4
1
1
Kurdista n
EU266062
444
316/128
444
444
399/45
220/196/28
5
1
2
AL-164
FJ808574
444
316/128
280/164
276/168
399/45
224/220
6
35
2
ARGSF8
AF485773
444
316/128
280/164
276/168
399/45
444
7
1
2
AL-1453
FJ808577
444
316/128
280/164
276/168
444
224/220
8
1
3
JPFU
EF065650
444
316/128
444
276/168
399/45
444
9
4
4
BG
EF065638
444
444
280/164
276/168
399/45
224/220
10
16
4
N10
HM102355
444
444
280/164
168/162/114
399/45
224/220
11
1
4
3
U87872
444
444
444
168/162/114
399/45
224/220
12
1
5
CRAS-2
EF065636
444
316/128
280/164
276/168
399/45
224/181/39
13
8
5
CRLC-1
EF065655
444
316/128
280/164
276/168
444
224/181/39
14
2
6
PL-1238
FJ808582
444
316/128
444
276/168
399/45
224/220
15
10
7
N28
HM102356
444
316/128
444
276/168
444
224/137/83
16
3
7
I2
S83530
444
316/128
444
276/168
444
224/220
17
1
7
14
AY515274
444
316/128
444
276/168
444
145/137/83/ 79
18
1
7
30
DQ059417
444
316/128
444
276/168
444
444
19
1
Обобщенная информация с интерпретацией полученных env - ПЦР - ПДРФ - профилей ВЛКРС представлена в табл . 2.
Установлено , что для выделенных нами изолятов характерны соответствующие комбинации ПЦР - ПДРФ - профилей : для изолята «N10» – комбинация № 11; для изолята «N28» – комбинация № 16 ( табл . 2).
Следует подчеркнуть , что рассчитанная в результате секвенирования продуктов ПЦР - амплификации точная картина ожидаемых ПДРФ - фрагментов ДНК изолятов «N10» и «N28» ВЛКРС , в дальнейшем , была воспроизведена нами экспериментально .
Обозначения: Г – генотип; К – комбинация; N – количество известных изолятов BLV с соответствующей комбинацией ПЦР-ПДРФ-профилей.
Распределение фрагментов рестрикции , полученных при эндонуклеазном расщеплении ПЦР - продуктов ферментами BamHI, PvuII , DdeI , SspI и AsuHPI, представлено на рис . 1.
На рис . 1 видно , что при обработке ПЦР - продукта от изолята «N10» ферментом BamHI ПДРФ - фрагментов не образуется ввиду отсутствия соответствующего сайта рестрикции в анализируемом локусе env - гена . PvuII расщепляет ампликон на 2 фрагмента – 280 и 164 п . н ., DdeI – на фрагменты 168, 162 и 114 п . н ., SspI – 399 и 45 п . н ., AsuHPI – 224 и 220 п . н .
Полученный ампликон от изолята «N28» расщепляется эндонуклеазой рестрикции BamHI на фрагменты длиной 316 и 128 п . н ., DdeI – 276 и 168 п . н ., а рестриктазой AsuHPI на 224, 137 и 83 п . н .. Ферменты PvuII и SspI в данном локусе env - гена изолята «N28» ПДРФ - фрагментов не образуют из - за отсутствия соответствующих сайтов рестрикции ( рис . 1).
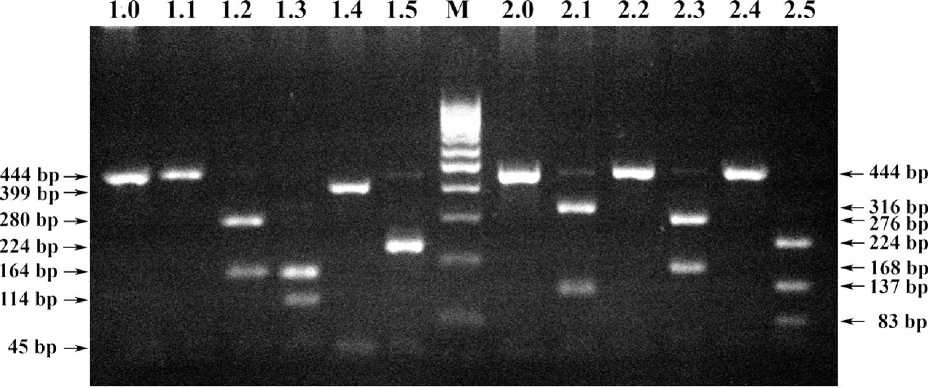
1. Электрофореграмма env - ПЦР - ПДРФ - профилей изолятов N10 и N28 BLV Обозначения : 1.0-1.5) ПЦР - ПДРФ - профиль изолята N10 BLV: 1.0) ПЦР - продукт (444 bp); 1.1) BamHI - ПДРФ (444 bp); 1.2) PvuII - ПДРФ (280/164 bp); 1.3) DdeI - ПДРФ (168/162/114 bp); 1.4) SspI - ПДРФ (399/45 bp); 1.5) AsuHPI - ПДРФ (224/220 bp). М ) ДНК - маркеры 100-1000 bp ( СибЭнзим ). 2.0-2.5) ПЦР - ПДРФ - профиль изолята N28 BLV: 2.0) ПЦР - продукт (444 bp): 2.1) BamHI - ПДРФ (316/128 bp); 2.2) PvuII - ПДРФ (444 bp); 2.3) DdeI - ПДРФ (276/168 bp); 2.4) SspI - ПДРФ (444 bp); 2.5) AsuHPI - ПДРФ (224/137/83 bp).
Заключение . Подобранные нами условия проведения ПЦР - ПДРФ - анализа с использованием 5 ферментов BamHI, PvuII , DdeI , SspI и AsuHPI позволяют идентифицировать генотипы ВЛКРС в соответствии с филогенетической классификацией BLV [4. 5].
Следует отметить , что 1- ый генотип ВЛКРС характеризуется наличием пяти комбинаций ПЦР - ПДРФ профилей ( К 1-5); 2- ой генотип – трех комбинаций ( К 6-8), 3- ий генотип – наличием одной комбинации ( К 9); 4- ый генотип – трех комбинаций ( К 10-12); 5- ый генотип – двух комбинаций ( К 13-14); 6- ой генотип – одной комбинации ( К 15), и 7- ой генотип – наличием четырех комбинаций ( К 16-19) ( табл . 2).
Примечательно , что для идентификации 1- го генотипа достаточно даже одной эндонуклеазы рестрикции – DdeI . С использованием двух ферментов могут быть определены представители 4- го ( BamHI и DdeI ), 5 го ( PvuII и AsuHPI ) и 7- го генотипов ( PvuII и SspI ) BLV ( табл . 2).
В целях же исчерпывающей идентификации генотипов ВЛКРС целесообразно руководствоваться полной картиной env - ПЦР - ПДРФ - профилей с сопоставлением полученных данных с результатами секвенирования ПЦР - продуктов и последующим филогенетическим анализом представителей BLV по env - гену данного возбудителя .
ЛИТЕРАТУРА : 1. Beier, D. Identification of different BLV provirus isolates by PCR, RFLPA and DNA sequencing / D. Beier, P. Blankenstein, O.
Marquardt, J. Kuzmak // Berl Munch Tierarztl Wochenschr – 2001. – V. 114. – № 7-8. – P. 252-256. 2. Fechner, H. Provirus variants of the bovine leukemia virus and their relation to the serological status of naturally infected cattle / H. Fechner, P. Blankenstein, A.C. Looman, J. Elwert, L. Geue, C. Albrecht, A. Kurg, D. Beier, O. Marquardt, D. Ebner // Virology –1997. – V. 237. – № 2. – P. 261-269. 3. Licursi, M. Genetic heterogeneity among bovine leukemia virus genotypes and its relation to humoral responses in hosts / M. Licursi, Y. Inoshima, D. Wu, T. Yokoyama, E.T. González, H. Sentsui // Virus Res. – 2002. – V. 86. – № 1-2. – P. 101-110. 4. Moratorio, G. Phylogenetic analysis of bovine leukemia viruses isolated in South America reveals diversification in seven distinct genotypes / G. Moratorio, G. Obal, A. Dubra, A. Correa, S. Bianchi, A. Buschiazzo, J. Cristina, O. Pritsch // Arch. Virol. – 2010. – V. 155. – № 4. – P. 481-489. 5. Rodriguez, S.M. Bovine leukemia virus can be classified into seven genotypes: evidence for the existence of two novel clades / S.M. Rodriguez, M.D. Golemba, R.H. Campos, K. Trono, L.R. Jones //J Gen Virol. – 2009. – V. 90. – № 11. – P. 2788-2797.
РАЗРАБОТКА СХЕМЫ ПЦР - ПДРФ - ГЕНОТИПИРОВАНИЯ BLV В СООТВЕТСТВИИ С ФИЛОГЕНЕТИЧЕСКОЙ КЛАССИФИКАЦИЕЙ
Шаева А . Ю ., Вафин Р . Р ., Хазипов Н . З ., Камалов Б . В ., Алимов А . М ., Тагиров М . Ш .
Резюме
Целью данной работы являлся сравнительный анализ представителей ВЛКРС на предмет их классификации в контексте предложенных стратегий типизации с дальнейшим усовершенствованием схемы генотипической идентификации BLV по локусу env - гена возбудителя . Подобранные нами условия проведения ПЦР - ПДРФ - анализа с использованием 5 ферментов BamHI, PvuII , DdeI , SspI и AsuHPI позволяют идентифицировать генотипы ВЛКРС в соответствии с филогенетической классификацией BLV.
DEVELOPMENT OF THE SCHEME OF PCR-RFLP-GENOTYPING BLV IN ACCORDANCE WITH PHYLOGENETIC CLASSIFICATION
Shaeva A.Y., Vafin R.R., Khazipov N.Z., Kamalov B.V., Alimov A.M., Tagirov M.Sh.


