Результаты анализа состава микрофлоры рубца коров под влиянием пробиотика методом NGS-секвенирования
Автор: Йылдырым Елена Александровна, Лаптев Георгий Юрьевич, Дубровина Елена Германовна, Ильина Лариса Александровна, Тюрина Дарья Георгиевна, Филиппова Валентина Анатольевна, Дубровин Андрей Валерьевич, Калиткина Ксения Андреевна, Скляров Сергей Павлович
Журнал: Вестник Красноярского государственного аграрного университета @vestnik-kgau
Рубрика: Зоотехния и ветеринария
Статья в выпуске: 1, 2024 года.
Бесплатный доступ
Цель исследования - изучение влияния пробиотика «Профорт-Т» на состав микробиома рубца дойных коров методом NGS-секвенирования, продуктивность и гематологические показатели сыворотки крови животных. Эксперимент проводили на коровах черно-пестрой породы в течение 60 сут. Были сформированы группы-аналоги по 20 голов каждая: контрольная I (получавшая основной рацион (ОР)) и опытная II (получавшая ОР и пробиотик «Профорт-Т»). ОР состоял: из силоса - 42,26 кг; сена - 1,97; комбикорма - 14,67 кг; соли, мела, соды. Коровы опытной группы II получали пробиотик «Профорт-Т» из расчета 0,03 кг на 1 голову со дня поступления в новотельную группу. Кормовая добавка раздавалась вручную каждой голове опытной группы II в новотельный период и частично в раздой. Животные находились в одинаковых условиях содержания. Содержание животных - привязное. Бактериальное сообщество рубца оценивали методом NGS-секвенирования на секвенаторе MiSeq (Illumina, Inc., США). Применение пробиотика (опытная группа II) позволило увеличить среднесуточные надои от 14,9 до 22,0 % по сравнению с контролем (P ≤ 0,05) и снизить содержание соматических клеток в молоке коров от 21,6 до 33,5 % (P ≤ 0,05). Проведение NGS-секвенирования продемонстрировало, что в составе микрофлоры рубца исследованных коров был обнаружен 21 филум микроорганизмов. Наиболее представленными (P ≤ 0,05) в рубце были бактерии филума Bacteroidetes - от 46,6 ± 4,48 до 58,1 ± 5,52 %. Доля целлюлозолитических бактерий Candidatus Saccharibacteria увеличивалась в опытной группе II в конце эксперимента по сравнению с контролем в 1,9 раза (P ≤ 0,05). Микроорганизмы Proteobacteria и Fusobacteria снижались в опытной группе II по сравнению с контролем в 5,7 и 1,8 раза соответственно (P ≤ 0,05), т. е. применение пробиотика «Профорт-Т» привело к улучшению структуры микробного сообщества рубца коров.
Микробиом, рубец, ngs-секвенирование, профорт-т, высокопродуктивные коровы
Короткий адрес: https://sciup.org/140304288
IDR: 140304288 | УДК: 636 | DOI: 10.36718/1819-4036-2024-1-108-118
Текст научной статьи Результаты анализа состава микрофлоры рубца коров под влиянием пробиотика методом NGS-секвенирования
Введение. Жвачные животные не продуцируют целлюлолитические ферменты самостоятельно, полностью «полагаясь» на деятельность микроорганизмов рубца по расщеплению сложных растительных полисахаридов [1]. Микробиом рубца – это самая разнообразная экосистема пищеварительной системы животных, состоящая из бактерий, простейших, грибов, архей и фагов [2]. Основными продуктами ферментации рубца являются короткоцепочечные летучие жирные кислоты (ЛЖК) (преимущественно ацетат, пропионат и бутират) и микробный белок. ЛЖК служат важным источником энергии, обеспечивая до 70–80 % энергетических потребностей организма хозяина [3].
Ранее были предприняты масштабные исследования для изучения состава микробного сообщества рубца и его динамики с учетом таких параметров, как рацион кормления, возраст и генетика хозяина с использованием культуральных методов [4]. Достижения в области высокопроизводительных технологий секвенирования, не зависящих от культивирования, значительно расширили возможности изучения микробиома рубца, позволив более качественно анализировать структуру и функции его экосистемы.
Рацион кормления был признан одним из основных факторов, влияющих на активность микроорганизмов в рубце [5], которые, в свою очередь, влияют на продуктивность хозяина. Кормовые добавки, такие как пробиотики, все чаще используются для улучшения здоровья жвачных животных. Пробиотики участвуют в переваривании кормов и секреции органических кислот и других метаболитов, которые регулируют состав микробного сообщество рубца [6], что приводит к повышению продуктивности животных [7]. Несмотря на масштабные исследования данного вопроса, в настоящее время значительный прогресс в управлении микробиомами рубца у молочных коров для улучшения состояния здоровья и продуктивности пока не достигнут. Поэтому поиск новых натуральных биологических способов коррекции микробных сообществ рубца коров представляется актуальным.
Цель исследования – изучение влияния пробиотика «Профорт-Т» на состав микробиома рубца дойных коров методом NGS-секвенирования, продуктивность и гематологические показатели сыворотки крови животных.
Объекты и методы. Эксперимент проводили в коммерческом хозяйстве под условным номером «1» Кировской области. Коровы чернопестрой породы 2-й и 3-й лактации со средней продуктивностью 12 215 кг и живой массой 665 кг получали пробиотик «Профорт-Т» в течение 60 сут. Были сформированы группы-аналоги: контрольная I (получавшая ОР) и опытная II (получавшая ОР и «Профорт-Т») по 20 животных в каждой группе. Рационы коров были рассчитаны автоматически с использованием программы (URL: в соответствии с общепринятыми требованиями. ОР состоял: из силоса – 42,26 кг; сена – 1,97; комбикорма – 14,67 кг; соли, мела, соды. Коровы опытной группы II получали пробиотик «Профорт-Т» из расчета 0,03 кг на 1 голову со дня поступления в новотельную группу. Кормовая добавка раздавалась вручную каждой голове опытной группы II в новотельный период и частично в раздой. Животные находились в одинаковых условиях содержания. Содержание животных – привязное.
Пробиотик «Профорт-Т» содержит в составе 2 штамма полезных пробиотических бактерий Bacillus sp ., которые обуславливают его действие – нормализацию микрофлоры желудочнокишечного тракта сельскохозяйственных животных и подавление жизнедеятельности патогенных и условно-патогенных микроорганизмов. «Профорт-Т» имеет выраженное антагонистическое действие в отношении патогенных микроорганизмов, что отмечено в серии опытов in vitro (согласно ОФС.1.7.2.0009.15 «Определение специфической активности пробиотиков»). Помимо этого штаммы бактерий в составе пробиотика способствуют повышению переваримости и усвояемости питательных веществ, входящих в состав кормов.
Среднесуточный удой коров оценивали ежемесячно методом контрольных доек. Количество соматических клеток в молоке оценивали ежемесячно по ГОСТ Р 54761-201.
Отбор проб крови у животных осуществляли (в трех повторностях) до начала эксперимента (в новотельный период) и в конце эксперимента (в период раздоя) в утреннее время до начала кормления (натощак) и проводили гематологический анализ по стандартным методикам [8].
Отбор проб содержимого рубца коров (в трех повторностях) проводили до начала эксперимента (в новотельный период) и в конце эксперимента (в период раздоя). Отбор проб химуса рубца (30–50 г) проводили из верхней части вентрального мешка рубца коров с максимально возможным при данном методе соблюдением условий асептики вручную с использованием стерильного зонда. Отобранные образцы немедленно помещали в стерильные пластиковые пробирки. Все образцы замораживали при -20 ° С и транспортировали в сухом льду в лабораторию для последующего выделения ДНК.
Тотальную ДНК из образцов выделяли с использованием набора Genomic DNA Purification Kit (Thermo Fisher Scientific, Inc., США) согласно прилагаемой инструкции. Бактериальное сообщество оценивали методом NGS- секвенирования, на автоматическом секвенаторе MiSeq (Illumina, Inc., США) с применением праймеров для V3-V4 региона 16S рРНК: 5´- TCGTCGG-CAGCGTCAGATGTGTATAAGAGACAGCCTACG GGNGGCWGCAG- 3´ (прямой праймер), 5´-GTCTCGTGGGCTCGGAGATGTGTATAAGAGAC AGGACTACHVGGGTATCTAATCC- 3´ (обратный праймер). Условия ПЦР были следующими: 3 мин при 95 ° С; 30 с при 95 ° С, 30 с при 55 ° С, 30 с при 72 ° С (необходимо для удлинения последовательности) (25 циклов); 5 мин при 72 ° С (окончательное удлинение). Секвенирование проводили при помощи реагентов для подготовки библиотек Nextera® XT IndexKit (Illumina, Inc., США), для очистки ПЦР-продуктов Agencourt AMPure XP (Beckman Coulter, Inc., США) и для проведения секвенирования MiSeq® ReagentKit v2 (500 cycle) (Illumina, Inc., США). Максимальная длина полученных последовательностей составила 2 × 250 п.н.
Автоматический биоинформатический анализ данных выполняли с помощью программного обеспечения QIIME2 ver. 2020.8 (URL: . После импорта последовательностей в формате .fastq из сек-венирующего прибора и создания необходимых для работы файлов сопоставления (содержащих метаданные изучаемых файлов) парные строки прочтений были выровнены. Далее по- следовательности фильтровали по качеству с использованием параметров настроек по умолчанию. Фильтрацию шумовых последовательностей проводили с помощью встроенного в программное обеспечение QIIME2 пакета DADA2, включающего информацию о качестве последовательностей в свою модель ошибок (фильтрацию химерных последовательностей, артефактов, адаптеров), что делает алгоритм устойчивым к последовательности более низкого качества. При этом использовали максимальную длину последовательности обрезки, равную 250 п.н. (URL: . Для построения филогении de novo выполнили множественное выравнивание последовательностей, применяя программный пакет MAFFT, далее проводили маскированное выравнивание, чтобы удалить позиции, которые значительно различались. Для назначения таксономии использовали программное обеспечение QIIME2, которое присваивает последовательности таксономическую идентификацию на основе данных ASV (методами BLAST, RDP, RTAX, mothur и uclust), используя базу данных по 16s rRNA Silva 138.1 (URL: documentation/release-138.1).
Математическую и статистическую обработку результатов осуществляли методом многофакторного дисперсионного анализа (multifactor ANalysis Of VAriance, ANOVA) в программах MS Excel XP/2003, R-Studio (Version 1.1.453) (URL: . Достоверность различий устанавливали по t-критерию Стьюдента, различия считали статистически значимыми при Р ≤ 0,05. Средние значения сравнивали с использованием теста достоверно значимой разницы Тьюки (HSD) и функции TukeyHSD в пакете R Stats Package.
Результаты и их обсуждение. Результаты исследования показали, что применение пробиотика «Профорт-Т» (опытная группа II) на поголовье крупного рогатого скота позволило увеличить среднесуточные надои от 14,9 до 22,0 % по сравнению с контролем I (P ≤ 0,05) и снизить содержание соматических клеток в молоке коров от 21,6 до 33,5 % (P ≤ 0,05) (табл. 1).
Среднесуточный удой и содержание соматических клеток в молоке коров черно-пестрой породы под влиянием пробиотика «Профорт-Т»
Таблица 1
|
Показатель |
1 мес. после начала эксперимента |
В конце эксперимента |
||
|
Контроль I |
Опыт II |
Контроль I |
Опыт II |
|
|
Среднесуточный удой, кг |
39,0±1,86 |
44,8±2,35* |
35,5±1,59 |
43,3±2,10* |
|
Соматические клетки в молоке, тыс/см3 |
639,8±30,57 |
425,5±26,80* |
463,2±32,51 |
363,2±40,15* |
*P ≤ 0,05 при сравнении опытной группы II с контролем I согласно критерию Стьюдента.
Как видно из таблицы 2, применение пробиотика «Профорт-Т» не оказало значительного влияния на гематологические показатели сыворотки крови. Тем не менее, через 1 месяц использования биопрепарата отмечено снижение содержания мочевой кислоты и аспартатаминотрансферазы (АсАТ) в опытной группе II по сравнению с контрольной группой I (P ≤ 0,05).
Стоит отметить, что среднее значение АсАТ в контрольной группе I было приближено к верхней границе нормы. Это, вероятно, свидетельствует о позитивном влиянии пробиотика «Профорт-Т» на организм коров, поскольку увеличение уровня АсАТ у животных наблюдается при заболеваниях печени, кетозе, эндометрите и др. [9, 10].
Таблица 2
|
Показатели |
1 мес. после начала эксперимента |
В конце эксперимента |
Норма |
||
|
Контроль I |
Опыт II |
Контроль I |
Опыт II |
||
|
Гемоглобин, г/л |
85,5±1,55 |
84,0±1,29 |
88,5±2,96 |
89,3±2,87 |
90–120 |
|
Эритроциты, ×1012/л |
5,6±0,40 |
5,17±0,68 |
5,21±0,08 |
5,3±0,33 |
5–10 |
|
Лейкоциты, ×109/л |
9,6±1,24 |
8,2±2,71 |
10,6±1,61 |
10,7±2,75 |
4–12 |
|
Общий белок, г/л |
76,7±1,58 |
76,5±1,61 |
78,9±3,55 |
79,9±3,18 |
70–80 |
|
Альбумины, г/л |
37,6±0,57 |
38,4±0,43 |
37,9±0,46 |
39,1±0,73 |
25,8–39,7 |
|
Глобулины, г/л |
39,1±1,93 |
38,2±1,84 |
41,0±3,69 |
40,9±2,83 |
– |
|
А/Г |
0,97±0,06 |
1,01±0,06 |
0,95±0,10 |
0,97±0,06 |
0,8–1,2 |
|
Мочевина, ммоль/л |
4,3±0,56 |
5,3±0,87 |
4,3±0,25 |
5,3±0,39 |
3,1–9,2 |
|
Триглицериды, ммоль/л |
0,11±0,01 |
0,12±0,02 |
0,09±0,01 |
0,12±0,01 |
0–1,7 |
|
Глюкоза, ммоль/л |
2,3±0,16 |
2,2±0,19 |
2,9±0,16 |
2,8±0,12 |
3,4–6 |
|
Мочевая кислота, мкмоль/л |
125,5±1,71 |
111,5±5,66* |
139,5±8,66 |
120,8±10,30 |
155–428 |
|
Общие иммуноглобулины, мг % |
213,7±21,54 |
202,5±13,62 |
202,9±24,78 |
201,9±22,80 |
– |
|
АлАТ, Ед/л |
20,4±1,71 |
23,4±0,93 |
25,3±1,56 |
32,3±2,29* |
8,2–57,3 |
|
АсАТ, Ед/л |
46,6±8,30 |
23,4±0,93* |
125,9±8,08 |
135,4±9,60 |
8,6–48,5 |
|
Щелочная фосфатаза, Ед/л |
88,9±9,42 |
95,5±10,32 |
129,4±29,37 |
150,3±38,97 |
55–80 |
*P ≤ 0,05 при сравнении опытной группы II с контролем I согласно критерию Стьюдента.
Гематологические показатели сыворотки крови дойных коров под влиянием пробиотика «Профорт-Т»
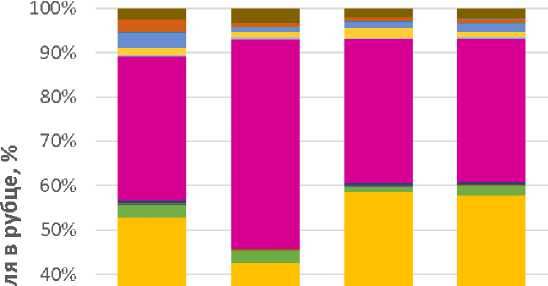
Проведение NGS-секвенирования рубца коров обеих групп показало, что в составе микрофлоры рубца исследованных коров до начала и в конце эксперимента в целом был обнаружен 21 филум микроорганизмов (рис. 1). Доминирующими были филумы Bacteroidetes и Firmicu- tes. Наиболее представленными (P ≤ 0,05) в рубце были бактерии филума Bacteroidetes – от 46,6 ± 4,48 до 58,1 ± 5,52 %. В исследованиях, опубликованных на сегодняшний день, Firmicu-tes и Bacteroidetes неизменно являются наиболее преобладающими филумами у жвачных жи- вотных, включая молочных коров [11, 12]. Так, в рамках проекта Hungate 1000 в 2012 г. было изучено 480 бактериальных геномов из различных образцов рубца. Исследования показали, что представители филумов Firmicutes и Bacte-roidetes преобладают, при этом на их долю приходится 68 и 12,8 % последовательностей генома соответственно [13]. Проведенный G. Hen- derson с соавторами [5] мониторинг рубцовых микробиомов, который охватывал 742 образца от 32 различных видов жвачных из разных географических регионов, показал, что 30 наиболее распространенных групп бактерий, включая Firmicutes и Bacteroidetes, были обнаружены почти во всех образцах, на долю приходилось 89,4 % всех последовательностей.
о
1:1 30%
20%
10%
0% ।
Контроль I Опыт II Контроль I Опыт II
До начала опыта В конце опыта
-
■ Неидентифицированные бактерии
-
■ Verrucomicrobia
-
■ Tenericutes
-
■ Synergistetes
-
■ SRI
-
■ Spirochaetes
Proteo bacteria
Planctomycetes
Lentisphaerae
-
■ Fuso bacteria
-
■ Firmicutes
-
■ Fibrobacteres
-
■ Euryarchaeota
-
■ Elusimicrobia
-
■ Cyanobacteria/Chloroplast
-
■ Chloroflexi
-
■ Candidatus_Saccharibacteria
-
■ BRC1
Bacteroidetes
-
■ Armatimonadetes
-
■ Actinobacteria
-
■ Acidobacteria
Рис. 1. Состав микрофлоры рубца коров черно-пестрой породы на уровне бактериальных филумов по данным NGS-секвенирования ампликонов гена 16S рРНК
Интересно, что доля бактерий филума Candi-datus Saccharibacteria увеличивалась в опытной группе II в конце эксперимента по сравнению с контролем I в 1,9 раза (P ≤ 0,05). При этом микроорганизмы из филумов Proteobacteria и Fusobacteria снижались в опытной группе II по сравнению с контролем I в 5,7 и 1,8 раза соответственно (P ≤ 0,05). Кроме того, содержание Fibrobacteres увеличивалось в 11,2 раза в опытной группе II при сравнении данных до начала эксперимента и после его окончания (P ≤ 0,05). Это может свидетельствовать о позитивном действии пробиотика «Профорт-Т» на регуляцию состава микробиома. Дело в том, что Candidatus Saccharibacteria являются важнейшей группой микроорганизмов в рубце, поскольку «отвечают» за переваривание целлюлозы [14]. Fibrobacteres также содержат несколько генов, кодирующих различные углеводноактивные ферменты (CAZymes), которые действуют синергически для разложения растительной лигноцеллюлозы [15]. Ведь все метаболические процессы в рубце начинаются с микробиологического разложения растительных полимеров на более мелкие растворимые сахара. Это дает огромное количество метаболитов, которые поступают в организм животного-хозяина и удовлетворяют до 70 % его энергетических потребностей [16]. Поскольку целлюлоза является наиболее распространенным углеводным полимером, на долю которого приходится до 40 % общего сухого вещества и более 50 % клеточной стенки растений, разложение целлюлозы является наиболее важным процессом для обеспечения энергией жвачных животных [17]. Proteobacteria же, в свою очередь, напро- тив, менее нежелательны в рубце, поскольку включают патогенов животных и человека, таких как Aeromonadaceae, в т. ч. роды Aeromonas и Enterobacteriaceae и Escherichia coli, содержание которых зависит от особенностей кормления [18]. Fusobacteria также принадлежат к нежелательным формам, поскольку на фоне высококонцентратного кормления они нередко получают конкурентное преимущество, ведь низкий уровень рН благоприятен для их развития [19]. Fusobacterium necrophorum, например, спо- собен вызывать целый спектр патологий у крупного рогатого скота, включая некробактериоз стенок рубца, ламиниты, абсцессы печени, маститы [20].
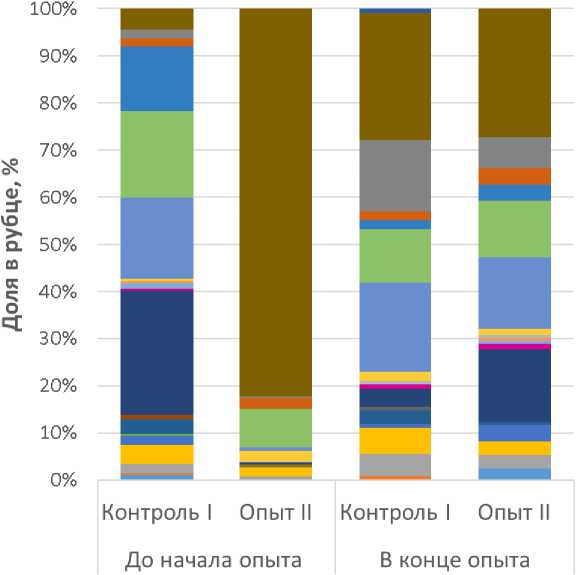
По результатам детального анализа микрофлоры содержимого рубца исследованных коров на уровне родов и видов удалось обнаружить изменения в содержании групп микроорганизмов, среди которых нередко встречаются патогенные и условно-патогенные формы (рис. 2).
-
■ Esherichia/Shigella
-
■ Bulleidia extructa
-
■ Holdemania massiliensis
-
■ Treponema pectinovorum
-
■ Treponema socranskii Treponema brennaborense
-
■ Treponema maltophilum Streptococcus sp.
Orientia tsutsugamushi Porphyromonas circumdentaria
-
■ Porphyromonas gingivicanis
-
■ Porphyromonas somerae
-
■ Porphyromonas cangingivalis
-
■ Peptococcus niger
-
■ Histophilus somni
-
■ Mycoplasma penetrans
-
■ Mycoplasma sp.
-
■ Clostridium intestinale
-
■ Clostridium innocuum Clostridium colinum
-
■ Catabacter hongkongensis
-
■ Campylobacter canadensis
-
■ Campylobacter sp.
Рис. 2. Состав условно-патогенной и патогенной микрофлоры рубца коров черно-пестрой породы на уровне бактериальных родов и видов по данным NGS-секвенирования ампликонов гена 16S рРНК
Стоит обратить внимание на то, что обнаруженные нежелательные микроорганизмы присутствовали в пробах, в основном в минорных концентрациях. Исключение составлял микроорганизм Bulleidia extructa. Его содержание в опытной группе II до начала эксперимента составляло 1,2 ± 0,08 %. Количество данной бактерии к концу эксперимента снижалось под влиянием пробиотика «Профорт-Т» (опытная группа II) в 8,7 раза по сравнению с изначальными значениями (P ≤ 0,05). Считается, что роль данного микроорганизма в процессах пато- генеза недооценена, поскольку Bulleidia ext-ructa – труднокультивируемый микроорганизм, для его детекции требуются такие методы идентификации, как секвенирование генов [21]. В последнее время признан патогенный потенциал B. extructa в качестве возбудителя заболеваний суставов, что крайне актуально и для крупного рогатого скота в условиях интенсивного ведения животноводства.
Также было выявлено снижение под воздействием пробиотика «Профорт-Т» таких бактерий, как Clostridium colinum, Catabacter hongkon- gensis, Holdemania massiliensis и Esherichia/ Shigella, которые могут участвовать в процессах патогенеза (P ≤ 0,05). Clostridium colinum способен вызывать клостридиозные энтериты [22]. Клинически клостридиоз, как правило, проявляется воспалениями желудочно-кишечного тракта, что влечет за собой появление крови и фибринозных тяжей в каловых массах. В отдельных случаях может отмечаться хромота, как следствие проникновения клостридий в суставы. Результаты экспериментов показали, что нарушение микробиоты пищеварительной системы может вызывать повреждение тканей и проникновение бактерий и их токсинов в кровоток [23]. Поэтому поддержание целостности барьера эпителия пищеварительной системы очень важно.
Полученные нами результаты, свидетельствующие о восстановлении состава микробиома рубца под влиянием пробиотика, перекликаются с данными предыдущих исследований. Так, ранее показано, что добавление пробиотика на основе Lactobacillus rhamnosus и Enterococcus faecalis в рацион коз способствовало доминированию в рубце полезных фибролитических или целлюлолитических бактерий [24]. Аналогичные данные были получены при применении пробиотика на поголовье крупного рогатого скота [25]. Применение пробиотика на основе Bacillus subtilis, Lactobacillus acidophilus и Saccharomy-ces cerevisiae на телятах позволило значительно повысить среднесуточный прирост живой массы в первые восемь недель после рождения при одновременном улучшении иммунной функции и снижении частоты диареи. Этот эффект был связан с увеличением относительного количества разрушающих клетчатку Ruminococ-caceae и бифидобактерий, которые играют важную роль в поддержке иммунитета [26].
Заключение. Результаты нашего исследования, полученные с помощью метода NGS-секвенирования, показали, что у коров в период раздоя возникают дисбиотические нарушения микрофлоры рубца, которые могут подвергнуть их высокому риску метаболических заболеваний. Применение пробиотика «Профорт-Т» привело к улучшению структуры микробного сообщества рубца, что способствовало увеличению продуктивности и снижению соматических клеток в молоке коров.
Список литературы Результаты анализа состава микрофлоры рубца коров под влиянием пробиотика методом NGS-секвенирования
- Characteristics and Functions of the Rumen Microbial Community of Cattle-Yak at Different Ages / Y. Sha [et al.] // Biomed Res Int. 2020; 3482692.
- Newbold C.J., Ramos-Morales E. Review: Ruminal Microbiome and Microbial Meta-bolome: Effects of Diet and Ruminant Host // Animal 2020; № 14. P. 78–86.
- Unravelling the Role of Rumen Microbial Communities, Genes, and Activities on Milk Fatty Acid Profile Using a Combination of Omics Approaches / S. Stergiadis [et al.] // Front Microbiol. 2021; 11:590441. DOI: 10.3389/fmicb.2020.
- Diversity and functional analysis of rumen and fecal microbial communities associated with dietary changes in crossbreed dairy cattle / F.M. Kibegwa [et al.] // PLoS One. 2023;18(1):e0274371. DOI: 10.1371/journal. pone.0274371.
- Rumen Microbial Community Composition Varies with Diet and Host, but a Core Microbiome Is Found across a Wide Geo-graphical Range / G. Henderson [et al.] // Sci. Rep. 2015; 5:14567.
- Selected Alternative Feed Additives Used to Manipulate the Rumen Microbiome / M. Micha-lak [et al.] // Animals. 2021; 11:1542.
- Mani S., Aiyegoro O.A., Adeleke M.A. Charac-terization of Rumen Microbiota of Two Sheep Breeds Supplemented with Direct-Fed Lactic Acid Bacteria // Front. Vet. Sci. 2021; 7:570074.
- Kondrakhin I.P. Methods of veterinary clinical laboratory diagnostics. M.: Kolos, 2004. 520 p.
- Waage S. Milk fever in the cow-course of di-sease in relation to the serum activity of aspar-tate aminotransferase, alanine aminotransfe-rase, creatine kinase and gamma-glutamyltran-sferase // Nord Vet Med. 1984; 36(9-10):282-95.
- Elevated Apoptosis in the Liver of Dairy Cows with Ketosis / X. Du [et al.] // Cell Physiol Biochem. 2017; № 43(2). P. 568–578. DOI: 10.1159/000480529.
- Temporal Changes in the Fecal Bacterial Community in Holstein Dairy Calves from Birth through the Transition to a Solid Diet / M.L. Hennessy [et al.] // PLoS ONE. 2020; 15:e0238882.
- Assessment of Rumen Bacteria in Dairy Cows with Varied Milk Protein Yield / M.Y. Xue [et al.] // J. Dairy Sci. 2019; 102:5031–5041.
- Cultivation and Sequencing of Rumen Microbiome Members from the Hungate1000 Collection / R. Seshadri [et al.] // Nat. Biotechnol. 2018. № 36. P. 359–367.
- Opdahl L.J., Gonda M.G., St-Pierre B. Identifi-cation of Uncultured Bacterial Species from Firmicutes, Bacteroidetes and CANDIDATUS Saccharibacteria as Candidate Cellulose Uti-lizers from the Rumen of Beef Cows // Mic-roorganisms. 2018. № 6(1). 17. DOI: 10.3390/ microorganisms6010017.
- Biotechnological Potential of Rumen Micro-biota for Sustainable Bioconversion of Lignocellulosic Waste to Biofuels and Value-Added Products / S.K. Bhujbal [et al.] // Sci. Total Environ. 2022; 814:152773.
- Gleason C.B., Beckett L.M., White R.R. Ru-men Fermentation and Epithelial Gene Ex-pression Responses to Diet Ingredients De-signed to Differ in Ruminally Degradable Pro-tein and Fiber Supplies // Sci. Rep. 2022. № 12. P. 29–33.
- Weimer P.J. Cellulose Degradation by Rumi-nal Microorganisms // Crit. Rev. Biotechnol. 1992. № 12. P. 189–223.
- Bäumler A.J., Sperandio V. Interactions be-tween the microbiota and pathogenic bacteria in the gut // Nature. 2016. Vol. 535. P. 85–93. DOI: 10.1038/nature18849.
- Fusobacterium necrophorum: a ruminal bacte-rium that invades liver to cause abscesses in cattle / S. Tadepalli [et al.] // Anaerobe. 2009. № 15(1-2). P. 36–43. DOI: 10.1016/j.anaero-be.2008.05.005.
- Shuttleworthia satelles gen. nov., sp. nov., isolated from the human oral cavity / J. Dow-nes [et al.] // International Journal of Systema-tic and Evolutionary Microbiology. 2002. № 52(5). P. 1469–1475. DOI: 10.1099/ 00207713-52-5-1469.
- Margie A. Morgan, Ellie J.C. Goldstein, Bullei-dia extructa: An underappreciated anaerobic pathogen // Anaerobe. 2021; 69:102339. DOI: 10.1016/j.anaerobe.2021.102339.
- Comparative pathogenesis of enteric clostridial infections in humans and animals / F.A. Uzal [et al.] // Anaerobe. 2018. № 53. P. 11–20. DOI: 10.1016/j.anaerobe.2018.06.002.
- Anders H.J., Andersen K., Stecher B. The in-testinal microbiota, a leaky gut, and abnormal immunity in kidney disease // Kidney. Int. 2013. № 83. P. 1010–1016. DOI: 10.1038/ ki.2012.440.
- Maake T.W., Aiyegoro O.A., Adeleke M.A. Effects of Lactobacillus Rhamnosus and En-terococcus Faecalis Supplementation as Di-rect-Fed Microbials on Rumen Microbiota of Boer and Speckled Goat Breeds // Vet. Sci. 2021; 8:103.
- The Effects of a Probiotic Yeast on the Bacte-rial Diversity and Population Structure in the Rumen of Cattle / E. Pinloche [et al.] // PLoS ONE. 2013; 8:e67824.
- Effect of a Multispecies Probiotic Mixture on the Growth and Incidence of Diarrhea, Im-mune Function, and Fecal Microbiota of Pre-Weaning Dairy Calves / Y. Wu [et al.] // Front. Microbiol. 2021; 12:681014.


