Видовой состав и антибиотикорезистентность бактериальных изолятов, выделенных от животных Омской области
Автор: Антоневский И.В., Плешакова В.И., Локтева А.С., Лещева Н.А.
Журнал: Вестник Красноярского государственного аграрного университета @vestnik-kgau
Рубрика: Зоотехния и ветеринария
Статья в выпуске: 6, 2023 года.
Бесплатный доступ
Цель исследования - изучить видовой состав микроорганизмов, выделенных от сельскохозяйственных и мелких домашних животных Омской области в 2022 г., определить чувствительность к антибактериальным средствам превалирующих культур в общей структуре изолятов. Бактериологическое исследование проводилось на базе БУ «Омская областная ветеринарная лаборатория» согласно действующим методикам по выделению, идентификации и определению чувствительности микроорганизмов к антибактериальным препаратам. Материалом исследования являлись пробы клинического материала (106), отобранные от сельскохозяйственных и мелких домашних животных в различных географических районах Омской области. Были выделены культуры этиологически значимых микроорганизмов, относящихся к родам: Staphylococcus, Escherichia, Proteus, Enterococcus, Streptococcus, Enterobacter, Bordetella, Citrobacter, Salmonella, Morganella, Neisseria, Malassezia, Listeria, Bacillus, Klebsiella. В общей структуре полученных культур доминировали микроорганизмы рода Staphylococcus (36 %) и Escherichia (22 %). На основании полученных данных было проведено исследование антибиотикорезистентности выделенных микроорганизмов рода Staphylococcus и вида Escherichia coli к антибактериальным средствам диско-диффузионым методом на среде Гивенталя-Ведьминой. В результате исследования установлено, что наибольшая резистентность этих микроорганизмов регистрировалась к цефалексину, стрептомицину, пефлоксацину, канамицину. Дальнейшее исследование феномена антибиотикорезистентности микроорганизмов открывает большие возможности в повышении эффективности терапии бактериальных болезней животных. Специалистам в клинической ветеринарной практике необходимо учитывать чувствительность к антибиотикам в каждом конкретном случае заболевания животных бактериальными болезнями. Такой осознанный подход к назначению терапии антибактериальными препаратами в перспективе приведет к снижению накопления генов антибиотикорезистентности среди циркулирующих штаммов микроорганизмов.
Животные, бактерии, антибиотики, антибиотикорезистентность
Короткий адрес: https://sciup.org/140300827
IDR: 140300827 | УДК: 579.22 | DOI: 10.36718/1819-4036-2023-6-97-103
Текст научной статьи Видовой состав и антибиотикорезистентность бактериальных изолятов, выделенных от животных Омской области
Введение. Развитие инфекционных процессов в организме животных зачастую происходит в результате действия ассоциаций различных микроорганизмов. При этом большая роль в этиологии таких инфекций отводится условнопатогенным микроорганизмам, которые способны вызывать гнойно-воспалительные, раневые инфекции, пищевые отравления, маститы и другие патологии. Особое значение таких инфекционных болезней в ветеринарной практике обусловливает повышенную устойчивость циркулирующих в окружающей среде возбудителей к антимикробным средствам [1, 2].
В современной клинической ветеринарной практике большое значение имеет рациональное использование антибактериальных препа- ратов. Известно, что необоснованное назначение антибиотиков при терапии животных неизбежно приводит к повышению уровня толерантности к этим средствам у циркулирующих штаммов патогенных и условно-патогенных микроорганизмов.
Такая стратегия лечения бактериальных болезней животных обуславливает сложившуюся в настоящее время тенденцию к повсеместному снижению эффективности антибактериальных препаратов [3, 4].
Для противодействия дальнейшему накоплению антибиотикотолерантности в геноме этиологически значимых микроорганизмов необходимо осуществлять рациональный подход к назначению антибактериального лечения животным [5].
Важно учитывать чувствительность к антибиотикам у выделенных микроорганизмов в каждом конкретном случае [6, 7].
Цель исследования – определить видовой состав микроорганизмов, выделенных от продуктивных и мелких домашних животных, изучить антибиотикорезистентность выделенных культур.
Материалы и методы. Материалом для исследования служили пробы клинического материала (соскобы, смывы, моча, фекалии), полученные от сельскохозяйственных (n = 67) и мелких домашних животных (n = 39) различных географических районов Омской области в 2022– 2023 гг. Исследование проводили на базе БУ «Омская областная ветеринарная лаборатория».
Идентификацию и определение биохимических свойств выделенных микроорганизмов осуществляли общепринятыми в ветеринарной лабораторной практике методами. Чувствительность выделенных культур микроорганизмов к антибактериальным препаратам определяли диско-диффузионным методом на среде Гивен-таля-Ведьминой (АГВ). Для определения чувствительности использовали коммерческие диски, характеризующие действие следующих антибактериальных препаратов (АБП): азитромицин, гентамицин, доксициклин, канамицин, левомицетин, пефлоксацин, тетрациклин, ципрофлоксацин, стрептомицин, цефалексин, фосфоми-цин, энрофлоксацин, амоксиклав, левофлоксацин. Интерпретацию значений диаметров зон задержки роста при определении чувствитель- ности микроорганизмов к антимикробным препаратам на среде АГВ проводили в соответствии с МУК 4.2.1890-04 «Определение чувствительности микроорганизмов к антибактериальным препаратам».
Результаты и их обсуждение. При бактериологическом исследовании клинического материала от животных Омской области было получено большое разнообразие микроорганизмов. Наиболее часто при этом выделяли культуры Staphylococcus aureus (n = 23) и Escherihia coli (n = 22). Помимо этого, выделены культуры S. epidermidis (n = 15), микроорганизмы рода Proteus , в том числе P. mirabilis (n = 8) и P. vulgaris (n = 4), Streptococcus pneumoniae (n = 5). Незначительное количество выделенных культур представляли микроорганизмы рода Enterococcus – E. faecalis (n = 3) и E. faecium (n = 2), а также энтеробактерии Klebsiella aerogenes (n = 4) и Citrobacter diversus (n = 3). В единичных случаях выделенные изоляты представлены такими микроорганизмами, как Salmonella, Neisseria, Bordetella, Bacillus, Malassezia, Listeria monocytogenes, Klebsiella pneumoniae, Pseudomonas apisepticus, Ps. aeruginosa, Morganella morganii (рис. 1).
В общей структуре выделенных микроорганизмов наиболее часто выделяли стафилококки, а именно S. aureus и S. epidermidis , а также E.coli . На основании полученных данных у этих микроорганизмов изучали чувствительность к антибактериальным средствам.
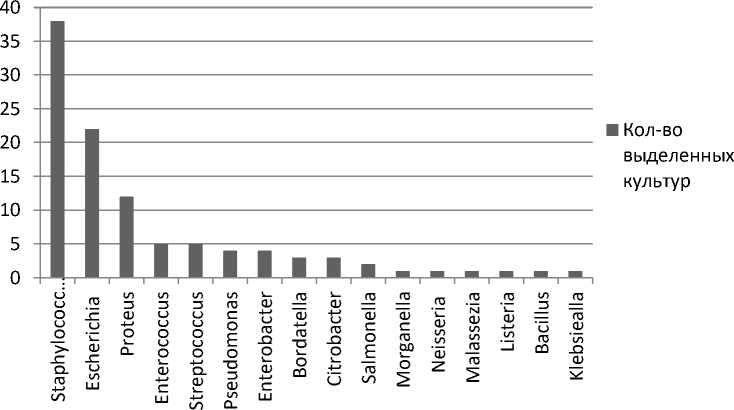
Рис. 1. Родовой состав культур микроорганизмов, выделенных от продуктивных и мелких домашних животных в 2022–2023 гг.
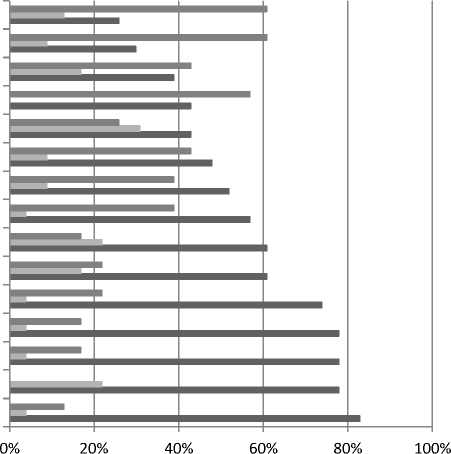
Анализ чувствительности к антибиотикам выделенных культур S. aureus показал, что данный вид микроорганизмов (более 75 % культур) был наиболее чувствителен к левофлоксацину, азитромицину, амоксиклаву, энрофлоксацину, фос-фомицину. Средний результат регистрировали при использовании таких препаратов, как гентамицин, ципрофлоксацин, левомицетин, амикацин. Чувствительность к этим антибиотикам по- казали 50–60 % выделенных микроорганизмов. Самая низкая чувствительность исследуемых культур S. aureus зафиксирована к таким препаратам, как стрептомицин, цефалексин, канами-цин, тетрациклин, пефлоксацин, доксициклин. Устойчивость микроорганизмов к этим препаратам зафиксирована у 40–60 % от общего числа выделенных культур (рис. 2).
Стрептомицин Цефалексин Канамицин Тетрациклин Пефлоксацин Доксициклин Амикацин Левомицетин Ципрофлоксацин Гентамицин Фосфомицин Энрофлоксацин Амоксиклав Азитромицин Левофлоксацин
■ Устойчиво
■ Умеренно чувствительно
■ Чувствительно
Рис. 2. Чувствительность выделенных микроорганизмов вида Staphylococcus aureus к антибактериальным средствам, %
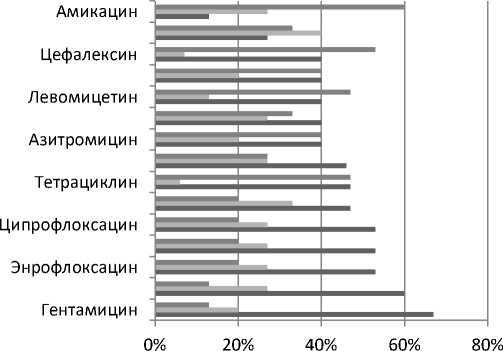
Среди выделенных культур вида Staphylococcus epidermidis более чем у 60 % микроорганизмов регистрировали чувствительность к действию гентамицина и амоксиклава. Выделенные культуры оказались наиболее устойчивы к дей ствию амикацина, цефалексина, левомицетина пефлоксацина (рис. 3).
■ Устойчиво
■ Умеренно чувствительно
■ Чувствительно
Рис. 3. Чувствительность выделенных микроорганизмов вида Staphylococcus epidermidis к антибактериальным средствам, %
Проведенный анализ резистентности микроорганизмов к антибактериальным средствам показал, что более 40 % изолированных культур S. epidermidis проявили резистентность к препаратам амикацина, цефалексина, левомицетина.
При этом более 50 % от числа исследуемых культур проявили чувствительность к гентамицину, амоксиклаву, энрофлоксацину, левофлоксацину, ципрофлоксацину.
■ Устойчиво
■ Умеренно чувствительно
■ Чувствительно
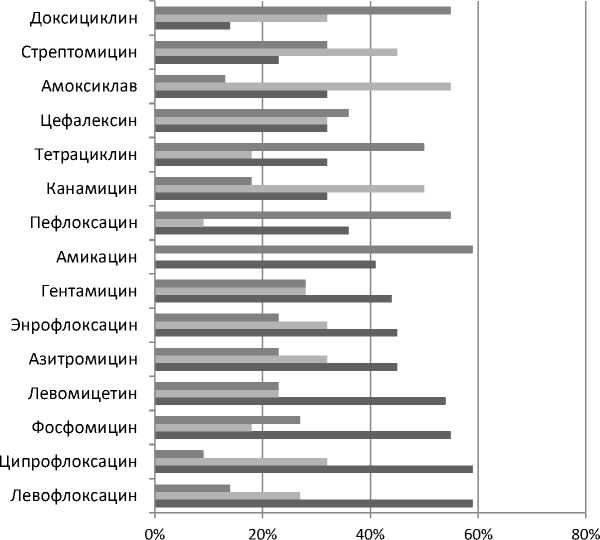
Рис. 4. Чувствительность выделенных микроорганизмов вида Escherihia coli к антибактериальным средствам, %
Среди выделенных культур E.coli определены патогенные штаммы: О117, О126, О115, О137, О86. При изучении антибиотикорези-стентности изолированных культур E.coli отмечалась повышенная устойчивость этих микроорганизмов к тетрациклину и доксициклину, пеф-локсацину, амикацину. К этим антибиотикам оказались устойчивы более 50 % от числа всех выделенных культур E. coli. При этом больше половины изученных культур E. coli оказались чувствительны к действию препаратов группы фторхинолонов: левофлоксацину и ципрофлоксацину, а также препаратам фосфомицин и левомицетин.
Заключение. По данным проведенного исследования можно сделать вывод, что в родовой структуре микроорганизмов, выделенных от сельскохозяйственных и мелких домашних жи- вотных Омской области, превалируют культуры рода Staphylococcus (36 %), из них S. aureus – 22 %, S. epidermidis – 14 % от общего количества, а также культуры E.coli – 22 %.
На основании этих данных провели исследование чувствительности к антибиотикам у представителей родов Staphylococcus и Escherichia . Выделенные культуры проявили резистентность к части используемых антимикробных средств. Так, малую эффективность в отношении этих микроорганизмов показало использование цефалексина, стрептомицина, пефлоксацина, ка-намицина. Наибольшее антибактериальное действие в отношении исследуемых культур показали препараты ципрофлоксацин, левофлоксацин, гентамицин, энрофлоксацин.
Список литературы Видовой состав и антибиотикорезистентность бактериальных изолятов, выделенных от животных Омской области
- Антибиотикочувствительность патогенных культур кишечной палочки, циркулирующих на промышленной птицефабрике Омской области / Т.И. Лоренгель [и др.] // Вестник Алтайского государственного аграрного университета. 2019. № 4 (174). С. 122-127.
- Верба Е.А., Кошкин И.Н., Конев А.В. Роль стафилококков в развитии гнойных патологий и их коррекция биопрепаратами // Актуальные проблемы ветеринарной науки и практики. Омск, 2021. С. 299-302.
- Лещева Н.А., Конищева А.С. Резистентность патогенных микроорганизмов, выделенных от птицы при промышленном содержании // Актуальные проблемы ветеринарной науки и практики. Омск, 2020. С. 201-204.
- Ручко Е.Н., Лещева Н.А., Плешакова В.И. Антибиотикорезистентность микроорганизмов рода Staphylococcus, выделенных от животных Омской области // Вестник КрасГАУ. 2022. № 8 (185). С. 116-121.
- Хрянин А.А. Бактериальные биопленки как факторы антибиотикорезистентности // Актуальные вопросы дерматовенерологии, косметологии и репродуктивного здоровья: сб. ст. междисциплинарной регион. науч.-практ. дерматологов и косметологов. Красноярск, 2020. С. 257-268.
- Understanding antimicrobial resistance (AMR) profiles of Salmonella biofilm and planktonic bacteria challenged with disinfectants commonly used during poultry processing / M. Cadena [et al.] // Foods, 2019;8(7):275.
- Understanding the mechanism of bacterial biofilms resistance to antimicrobial agents / S. Singh [et al.] The open microbiology journal. 2017; 11:53-57.


