Разработка алгоритма создания информационных ресурсов для прогнозирования гидролиза молочных белков
Автор: Чанов И.М., Зинина О.В.
Рубрика: Биохимический и пищевой инжиниринг
Статья в выпуске: 2 т.13, 2025 года.
Бесплатный доступ
В молочной промышленности, как и в других отраслях, образуется большое количество вторичных сырьевых ресурсов, одним из наиболее весомых является молочная сыворотка. Данное сырье богато белками с высоким биологическим потенциалом, реализуемым при гидролизе белка и получении биоактивных пептидов. Выбор ферментов для проведения гидролиза с целью получения тех или иных биоактивных пептидов сопряжен с длительностью и затратностью, что можно оптимизировать применением методов in silico. Целью работы является разработка алгоритма создания базы данных для моделирования влияния ферментов на сывороточные белки, а также программного обеспечения для оптимизации целенаправленного гидролиза белков молочной сыворотки ферментами, воздействующими на β-лактоглобулин и α-лактальбумин, для получения специфичных биологически активных пептидов. Молочная сыворотка является источником белков β-лактоглобулина и α-лактальбумина. Информация для формирования базы данных была получена из известных источников (BIOPEP-UWM, PeptideCutter, UniProtKB и InterPro). База данных создавалась с использованием системы управления базами данных SQLite. Программа разрабатывалась на основе созданной базы данных и с применением графической библиотеки виджетов Tkinter с использованием языка программирования Python. Проверена работоспособность программы, она выдаёт точный набор ферментов для получения пептида или цепочки пептидов в зависимости от предварительно выбранного для гидролиза молочного белка.
Молочная сыворотка, сывороточные белки, база данных, гидролиз белка
Короткий адрес: https://sciup.org/147250714
IDR: 147250714 | УДК: 637.045 | DOI: 10.14529/food250212
Текст научной статьи Разработка алгоритма создания информационных ресурсов для прогнозирования гидролиза молочных белков
Пищевые отходы благодаря своему органическому составу и масштабному производству представляют собой значительный ресурс для вторичной переработки [1]. Эти отходы образуются на различных стадиях производственного цикла – начиная от производства, последующей обработки и заканчивая переработкой пищевых продуктов [2]. ООН ставит цель: уменьшить количество пищевых отходов вдвое к 2030 году. Решение этой проблемы также имеет важное экономическое значение [3]. Ежегодно в пищевой отрасли образуется несколько миллионов тонн вторичных ресурсов и отходов [4]. Согласно данным Федерального классификационного каталога отходов (ФККО), утверждённого приказом Росприроднадзора от 22.05.2017 № 242 (ред. от 20.12.2024), основными отходами молочной промышленности являются пахта, обезжиренное молоко и молочная сыворотка [5].
Молочная сыворотка представляет собой ценный источник сывороточных белков, обогащённых минералами, витаминами и другими полезными веществами [6]. Под действием ферментативного катализа она подвергается гидролизу, который обеспечивает получение белков с уникальными функциями благодаря наличию биоактивных пептидов [7]. Биоактивные пептиды – это белковые фрагменты, полезные для функционирования различных систем организма и общего состояния здоровья человека [8].
Ферментативный гидролиз белков молочной сыворотки представляет собой перспек-
тивный метод модификации функциональных свойств белкового сырья, направленный на получение ценных пептидных фракций, обладающих широким спектром биологической активности. Данный процесс позволяет целенаправленно расщеплять полипептидные цепи белков молочной сыворотки под действием протеолитических ферментов, высвобождая пептиды с определенными аминокислотными последовательностями, которые могут проявлять антиоксидантные, антигипертензивные, иммуномодулирующие и другие полезные свойства [9–11].
Сегодня одним из эффективных методов прогнозирования активности ферментов является применение молекулярного моделирования [12], которое помогает существенно сократить время на выявление возможных биологических эффектов [13].
Метод in silico представляет собой мощный инструмент в исследовании ферментативного гидролиза белков молочной сыворотки, позволяющий оптимизировать протеолиз, прогнозировать образование биоактивных пептидов и сократить время и затраты на экспериментальные исследования. Применение вычислительных методов позволяет детально изучить взаимодействие протеаз с белками на молекулярном уровне, предсказывать места расщепления полипептидных цепей и идентифицировать пептиды с потенциальной биологической активностью.
Актуальным направлением является разработка информационных ресурсов, используемых для прогнозирования процесса гидролиза молочных белков с установлением выхода
необходимых биоактивных пептидов. Таким образом, целью работы является разработка алгоритма создания базы данных для моделирования влияния ферментов на сывороточные белки, а также программного обеспечения для оптимизации целенаправленного гидролиза белков молочной сыворотки ферментами, воздействующими на β-лактоглобулин и α-лактальбумин, для получения специфичных биологически активных пептидов.
Материалы и методы
Проведён поиск информации о сывороточных белках β-лактоглобулине и α-лакталь-бумине с использованием баз данных UniProtKB и InterPro.
UniProt – это база данных, включающая данные о последовательностях белков и их биологической роли, полученные из научных публикаций.
InterPro – инструмент, предоставляющий функциональный анализ белков путём группировки их по категориям и предсказания доменов и важных участков [14].
Для изучения влияния ферментов на биотехнологические характеристики белков методом in silico применялась база данных BIOPEP-UWM.
BIOPEP-UWM используется в исследованиях биологически активных пептидов, особенно полученных из пищевых субстратов [15].
Выбор ферментов производился с помощью базы данных PeptideCutter.
PeptideCutter предсказывает возможные точки расщепления, осуществляемые протеазами или химическими агентами в заданной белковой последовательности.
Последовательности белков β-лактогло-
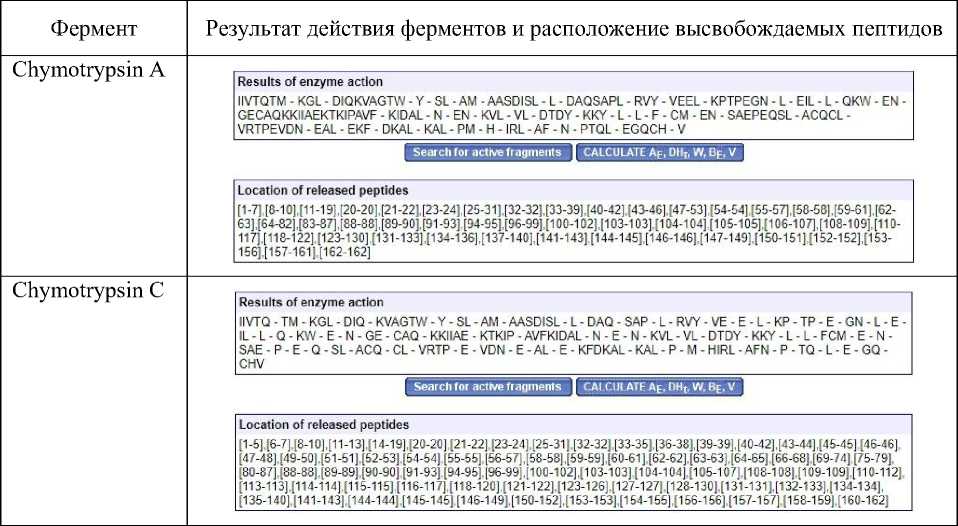
булина и α-лактальбумина были получены из базы данных белков UniProtKB или InterPro. PeptideCutter генерирует перечень ферментов, способных расщеплять данную аминокислотную последовательность, указывает число точек расщепления и их местоположение, а также выделяет ферменты, неспособные осуществить расщепление. Среди расщепляющих ферментов предпочтение было отдано тем, которые обеспечивают наибольшее количество расщеплений: Proteinase K; Thermolysin; Pepsin (pH 1.3); Pepsin (pH > 2); Trypsin; Chymotrypsin A; Chymotrypsin C.
Все данные, собранные из базы данных BIOPEP-UWM, требуются для формирования новой базы данных. Для её разработки был использован язык программирования Python.
Python – это интерпретируемый высокоуровневый универсальный язык программирования широкого профиля.
Для описания структуры баз данных, управления данными, организации доступа к ним и объектов базы данных, а также управления транзакциями применяется язык SQL (Structured Query Language). Для работы с данными выбрана система управления базами данных SQLite.
SQLite – это реляционная СУБД, где вся база данных хранится в одном файле [16]. Для подключения программы на языке Python к системе управления базами данных SQLite, используется модуль “sqlite3”, который именуется синонимом “sq”.
Результаты исследований и обсуждение
Результаты действия ферментов на β-лактоглобулин и α-лактальбумин представлены в табл. 1, 2.
Результаты действия ферментов на α-лактальбумин
Таблица 1
Фермент
Результат действия ферментов и расположение высвобождаемых пептидов
Proteinase K
Results of enzyme action
EQL - TKCEAF - QKL- KDL - KDY - GGV - SL - P - EW - V - CTAF - HTSGY - DTQAI - V - QNNDSTEY - GL - F - QI - NNKI -W - CKDDQNP - HSRNI ■ CNI - SCDKF - L - DDDL - TDDI - V - CAKKI ■ L - DKV - 61 - NY - W - L - AHKAL - CSEKL - DQW -L-CEKL
Search for active fragments

Location of released peptides
[1-3].[4-9].[W-12].[13-15].[16-18].[19-21].[22-23].[24-24].[2&-26].[27-27]T[28-31].p2-36].p7-41].[42-42].[43-50];51-S2].[53-53]. [54-55],[56-59],[60-60],[61-67],[68-7^,|73-75|, [76-80] .[81-81| ,[82-85],[86-89],[90-9D|.[S 1 ■9S]1[36-«|.[97S9|.[1 Ш 01].[l 02-103],|104-1М|.|1К-1й]1[106-110],|111-115].1116-118],|119-119]|121-123]
Окончание табл. 1
|
Фермент |
Результат действия ферментов и расположение высвобождаемых пептидов |
||
|
Thermolysin |
Results of enzyme action EQ - ИКСЕ - A - FQK - LKD - LKD - YGG - VS - LPEW - VCT - A - FHTSG - YDTQ - A-1 - VQNNDSTE - YG - L - FQ - INNK - IWCKDDQNPHSRN - ICN - ISCDK - F - LDDD - LTDD -1 - VC - АКК -1 - LDK ■ VG ■ IN - YW ■ L ■ AHK - A - LCSEK -LDQW-LCEK-L |
||
|
So ch for active fragments |
1 ije <«!,-, (й- л 3>. v ■ . |
||
|
Location of released peptides [1-2].[3-7].[8-S].[9-11].[12-14].|15-in.[18-20].pi-22].[23-26].[27-29].[3M0]^^^^ 52П53<54П55-58Н59-71П72П4].[75-79].[&0-80].[81-84].[85-88]з89-89].[90-91].[92-94].[95-95].[96-98],|99-100].Г101-102] [1O34M],[1O5-1O5]J1M-iO0lflO9^^^^ [119-122],[123-1231 |
|||
|
Pepsin pH >2 |
Results of enzyme action E-Q-L-T-K-CE-A-F-Q -VQ-N-N-D-ST-E-Y-G T-D-D-I-VCA-K-K-IL- Location of released peptide [1-1].[2-2]:[3-3].[4-4].[5-5]:[8-7] [21-23].[24-25][26-26].[27-29], [4M6].[«-4t][tM8]j4W9 [56-66][6?-71][72-74].i75-78], [S3-93].[S4-34].p5-S6].[97-17|. 1U].|114-114['|115-118];[11t-1 |
-K-L-K-D-L-K-D-Y-G -L-F-Q-IN-N-K-IWCK-D-K-VG-IN-Y-WL-A-Hi s H[9-S],[10-10].[11-11].[12-11 30-30|[31-31].|32-33],[34-35] [ 50-50|.[51-61].[52-62].[53-53] 75-79|[8Ч.|81-81][82-82].| 98-98|199-1M].[1SMl2].[1!3 6],[117-117Й118-119],[120-12 |
-G-VSL-PE-W-VCT-A-F-HT-SG-Y-D-T-Q-A-I D - D - G-N -PHSRN -iCN - iSCD - K-F - L- C -D - D - L- -A-L-CSE-K-L-D-Q-WL-CE-K-L ___________ ].:'13-13].F14-14i [15-15] [16-16i.[17-17].|18-18].[19-19:.[20-20]. 36-36].|37-37].[38-38].[39-39].[40-40].[41-41|.[42-43]J44-44]. 54-54]1|5^56]1[57-57].[58-58],[59-62].[63-63|.[64-64].F65-65]. 83-83]J84-84].[85-B5].|86-85].[87-S7][88-88|.[8MS]fJH2] 1C3].|lO4-1O5|.[1O6-1O6],[1O7-1O8][iO9-109].[11O-1^^ l|,[122-122],[123-123[..... .......................... |
|
Trypsin |
Results of enzyme action EQLTK-CEAFQK-LK-DLK- NICNISCDK - FLDDDLTDDIVC Location of released peptides [1-5Ц6-11]. [12-13] .[14-16],[17-£ |
DYGGVSLPEWVCTAFHTSG’ AK - К - ILDK - VGINYWLAHK 8], [59-62], [63-70], [71-79], [80-9 |
rDTQAIVQNNDSTEYGLFQINNK - IWCK - DDQNPHSR - -ALCSEK-LDQWLCEK-L ______________________ (....__.._:...:..,...._..._......_! 3].[94-94].[95-98].[99-108].[109-114],[115-122],[123-123] |
|
Chymotrypsin A |
Results of enzyme action EQL - TKCEAF - QKL - KDL - К CKDDQN - PH - SRN - ICN - IS |CEKL __________________ Location of released peptides [1-ЗМ4-9]. [10-12].[ 13-15L[16-1 a [58-6 0]. [61 -66]. [67-68]. [69-71 ]. [ 110], [111-115],[116-118], [119-11 |
DY - GGVSL - PEW - VCTAF -CDKF - L - DDDL - TDDIVCAK ]. [ 19-23], [24-26], [27-31 ], [32-32 ’2-74] 75-80] [81 -81 ], [82-85],[ 9] ,[120-123] |
H - TSGY - DTQAIVQN - N - DSTEY - GL - F - QIN - N - KIW -KIL - DKVGIN - Y - W - L - AH - KAL - CSEKL - DQW - L - L1„:..-_J ],[33-36],[37-44],[45-45].[46-50].[51-52].’53-53].i'54-56].[57-57] 36-96],[97-102],[103-103] [104-104] [105-105],[106-107].[108- |
|
Chymotrypsin C |
Results of enzyme action E - Q-L-TKCE-ArQ-KL- К IN-N-KIW-CKDDQ-N-P- OQ-W-L-CE-KL ______ Location of released peptide: [1-1][2-2].'3-3].[4-7|.[8-10].>11-49],[50-50].[51-52],|53-54].[55-' 1C3].[104-104],[W5-105]1[106-1 |
DL-KDY-GGVSL-P-E-W HSRN-ICN-ISCDKFL-DDD 2] [13-15].[16-18]. [19-23],[24-2 8] [57-57].[58-60].[61-65].F6G-6 10],[111-113},[114-116],[116-11 |
VCTAFHTSGY- DTQ - AIVQ - N - N ■ DSTE - Y-SL-FO- L - TDDIVCAKKIL - DKVGIN - Y - W - L - AHKAL - CSE - KL - [Ж^^^ 4],|25-28].|26-26]1[27-35].[37-39][4»-43].[44-44|[45-46].[46- 6][67-67].[68-71].[72-74],[75-81].[82-85].[86-96].[97-102j.f103- 7i [118-11 S].!119-119],[120-121]. 122-123] |
Таблица 2
|
Фермент |
Результат действия ферментов и расположение высвобождаемых пептидов |
||
|
Proteinase K |
Results of enzyme action l-i-V-TQTM-KGL-DS-QKV-AGTW-Y-SL-AM-AASDi-SL-L-DAQSAP-L-RV-Y-V-EEL-KP-TP-EGNL-EI-L-L-QKW-ENGECAQKKI-i-AEKTKI-P-AV-F-KI-OAL-NENKV-L-V- L-DTDY-№ - L-L-F - CM-ENSAEP-EQSL-ACQCL- V - RTF - EV - DNEAL- EKF - DKAL - KAL - P - M - Hi - RL - AF - NP - TQL -EGQCHV |
||
|
M ■ MMHlihU a |
1 CALCULATE AE.DHt.W.BE.V |
||
|
Location of released peptides [1-1],[2-2},[3-3].[4-7].[8-10] [11-12Ц13-15] [15-19].[20-20].[21-22].[23-24],[25-29].[3D-31],p2-32].[33-38].[39-39].[40-41].[42-42]l[43-43]l|44-4S]l[47-48].[«-50].p1-54][55-56].[57-57]l[5S-58|.i59-G1]{S2-71].[72-72]l[73-78]l[79-79]l[B0-31]l[82-82]l[83-84].[35-87].|S8-92].[93-93].[94-94].[95-95].[96-99].[WO-102].[103-103].[104-104].[1D5-1D5].[106-f07],[108-113].[114-117J. [113-122J.[123-123]l[124-12S].[127-128].[129-133]l[134-13&].[137-140].|141-143].[144-144].[145-145] [14G-147].[148-149]. дамэд^^ ________________________ |
|||
|
Thermolysin |
Results of enzyme action l-l-VTQTMKG-LD-IQK-V-AGfW-YS-L-AM-A-ASD-IS-L-LD-AQS-AP-LR-V-Y-VEE-LKPTPEGN-LE-I - L - LQKWENGEC - AQKK -1 -1 - AEKTK - IP - A - V - FK - ID-A - LNENK - V- L- V- LDTD- YKK-Y - L- L-FCMENS -AEPEQS - L - ACQC - L - VRTPE - VDNE - A - LEK - FDK - A- LK - A - LPMH - IR - L - A - FNPTQ - LEGQCH - V |
||
|
Location of released peptides (1-1И2-2|.'3-Э].[10-4] ;'12-14].ПЕ-1=]416-18] :'2С-21] 22-22|.'23-24i.[25-25’[28-28] [29-30l.[31-3i].[32-33l.[34-36];37-38]. [ЗЭ-41].[4141].[42-42].!4345].146-53].!54-55].[56-56].|57-57Ь|58«],[б™],[71-71]1[72-74[/ЗТ [82-83].[84-85].[86-86]|87-91].[92-92].-93-93].[94-94].|95-98|,[99-1C1]I[102-102].|1C3-103].[104-104],[105-110].[111-116], [117-117].[118-121]1[122-122].[123-127].[128-131],[132-132] [133-135].[136-138j.[139-139]l[140-141].[142-142] [143-146], ][Ш44ад49449Ц150^^______________________________ |
|||
|
Pepsin pH 1.3 |
Results of enzyme action IIVTQTMKGL - DIQKVAGTWYSL - AMAASDISL - L - DAQSAPL - RVYVEEL - KPT PEGNL - EL - L -QKWENGECAQKKIIAEKTKIPAVF - KIDAL - NENKVL - VL - DTDYKKYL - L - F - GMENSAEPEQSL - ACQCL -VRTPEVDNEAL - EKF - DKAL - KAL - PMHIRL - AF - NPTQL- EGQCHV |
||
|
Location of released peptides [1-10],[11-22],[23-31],[32-32].[33-39].[40-46].[47-54].[55-57].[58-58],[59-82],[83-87],[8S-93],[94-95],[96-103].[104-104],[105-105], [106-117], [118-122].[123-133],[134-136], [137-140], [141-143], [144-149], [150-151],[152-156], [157-162] |
|||
|
Pepsin pH >2 |
Results of enzyme action l-l-VT-Q-T-M-K-G-L-D-IQ-K-VA-G-T-WY-SL-A-M-A-A-SD-ISL-L-D - E - L - К - PT - PE - G - N - L - E - IL - L - Q - К - WE - M - G - E - CA - Q - К - К -1 - IA - E - К - N - E - N - К - VL - VL - D - T - D - Y - К - К - Y - L - L - F - CM - E - N - SA - E - PE - Q - SL - / N-E-A-L- E-K-F-D-K-A-L-K-A-L-PM-H - IRL-A-F-N-PT-Q-L-E-G-Q |
-A-Q-SA-PL-R-VY-VE -T-К - IPA-VF - K-ID-А-L A CQ - CL - VRT - PE - VD - CH-V |
|
|
I CALCULATE AE, DHt W. BE. V |
|||
|
Location of released peptides [1-1],[2-2],[3-4],[5-5].[6-6].[7-7],[8-8].[9-9],[10-10].[11-11].[12-13].[14-14].[15-16].[17-17],[18-18],[19-20],[21-22],[23-23],[24-24], [25-25].[26-26].[27-28].[29-31].[32-32],[33-33],[34-34],[35-35],[36-37],[3&-39],[40-40],[41-42],[43-44].[45-45].[46-46].[47-47]. [48-49].[50-51].[52-52].[53-53],[54-54].[55-55].[56-57],[58-58],[5 9-59],[6 0-60],[61-62],[63-6 3],[64-64].[65-65].[66-67].[68-68]. [69-69],[70-70],[71-71],[72-73].[74-74].[75-75]. [76-76],[77-77],[78-80],[81-82],[83-83],[84-85],[86-86],[87-87],[88-88].[89-89] [90-90].[91-91].[92-93].[94-95].[96-96].[97-97].[98-98],[99-99].[100-100].[101-101],[102-102].[103-103],[104-104].[105-105], [1О6-1О7Ц108-108],[109-109],[110-111] [112-112],[113-114],[115-115],[116-117],[118-118],[119-120],[121-122Ц123-125],[126-127],[128-129] [130-130].[131-131 j.[132-132].[133-133],[134-134].[135-135],[136-136].И37-137],[138-138].’139-139],[140-140],[141-141].[142-142],[143-143].[144-145],[146-146],[147-149].[150-150],[151-151].[152-152],[153-154].[155-155],[156-[l56yi57-157]jl58-15i^ ____________________________________________ |
|||
|
Trypsin |
Results of enzyme action IIVTQTMK - GLDIQK - VAGTWYSLAMAASDISLLDAQSAPLR - VYVEELK - PTPEGNLEILLQK - WENGECAQK - К - ПАЕК -TK - 1PAVFK - IDALNENK - VLVLDTDYK - К - YLLFCMENSAEPEQSLACQCLVR - TPEVDNEALEK - FDK - ALK - ALPMHIR - LAFNPTQLEGQCHV |
||
|
1 CALCULATE AE1 DHT, W, BE, V |
|||
|
Location of released peptides [1-8],[9-14J.[15-40],[41-47],[48-60],[61-69],[7 0-70],[71-75],[76-77].[78-83].[84-91],[92-10 0].[101-101].[10 2-124],[12 5-135].[13613 8], [ 139-141 ], [142-148], [149-162] |
|||
Окончание табл. 2
Начальная часть кода для работы с базой данных показана на рис. 1.
Рис. 1. Код привязки языка Python с системой баз данных SQLite
В методе “execute” выполняется запрос “CREATE TABLE alphalactalbumin”, что означает создание таблицы для необходимого белка α-лактальбумина, аналогичные действия выполняются при заполнении таблицы данных для β-лактоглобулина. Получившийся код создания таблиц в базе данных представлен на рис. 2.
cur.execute("""CREATE TABLE IF NOT EXISTS alphalactalbumin ( Фермент TEXT, Высвобожденные пептиды TEXT
) n n n )
cur.execute("""CREATE TABLE IF NOT EXISTS betalactoglobulin ( Фермент TEXT, Высвобожденные пептиды TEXT
I nnnt
Рис. 2. Код создания таблиц в базе данных
Для подтверждения создания запрашиваемой таблицы используется метод “commit”.
Код получившейся базы данных представлен на рис. 3.
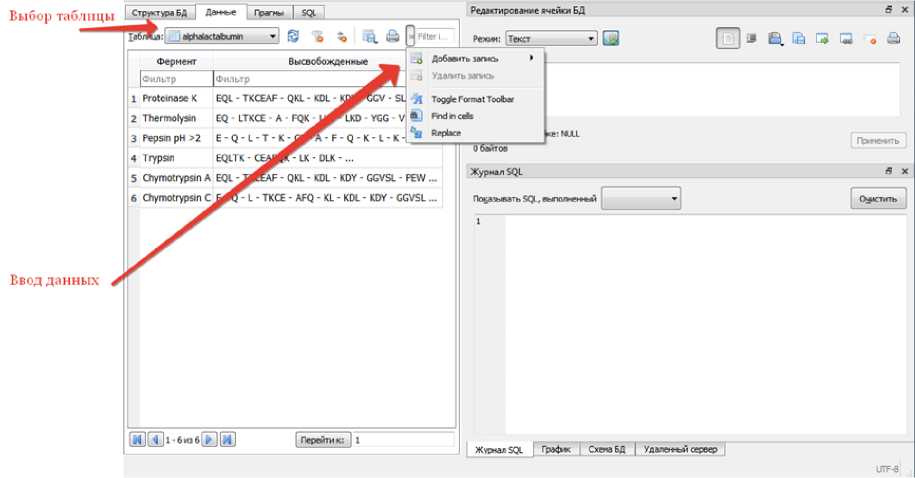
Для внесения новых данных в полученную базу данных используется система управления баз данных SQLite. Для внесения данных о высвобождаемых пептидах необхо-
димо нажать на вкладку «Данные», в списке таблиц выбирается “alphalactalbumin”, далее – «Добавить запись», как показано на рис. 4, и вводятся все необходимые данные для данной таблицы. Аналогичные действия производятся с таблицей “betalactoglobulin”.
Итоговые таблицы с данными о пептидах представлены на рис. 5.
Рис. 3. Код создания базы данных
Результаты действия ферментов на β-лактоглобулин
Рис. 4. Ввод данных в базу данных
Таблица: , L alphalactalbumm » § о 49 ей, Q ” Fllt^ 1
Фермент Высвобожденные
|
Фильтр |
Фильтр |
|
1 Proteinase К |
EQL - TKCEAF - QKL - KDL - KDY - GGV - SL - P -... |
|
2 Thermolysin |
EQ - LTKCE - A - FQK - LKD - LKD - YGG - VS - ... |
|
3 Pepsin pH >2 |
E-Q-L-T-K-CE-A-F-Q-K-L-K-D-L.. |
|
4 Trypsin |
EQLTK - CEAFQK - LK - DLK -... |
|
5 Chymotrypsin A |
EQL - TKCEAF - QKL - KDL - KDY - GGVSL - PEW ... |
|
6 Chymotrypsin C |
E - Q - L - TKCE - AFQ - KL - KDL - KDY - GGVSL ... |

|
Фермент |
Высвобожденные |
|
|фильтр |
Фильтр |
|
1 Proteinase К |
I - I - V - TQTM - KGL - DI - QKV - AGTW - Y - S... |
|
2 Thermolysin |
I - I - VTQTMKG - LD - IQK - V - AGTW - YS - L - ... |
|
3 Pepsin pH 1.3 |
IIVTQTMKGL - DIQKVAGTWYSL - AMAASDISL - L... |
|
4 Pepsin pH >2 |
I - I - VT - Q - T - M - К - G - L - D - IQ - К - VA -... |
|
5 Trypsin |
HVTQTMK - GLDIQK - ... |
6 Chymotrypsin A IIVTQTM - KGL • DIQKVAGTW - Y - SL - AM - ...
7 Chymotrypsin C HVTQ - TM - KGL - DIQ - KVAGTW - Y - SL - AM ...
Рис. 5. Вид готовых таблиц с данными
Для создания программы для поиска ферментов по заданным пептидам или цепочке пептидов белков α-лактальбумин и β-лакто-глобулин требуется подключение всех необходимых библиотек, также необходимо разработать интерфейс для программы и саму программу.
При запуске программы появляется окно (рис. 6), в котором можно выбрать alphalactalbumin или betalactoglobulin. В поле «Введите пептид» можно ввести пептид или цепочку пептидов в зависимости от их однобуквенного кода (табл. 3). В поле «Результаты поиска» отображаются результаты поиска после введения пептида или цепочки пептида и нажатия кнопки «Найти ферменты».
На рис. 7 приведен пример подбора ферментов для гидролиза α-лактальбумина с целью получения пептида EQL и гидролиза β-лактоглобулина с целью получения свободной аминокислоты изолейцина.
Примеры работы программы показали исправность работы программы, а именно программа выдаёт точный набор ферментов для получения пептида или цепочки пептидов в зависимости от предварительно выбранного для гидролиза белка.
Выводы
Проведен анализ баз данных, выявлены ферменты, необходимые для расщепления α-лактальбумина и β-лактоглобулина, а также определены результаты их действия и локализация выделяемых пептидов. Отбор ферментов проводился с использованием баз данных UniProtKB, AlphaFold Protein Structure Database и InterPro. Результаты действия ферментов и расположение освобожденных пептидов были получены с помощью BIOPEP-UWM.
На основании собранных данных разработан алгоритм создания базы данных для оценки воздействия ферментов на α-лакталь-бумин и β-лактоглобулин с использованием реляционной системы управления базами данных SQLite.
С помощью готовой базы данных и библиотеки TKinter на языке программирования Python разработана программа для подбора ферментов по указанному пользователем пептиду.
Тестирование показало корректную работу программы по подбору ферментов согласно заданному пептиду.
Таблица 3
|
Аминокислота |
Однобуквенный код |
Аминокислота |
Однобуквенный код |
|
Глицин |
G |
Треонин |
T |
|
Аланин |
A |
Аспарагиновая кислота |
D |
|
Валин |
V |
Глутаминовая кислота |
E |
|
Лейцин |
L |
Аспарагин |
N |
|
Изолейцин |
I |
Глутамин |
Q |
|
Пролин |
P |
Цистеин |
C |
|
Фенилаланин |
F |
Метионин |
M |
|
Тирозин |
Y |
Гистидин |
H |
|
Триптофан |
W |
Лизин |
K |
|
Серин |
S |
Аргинин |
R |


